Сотовая модель - Cellular model

Создание сотовая модель была особенно сложной задачей системная биология и математическая биология. Это предполагает разработку эффективных алгоритмы, структуры данных, визуализация и средства связи для организации интеграции больших объемов биологических данных с целью компьютерное моделирование.
Он также напрямую связан с биоинформатика, вычислительная биология и Искусственная жизнь.
Это предполагает использование компьютерное моделирование из многих Сотовая связь подсистемы, такие как сети метаболитов и ферменты которые включают метаболизм, преобразование сигнала пути и сети регуляции генов как анализировать, так и визуализировать сложные связи этих клеточных процессов.
Сложная сеть процессов биохимических реакций / переноса и их пространственная организация делают разработку прогнозной модели живой клетки серьезной задачей 21 века.
Обзор
Эукариотический клеточный цикл очень сложна и является одной из наиболее изучаемых тем, поскольку ее неправильное регулирование приводит к раки Возможно, это хороший пример математической модели, поскольку она имеет дело с простыми вычислениями, но дает достоверные результаты. Две исследовательские группы[1][2] создали несколько моделей клеточного цикла, имитирующих несколько организмов. Недавно они создали общую модель эукариотического клеточного цикла, которая может представлять конкретного эукариота в зависимости от значений параметров, демонстрируя, что идиосинкразии отдельных клеточных циклов обусловлены различными концентрациями белка и сродством, в то время как лежащие в основе механизмы сохраняются (Csikasz -Nagy et al., 2006).
С помощью системы обыкновенные дифференциальные уравнения эти модели показывают изменение во времени (динамическая система ) белка внутри одной типичной клетки; этот тип модели называется детерминированный процесс (тогда как модель, описывающая статистическое распределение концентраций белка в популяции клеток, называется случайный процесс ).
Чтобы получить эти уравнения, необходимо выполнить итеративную серию шагов: сначала несколько моделей и наблюдений объединяются, чтобы сформировать консенсусную диаграмму, и соответствующие кинетические законы выбираются для написания дифференциальных уравнений, таких как кинетика скорости для стехиометрических реакций, Кинетика Михаэлиса-Ментен для ферментативных субстратных реакций и Кинетика Гольдбетера – Кошланда для сверхчувствительных факторов транскрипции после этого параметры уравнений (константы скорости, коэффициенты эффективности ферментов и константы Михаэлиса) должны быть подогнаны для соответствия наблюдениям; когда они не могут быть установлены, кинетическое уравнение пересматривается, а когда это невозможно, изменяется электрическая схема. Параметры подбираются и проверяются с использованием наблюдений как за диким типом, так и за мутантами, такими как период полужизни белка и размер клеток.
Чтобы подобрать параметры, необходимо изучить дифференциальные уравнения. Это можно сделать либо путем моделирования, либо путем анализа.
В моделировании с учетом стартового вектор (список значений переменных), прогрессия системы рассчитывается путем решения уравнений в каждом временном интервале с небольшими приращениями.
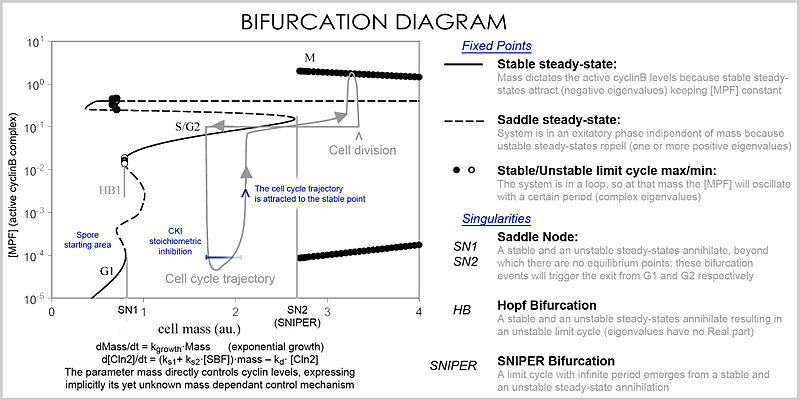
При анализе свойства уравнений используются для исследования поведения системы в зависимости от значений параметров и переменных. Систему дифференциальных уравнений можно представить в виде векторное поле, где каждый вектор описывает изменение (в концентрации двух или более белков), определяющее, куда и как быстро движется траектория (моделирование). Векторные поля могут иметь несколько особых точек: стабильная точка, называемый стоком, который притягивается во всех направлениях (заставляя концентрации быть на определенном уровне), нестабильная точка, либо источник, либо точка перевала который отталкивает (заставляя концентрации изменяться от определенного значения), и предельный цикл, замкнутая траектория, к которой несколько траекторий движутся по спирали (заставляя концентрации колебаться).
Лучшее представление, которое может обрабатывать большое количество переменных и параметров, называется бифуркационная диаграмма (теория бифуркации ): наличие этих особых установившихся точек при определенных значениях параметра (например, массы) представлено точкой, и как только параметр переходит определенное значение, происходит качественное изменение, называемое бифуркацией, в которой характер пространственные изменения с глубокими последствиями для концентраций белков: клеточный цикл имеет фазы (частично соответствующие G1 и G2), в которых масса через стабильную точку контролирует уровни циклина, и фазы (фазы S и M), в которых концентрации изменяются независимо, но как только фаза изменилась при бифуркационном событии (контрольная точка клеточного цикла ), система не может вернуться на предыдущие уровни, так как при текущей массе векторное поле сильно отличается, и масса не может быть обращена обратно через событие бифуркации, что делает контрольную точку необратимой. В частности, контрольно-пропускные пункты S и M регулируются с помощью специальных бифуркаций, называемых Бифуркация хопфа и бифуркация бесконечного периода.
Моделирование на молекулярном уровне
Клеточный коллектив[3] это программное обеспечение для моделирования, которое позволяет размещать динамические биологические данные, строить вычислительные модели, стимулировать, разбивать и воссоздавать модели. Разработкой руководит Томас Геликар,[4] исследователь в области вычислительной биологии. Он предназначен для биологов, студентов, изучающих вычислительную биологию, учителей, специализирующихся на преподавании наук о жизни, и исследователей в области наук о жизни. Сложности математики и информатики встроены в серверную часть, и можно узнать о методах, используемых для моделирования биологических видов, но сложные математические уравнения, алгоритмы, программирование не требуются и, следовательно, не будут препятствовать построению модели.
Математическая структура, лежащая в основе Cell Collective, основана на общей методике качественного (дискретного) моделирования, в которой регуляторный механизм каждого узла описывается логической функцией [для получения более полной информации о логическом моделировании см. [5][6]].
Валидация модели Модель была построена с использованием локальной (например, межбелкового взаимодействия) информации из первичной литературы. Другими словами, на этапе построения модели не было попыток определить локальные взаимодействия на основе каких-либо других более крупных фенотипов или явлений. Однако после того, как модель была завершена, проверка точности модели включала проверку ее способности воспроизводить сложные явления ввода-вывода, которые наблюдались в лаборатории. Для этого модель Т-клеток была смоделирована во множестве клеточных условий и проанализирована с точки зрения кривых «вход-выход - доза-реакция», чтобы определить, ведет ли модель себя так, как ожидалось, включая различные последующие эффекты в результате активации TCR. , Связанные с G-белком рецепторы, пути цитокинов и интегрина.[7]
Проект E-Cell[8] стремится «сделать возможным точное моделирование целых клеток на молекулярном уровне».[9]
CytoSolve - разработан В. А. Шива Айядурай и К. Форбс Дьюи младший из отдела биологической инженерии Массачусетский Институт Технологий - предоставил метод моделирования всей клетки путем динамической интеграции нескольких моделей молекулярных путей. . "[10][11]
В июльском номере журнала 2012 г. Клетка, команда во главе с Маркус Коверт в Стэнфорде опубликовали наиболее полную на сегодняшний день вычислительную модель клетки. Модель примерно 500-го гена Mycoplasma genitalium содержит 28 алгоритмически независимых компонентов, включающих работы из более чем 900 источников. Он учитывает взаимодействие всего геном, транскриптом, протеом, и метаболом организма, что является значительным достижением в этой области.[12][13]
Большинство попыток моделирования процессов клеточного цикла были сосредоточены на широких и сложных молекулярных взаимодействиях множества различных химических веществ, в том числе нескольких. циклин и циклин-зависимая киназа молекулы, поскольку они соответствуют S, M, G1 и G2 этапы клеточный цикл. В статье, опубликованной в 2014 г. в журнале «Вычислительная биология PLOS», сотрудники Оксфордский университет, Технологический институт Вирджинии и Institut de Génétique et Développement de Rennes создали упрощенную модель клеточного цикла, используя только одно взаимодействие циклин / CDK. Эта модель показала возможность управления полностью функциональным деление клеток посредством регулирования и манипулирования только одним взаимодействием, и даже позволял исследователям пропускать фазы за счет изменения концентрации CDK.[14] Эта модель может помочь понять, как относительно простые взаимодействия одного химического вещества переводятся в модель клеточного деления на уровне клетки.
Проекты
Несколько проектов в стадии реализации.[15]
- CytoSolve
- Synthecell
- Кариоте - Университет Индианы
- E-Cell проект
- Виртуальная ячейка - Центр здоровья Университета Коннектикута
- Кремниевая ячейка
- WholeCell - Стэндфордский Университет
- MCell - Национальный центр многомасштабного моделирования биологических систем (MMBioS)
Смотрите также
- Визуализация биологических данных
- Биологические приложения теории бифуркаций
- Программное обеспечение для молекулярного моделирования
- Мембранные вычисления задача моделирования именно клеточная мембрана.
- Биохимические переключатели в клеточном цикле
- Масару Томита
Рекомендации
- ^ "Лаборатория Джей Джей Тайсона". Технологический институт Вирджинии. Получено 2011-07-20.
- ^ "Группа исследования динамики молекулярных сетей". Будапештский технологический и экономический университет.
- ^ «Интерактивное моделирование биологических сетей».
- ^ «Геликар Лаб - Участники». Архивировано из оригинал на 2019-10-19. Получено 2016-02-15.
- ^ Моррис М.К., Саез-Родригес Дж, Соргер П.К., Лауффенбургер Д.А. Модели на основе логики для анализа сетей передачи сигналов в клетках. Биохимия (2010) 49 (15): 3216–24.10.1021 / bi902202q
- ^ Геликар Т., Коваль Б., Мадрахимов А., Шреста М., Педерсен Дж., Лимбу К. и др. Bio-Logic Builder: нетехнический инструмент для построения динамичных, качественных моделей. PLoS One (2012) 7 (10): e46417.10.1371 / journal.pone.0046417
- ^ Конрой Б.Д., Херек Т.А., Шу Т.Д., Латнер М., Ларсон Дж. Дж., Аллен Л. и др. Дизайн, оценка и оценка in vivo вычислительной модели, иллюстрирующей роль CAV1 в Т-лимфоцитах CD4. Фронт Иммунол. 2014; 5: 599 DOI: 10.3389 / fimmu.2014.00599
- ^ «Проект E-Cell».
- ^ «Архивная копия». Архивировано из оригинал на 2010-10-29. Получено 2010-11-09.CS1 maint: заархивированная копия как заголовок (ссылка на сайт)
- ^ Аль-Лазикани, Биссан; Банерджи, Удай; Уоркман, Пол (2012). «Комбинаторная лекарственная терапия рака в постгеномную эпоху». Природа Биотехнологии. 30 (7): 679–692. Дои:10.1038 / nbt.2284. PMID 22781697. S2CID 205277160.
- ^ Айядураи, В.А. Шива; Дьюи, К. Форбс (март 2011 г.). «CytoSolve: масштабируемый вычислительный метод для динамической интеграции моделей множественных молекулярных путей». Cell Mol Bioeng. 4 (1): 28–45. Дои:10.1007 / с12195-010-0143-х. ЧВК 3032229. PMID 21423324.
- ^ http://covertlab.stanford.edu/publicationpdfs/mgenitalium_whole_cell_2012_07_20.pdf[постоянная мертвая ссылка ]
- ^ «Стэнфордские исследователи создают первую полную компьютерную модель организма». 2012-07-19.
- ^ Жерар, Клод; Тайсон, Джон Дж .; Кудрез, Дэмиен; Новак, Бела (06.02.2015). «Контроль клеточного цикла с помощью минимальной сети CDK». PLOS Comput Biol. 11 (2): e1004056. Bibcode:2015PLSCB..11E4056G. Дои:10.1371 / journal.pcbi.1004056. ЧВК 4319789. PMID 25658582.
- ^ Гершон, Дайан (2002). «Кремниевые сны в лаборатории биологии». Природа. 417 (6892): 4–5. Bibcode:2002 Натур.417 .... 4G. Дои:10.1038 / nj6892-04a. PMID 12087360. S2CID 10737442.
