De novo пептидное секвенирование - De novo peptide sequencing - Wikipedia
В масс-спектрометрии, de novo пептидное секвенирование это метод, в котором пептид аминокислота последовательность определяется из тандемная масс-спектрометрия.
Знание аминокислотной последовательности пептидов из протеинового переваривания необходимо для изучения биологической функции протеина. В прежние времена это выполнялось Эдман деградация процедура.[1] Сегодня анализ с помощью тандемного масс-спектрометра является более распространенным методом определения последовательности пептидов. Как правило, существует два подхода: поиск в базе данных и секвенирование de novo. Поиск в базе данных - это простая версия, поскольку данные масс-спектров неизвестного пептида отправляются и запускаются для поиска совпадения с известной пептидной последовательностью, будет выбран пептид с наивысшей оценкой совпадения.[2] Этот подход не позволяет распознать новые пептиды, поскольку он может соответствовать только существующим последовательностям в базе данных. De novo секвенирование - это отнесение фрагментных ионов к масс-спектру. Различные алгоритмы[3]используются для интерпретации, и большинство инструментов поставляются с программами секвенирования de novo.
Фрагментация пептидов
Пептиды протонированный в режиме положительных ионов. Протон первоначально находится в N-конец или боковая цепь основного остатка, но из-за внутренней сольватация, он может двигаться по позвоночнику, ломаясь в разных местах, что приводит к образованию разных фрагментов. Правила фрагментации хорошо объясняются в некоторых публикациях.[4][5][6][7][8][9]
Три различных типа основных связей могут быть разорваны с образованием пептидных фрагментов: алкилкарбонил (CHR-CO), пептидная амидная связь (CO-NH) и аминоалкильная связь (NH-CHR).
Различные типы фрагментных ионов

Когда основные связи расщепляются, образуются шесть различных типов последовательных ионов, как показано на рис. 1. N-концевой заряженные ионы-фрагменты классифицируются как a, b или c, в то время как C-терминал заряженные классифицируются как x, y или z. Нижний индекс n - это количество аминокислотных остатков. Номенклатура была впервые предложена Roepstorff и Fohlman, затем Биманн изменил его, и это стало наиболее широко распространенной версией.[11][12]
Среди этих ионов последовательности a, b и y-ионы являются наиболее распространенными типами ионов, особенно в низкоэнергетических диссоциация, вызванная столкновением (CID) масс-спектрометры, поскольку пептидная амидная связь (CO-NH) является наиболее уязвимой, и потеря CO из b-ионов.
Масса b-ионов = ∑ (массы остатка) + 1 (H+)
Масса y-ионов = (массы остатка) + 19 (H2O + H+)
Масса a-ионов = масса b-ионов - 28 (CO)
Двойное расщепление основной цепи дает внутренние ионы типа ацилия, такие как H2N-CHR2-CO-NH-CHR3-CO + или типа иммония, например H2N-CHR2-CO-NH+= CHR3. Эти ионы обычно являются возмущениями в спектрах.
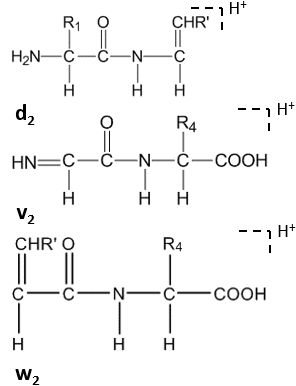
Дальнейшее расщепление происходит под высокоэнергетическим CID в боковой цепи C-концевых остатков, образуя dп, vп, wп-ионы.[8]
Сводка правил фрагментации
Большинство фрагментных ионов являются b- или y-ионами. a-ионы также часто наблюдаются при потере CO из b-ионов.[9]
Сателлитные ионы (Втп, vп, dп-ионы) формируются высокоэнергетическим CID.
Ser-, Thr-, Asp- и Glu-содержащие ионы вызывают нейтральную молекулярную потерю воды (-18).
Ионы, содержащие Asn-, Gln-, Lys-, Arg, вызывают потерю нейтральной молекулы аммиака (-17).
Нейтральная потеря аммиака из Arg приводит к образованию ионов фрагментов (y-17) или (b-17) с более высоким содержанием, чем их соответствующие ионы.
Когда С-конец имеет основной остаток, пептид генерирует (bп-1+18) ион.
Дополнительная пара ионов b-y может наблюдаться в спектрах многозарядных ионов. Для этой ионной пары b-y сумма их индексов равна общему количеству аминокислотных остатков в неизвестном пептиде.
Если C-конец - Arg или Lys, y1-ion можно найти в спектре, чтобы доказать это.
Методы пептидной фрагментации
При низкоэнергетической диссоциации, вызванной столкновениями (CID), b- и y-ионы являются основными продуктами-ионами. Кроме того, потеря аммиака (-17 Да) наблюдается во фрагменте с аминокислотами RKNQ в нем. Потерю воды (-18 Да) можно наблюдать во фрагменте с аминокислотами STED в нем. Ионы-сателлиты в спектрах не показаны.
В высокоэнергетическом CID можно наблюдать все различные типы фрагментных ионов, но без потерь аммиака или воды.
В диссоциация с переносом электрона (ETD) и диссоциация электронного захвата (ECD) преобладающими ионами являются c, y, z + 1, z + 2 и иногда w ионы.
Для распада пост-источника (PSD) в МАЛДИ Ионы a, b, y являются наиболее распространенными ионами-продуктами.
Факторы, влияющие на фрагментацию, - это состояние заряда (чем выше уровень заряда, тем меньше энергии требуется для фрагментации), масса пептида (чем больше масса, тем больше энергии требуется), индуцированная энергия (более высокая энергия приводит к большей фрагментации), первичная аминокислотная последовательность, режим диссоциации и столкновения газов.
Рекомендации по интерпретации
Для интерпретации,[14] во-первых, ищите ионы иммония одной аминокислоты (H2N+= CHR2). Соответствующие ионы иммония для аминокислот перечислены в таблице 1. Не обращайте внимания на несколько пиков в конце спектра с большой массой. Это ионы, которые теряют нейтральные молекулы (H2O, NH3, CO2, HCOOH) из [M + H]+ ионы. Найдите разницу масс при 28 Да, поскольку b-ионы могут образовывать a-ионы за счет потери CO. Ищите b2-ионы в конце спектра с малой массой, что помогает идентифицировать yп-2-ионы тоже. Масса b2-ионы перечислены в таблице 2, а также отдельные аминокислоты, которые имеют массу, равную b2-ионы.[15] Масса b2-ion = масса двух аминокислотных остатков + 1.

Идентифицируйте серию ионов последовательности по той же разнице масс, которая соответствует массе одного из аминокислотных остатков (см. Таблицу 1). Например, разница масс междуп ип-1, бп и бп-1, cп и cп-1 одинаковые. Определить yп-1-ион в самом массовом конце спектра. Затем продолжайте отождествлять yп-2, yп-3... ионов путем сопоставления разницы масс с массами аминокислотных остатков (см. Таблицу 1). Найдите соответствующие b-ионы идентифицированных y-ионов. Масса ионов b + y равна массе пептида +2 Да. После определения серии y-иона и серии b-иона назначьте аминокислотную последовательность и проверьте массу. Другой метод - сначала идентифицировать b-ионы, а затем находить соответствующие y-ионы.
Алгоритмы и ПО
Секвенирование de novo вручную требует больших затрат труда и времени. Обычно для интерпретации спектров применяются алгоритмы или программы, поставляемые с масс-спектрометром.
Разработка алгоритмов секвенирования de novo
Старый метод состоит в том, чтобы перечислить все возможные пептиды для иона-предшественника в масс-спектре и сопоставить масс-спектр для каждого кандидата с экспериментальным спектром. Возможный пептид, который имеет наиболее похожий спектр, будет иметь наибольшие шансы быть правильной последовательностью. Однако количество возможных пептидов может быть большим. Например, пептид-предшественник с молекулярной массой 774 имеет 21 909 046 возможных пептидов. Несмотря на то, что это делается на компьютере, это занимает много времени.[17][18]
Другой метод называется «субсеквенирование», при котором вместо перечисления всей последовательности возможных пептидов сопоставляются короткие последовательности пептидов, которые представляют только часть полного пептида. Когда обнаруживаются последовательности, которые сильно соответствуют ионам фрагментов в экспериментальном спектре, они расширяются остатками один за другим, чтобы найти лучшее совпадение.[19][20][21][22]
В третьем методе применяется графическое отображение данных, в котором ионы фрагментов, которые имеют одинаковую разность масс одного аминокислотного остатка, соединяются линиями. Таким образом легче получить четкое изображение ионных рядов одного типа. Этот метод может быть полезен для ручного секвенирования пептидов de novo, но не работает в условиях высокой производительности.[23]
Четвертый метод, который считается успешным, - это теория графов. Применение теории графов при секвенировании пептидов de novo было впервые упомянуто Бартелсом.[24] Пики в спектре преобразуются в вершины в графе, называемом «графом спектра». Если две вершины имеют одинаковую разность масс одной или нескольких аминокислот, будет применено направленное ребро. Алгоритм SeqMS,[25] Алгоритм Лютефиск,[26] Алгоритм Шеренги[27] несколько примеров этого типа.
Программные пакеты
По словам Андреотти и другие. в 2012,[28] Антилопа - это комбинация лагранжевой релаксации и адаптации k кратчайших путей Йена. Он основан на методе «спектрального графика» и содержит различные функции оценки, и может быть сопоставим по времени работы и точности с «популярными уровень развития программы «ПепНово» и «НовоХММ.
Гроссманн и другие.[29] представили AUDENS в 2005 году как автоматизированный инструмент для определения последовательности пептидов de novo, содержащий модуль предварительной обработки, который может распознавать пики сигнала и пики шума.
Lutefisk может решить de novo секвенирование по масс-спектрам CID. В этом алгоритме сначала обнаруживаются значимые ионы, а затем определяется список доказательств N- и C-конца. На основе списка последовательностей он генерирует полные последовательности в спектрах и сравнивает их с экспериментальным спектром. Однако результат может включать несколько возможных последовательностей, которые имеют лишь небольшое различие, поэтому трудно найти правильную пептидную последовательность. Вторая программа, CIDentify, которая представляет собой модифицированную версию алгоритма FASTA Билла Пирсона Алекса Тейлора, может применяться для различения этих сомнительных похожих кандидатов.
Пн и другие. представил алгоритм MSNovo в 2007 году и доказал, что он работает «лучше, чем существующие инструменты de novo на нескольких наборах данных».[30] Этот алгоритм может выполнять интерпретацию de novo секвенирования масс-спектрометров LCQ, LTQ и одно-, двух-, трехзарядных ионов. В отличие от других алгоритмов, он применяет новую функцию оценки и использует массив масс вместо графика спектра.
Фишер и другие.[31] предложили NovoHMM метод секвенирования de novo. Скрытая марковская модель (HMM) применяется как новый способ решения de novo секвенирования в байесовской структуре. Вместо оценки отдельных символов последовательности этот метод рассматривает апостериорные вероятности для аминокислот. В статье на множестве примеров спектров доказано, что этот метод имеет лучшую производительность, чем другие популярные методы секвенирования пептидов de novo, такие как PepNovo.
ПИКС представляет собой полный пакет программ для интерпретации масс-спектров пептидов. Он включает секвенирование de novo, поиск в базе данных, идентификацию PTM, поиск гомологии и количественную оценку при анализе данных. Ma et al. описал новую модель и алгоритм для секвенирования de novo в PEAKS и сравнил эффективность с Lutefisk нескольких триптических пептидов стандартных белков, квадруполь время полета (Q-TOF) масс-спектрометр.[32]
PepNovo - это высокопроизводительный инструмент для определения последовательности пептидов de novo, использующий вероятностную сеть в качестве метода оценки. Обычно интерпретация одного спектра занимает менее 0,2 секунды. Описано Фрэнком и другие., PepNovo работает лучше нескольких популярных алгоритмов, таких как Sherenga, PEAKS, Lutefisk.[33] Теперь доступна новая версия PepNovo +.
Чи и другие. представили pNovo + в 2013 году как новый инструмент для определения последовательности пептидов de novo с использованием дополнительных тандемных масс-спектров HCD и ETD.[34] В этом методе компонентный алгоритм pDAG в значительной степени ускоряет время получения пептидного секвенирования в среднем до 0,018 с, что в три раза быстрее, чем в другом популярном программном обеспечении для секвенирования de novo.
Как описал Чон и другие.По сравнению с другими инструментами для секвенирования пептидов do novo, которые хорошо работают только с определенными типами спектров, UniNovo является более универсальным инструментом, который имеет хорошую производительность для различных типов спектров или спектральных пар, таких как CID, ETD, HCD, CID / ETD, и т.д. Он имеет лучшую точность, чем PepNovo + или PEAKS. Более того, он генерирует частоту ошибок в указанных пептидных последовательностях.[35]
Ма опубликовал Novor в 2015 году в качестве механизма для определения пептидов de novo в реальном времени. Этот инструмент призван повысить скорость de novo на порядок и сохранить такую же точность, как и другие инструменты de novo на рынке. На ноутбуке Macbook Pro Новор достиг более 300 спектров МС / МС в секунду.[36]
Певцов и др. сравнили производительность пяти вышеуказанных алгоритмов секвенирования de novo: AUDENS, Lutefisk, NovoHMM, PepNovo и PEAKS. В анализе использовали данные масс-спектрометров QSTAR и LCQ, которые оценивали по значению относительного расстояния между последовательностями (RSD), которое представляло собой сходство между пептидным секвенированием de novo и истинной пептидной последовательностью, рассчитанной методом динамического программирования. Результаты показали, что все алгоритмы показали лучшую производительность в данных QSTAR, чем в данных LCQ, в то время как PEAKS как лучший имел показатель успешности 49,7% в данных QSTAR, а NovoHMM как лучший имел показатель успеха 18,3% в данных LCQ. Порядок производительности в данных QSTAR был PEAKS> Lutefisk, PepNovo> AUDENS, NovoHMM, а в данных LCQ был NovoHMM> PepNovo, PEAKS> Lutefisk> AUDENS. По сравнению с диапазоном качества спектра PEAKS и NovoHMM также показали лучшую производительность по обоим данным среди всех 5 алгоритмов. PEAKS и NovoHMM также показали лучшую чувствительность как по данным QSTAR, так и по данным LCQ. Однако ни один из оцененных алгоритмов не превышал 50% точной идентификации для обоих наборов данных.[37]
Рекомендации
- ^ Эдман, П .; Бегг, Г. (март 1967). «Белковый секвенатор». Европейский журнал биохимии. 1 (1): 80–91. Дои:10.1111 / j.1432-1033.1967.tb00047.x. PMID 6059350.
- ^ Уэбб-Робертсон, Б.-Дж. М .; Кэннон, У. Р. (20 июня 2007 г.). «Современные тенденции в области компьютерных выводов на основе протеомики на основе масс-спектрометрии» (PDF). Брифинги по биоинформатике. 8 (5): 304–317. Дои:10.1093 / нагрудник / bbm023. PMID 17584764.
- ^ Лу, Бингвен; Чен, Тинг (март 2004 г.). «Алгоритмы для секвенирования пептидов de novo с использованием тандемной масс-спектрометрии». Открытие лекарств сегодня: BIOSILICO. 2 (2): 85–90. Дои:10.1016 / S1741-8364 (04) 02387-X.
- ^ а б Папаяннопулос, Иоаннис А. (январь 1995 г.). «Интерпретация тандемных масс-спектров пептидов при диссоциации, вызванной столкновением». Обзоры масс-спектрометрии. 14 (1): 49–73. Bibcode:1995MSRv ... 14 ... 49P. Дои:10.1002 / mas.1280140104.
- ^ Дасс, Чхабил; Дезидерио, Доминик М. (май 1987 г.). "Масс-спектрометрический анализ с бомбардировкой быстрыми атомами опиоидных пептидов". Аналитическая биохимия. 163 (1): 52–66. Дои:10.1016/0003-2697(87)90092-3. PMID 2887130.
- ^ Ялчин, Талат; Csizmadia, Imre G .; Петерсон, Майкл Р .; Харрисон, Алекс Г. (март 1996 г.). «Структура и фрагментация ионов B n (n≥3) в пептидных спектрах». Журнал Американского общества масс-спектрометрии. 7 (3): 233–242. Дои:10.1016 / 1044-0305 (95) 00677-X. PMID 24203294.
- ^ Тан, Сюэ-Цзюнь; Бойд, Роберт К .; Бертран, М. Дж. (Ноябрь 1992 г.). «Исследование механизмов фрагментации дважды протонированных триптических пептидов». Быстрые коммуникации в масс-спектрометрии. 6 (11): 651–657. Bibcode:1992RCMS .... 6..651T. Дои:10.1002 / RCM.1290061105. PMID 1467549.
- ^ а б c Джонсон, Ричард С .; Мартин, Стивен А .; Биманн, Клаус (декабрь 1988 г.). "Индуцированная столкновением фрагментация (M + H) + ионов пептидов. Ионы специфической последовательности боковой цепи". Международный журнал масс-спектрометрии и ионных процессов. 86: 137–154. Bibcode:1988IJMSI..86..137J. Дои:10.1016/0168-1176(88)80060-0.
- ^ а б Дасс, Чхабил (2007). Основы современной масс-спектрометрии ([Online-Ausg.]. Ред.). Хобокен, штат Нью-Джерси: Wiley-Interscience. С. 317–322. Дои:10.1002/0470118490. ISBN 9780470118498.
- ^ Дасс, Чхабил (2001). Принципы и практика биологической масс-спектрометрии. Нью-Йорк, штат Нью-Йорк [u.a.]: Wiley. ISBN 978-0-471-33053-0.
- ^ Roepstorff, P; Фохлман, Дж (ноябрь 1984 г.). «Предложение по общей номенклатуре последовательностей ионов в масс-спектрах пептидов». Биомедицинская масс-спектрометрия. 11 (11): 601. Дои:10.1002 / bms.1200111109. PMID 6525415.
- ^ Макклоски, отредактированный Джеймсом А. (1990). Масс-спектрометрии. Сан-Диего: Academic Press. С. 886–887. ISBN 978-0121820947.CS1 maint: дополнительный текст: список авторов (связь)
- ^ Falick, A.M .; Hines, W. M .; Medzihradszky, K. F .; Болдуин, М. А .; Гибсон, Б. У. (ноябрь 1993 г.). «Ионы малой массы, полученные из пептидов в результате диссоциации, вызванной столкновениями высоких энергий, в тандемной масс-спектрометрии». Журнал Американского общества масс-спектрометрии. 4 (11): 882–893. Дои:10.1016 / 1044-0305 (93) 87006-X. PMID 24227532.
- ^ Дасс, Чхабил (2007). Основы современной масс-спектрометрии ([Online-Ausg.]. Ред.). Хобокен, штат Нью-Джерси: Wiley-Interscience. С. 327–330. ISBN 9780470118498.
- ^ Харрисон, Алекс G .; Csizmadia, Imre G .; Тан, Тин-Хуа (май 2000 г.). «Структура и фрагментация b2 ионы в масс-спектрах пептидов ». Журнал Американского общества масс-спектрометрии. 11 (5): 427–436. Дои:10.1016 / S1044-0305 (00) 00104-5. PMID 10790847.
- ^ Дасс, Чхабил (2007). Основы современной масс-спектрометрии ([Online-Ausg.]. Ред.). Хобокен, штат Нью-Джерси: Wiley-Interscience. п. 329. ISBN 9780470118498.
- ^ Сакураи, Т .; Matsuo, T .; Matsuda, H .; Катакусе И. (август 1984 г.). «PAAS 3: компьютерная программа для определения вероятной последовательности пептидов по масс-спектрометрическим данным». Биологическая масс-спектрометрия. 11 (8): 396–399. Дои:10.1002 / bms.1200110806.
- ^ Hamm, C.W .; Wilson, W. E .; Харван, Д. Дж. (1986). «Программа секвенирования пептидов». Биоинформатика. 2 (2): 115–118. Дои:10.1093 / биоинформатика / 2.2.115.
- ^ Биманн, К; Конус, C; Вебстер, BR; Арсено, ГП (5 декабря 1966 г.). «Определение аминокислотной последовательности в олигопептидах путем компьютерной интерпретации их масс-спектров высокого разрешения». Журнал Американского химического общества. 88 (23): 5598–606. Дои:10.1021 / ja00975a045. PMID 5980176.
- ^ Ishikawa, K .; Нива, Ю. (июль 1986 г.). «Компьютерное секвенирование пептидов методом масс-спектрометрии с бомбардировкой быстрыми атомами». Биологическая масс-спектрометрия. 13 (7): 373–380. Дои:10.1002 / bms.1200130709.
- ^ Сигель, ММ; Бауман, Н. (15 марта 1988 г.). «Эффективный алгоритм для секвенирования пептидов с использованием масс-спектральных данных бомбардировки быстрыми атомами». Биомедицинская и экологическая масс-спектрометрия. 15 (6): 333–43. Дои:10.1002 / bms.1200150606. PMID 2967723.
- ^ Джонсон, РС; Биманн, К. (ноябрь 1989 г.). «Компьютерная программа (SEQPEP) для помощи в интерпретации тандемных масс-спектров пептидов при столкновении высоких энергий». Биомедицинская и экологическая масс-спектрометрия. 18 (11): 945–57. Дои:10.1002 / bms.1200181102. PMID 2620156.
- ^ Scoble, Hubert A .; Биллер, Джеймс Э .; Биманн, Клаус (1987). «Ориентированная на графическое отображение стратегия для аминокислотного секвенирования пептидов с помощью тандемной масс-спектрометрии». Fresenius 'Zeitschrift für Analytische Chemie. 327 (2): 239–245. Дои:10.1007 / BF00469824.
- ^ Бартельс, Кристиан (июнь 1990 г.). «Быстрый алгоритм определения пептидов методом масс-спектроскопии». Биологическая масс-спектрометрия. 19 (6): 363–368. Дои:10.1002 / bms.1200190607. PMID 24730078.
- ^ Фернандес-де-Коссио, Дж .; Гонсалес, Дж; Бесада, V (август 1995 г.). «Компьютерная программа для помощи в секвенировании пептидов в экспериментах по разложению при столкновении». Компьютерные приложения в биологических науках (CABIOS). 11 (4): 427–34. Дои:10.1093 / биоинформатика / 11.4.427. PMID 8521052.
- ^ Тейлор, JA; Джонсон, RS (1997). «Поиск в базе данных последовательностей посредством секвенирования пептидов de novo с помощью тандемной масс-спектрометрии». Быстрые коммуникации в масс-спектрометрии. 11 (9): 1067–75. Bibcode:1997RCMS ... 11.1067T. Дои:10.1002 / (sici) 1097-0231 (19970615) 11: 9 <1067 :: aid-rcm953> 3.0.co; 2-l. PMID 9204580.
- ^ Данчик, Владо; Аддона, Тереза А .; Clauser, Karl R .; Vath, Джеймс Э .; Певзнер, Павел А. (октябрь 1999 г.). «Секвенирование пептидов с помощью тандемной масс-спектрометрии». Журнал вычислительной биологии. 6 (3–4): 327–342. CiteSeerX 10.1.1.128.2645. Дои:10.1089/106652799318300. PMID 10582570.
- ^ Андреотти, S; Клау, GW; Райнерт, К. (2012). «Антилопа - подход лагранжевой релаксации к проблеме секвенирования пептидов de novo». IEEE / ACM Transactions по вычислительной биологии и биоинформатике. 9 (2): 385–94. arXiv:1102.4016. Дои:10.1109 / tcbb.2011.59. PMID 21464512.
- ^ Гроссманн, Дж; Роос, ФФ; Cieliebak, M; Lipták, Z; Матис, LK; Мюллер, М; Груиссем, Вт; Багинский, С (2005). «AUDENS: инструмент для автоматического секвенирования пептидов de novo». Журнал протеомных исследований. 4 (5): 1768–74. CiteSeerX 10.1.1.654.169. Дои:10.1021 / pr050070a. PMID 16212431.
- ^ Мо, L; Dutta, D; Ван, Й; Чен, Т. (1 июля 2007 г.). «MSNovo: алгоритм динамического программирования для секвенирования пептидов de novo с помощью тандемной масс-спектрометрии». Аналитическая химия. 79 (13): 4870–8. Дои:10.1021 / ac070039n. PMID 17550227.
- ^ Фишер, Б. Рот, V; Роос, Ф; Гроссманн, Дж; Багинский, С; Widmayer, P; Груиссем, Вт; Бухманн, JM (15 ноября 2005 г.). «NovoHMM: скрытая марковская модель для секвенирования пептидов de novo». Аналитическая химия. 77 (22): 7265–73. CiteSeerX 10.1.1.507.1610. Дои:10.1021 / ac0508853. PMID 16285674.
- ^ Ма, Бин; Чжан, Кайчжун; Хендри, Кристофер; Лян, Чэнчжи; Ли, Мин; Доэрти-Кирби, Аманда; Лажуа, Жиль (30 октября 2003 г.). «ПИКС: мощное программное обеспечение для секвенирования пептидов novo методом тандемной масс-спектрометрии». Быстрые коммуникации в масс-спектрометрии. 17 (20): 2337–2342. Bibcode:2003RCMS ... 17.2337M. Дои:10.1002 / RCM.1196. PMID 14558135.
- ^ Франк, А; Певзнер, П. (15 февраля 2005 г.). «PepNovo: de novo пептидное секвенирование с помощью вероятностного сетевого моделирования». Аналитическая химия. 77 (4): 964–73. Дои:10.1021 / ac048788h. PMID 15858974.
- ^ Chi, H; Чен, Н; Он, К; Wu, L; Ян, Б; Солнце, RX; Лю, Дж; Цзэн, ВФ; Песня, CQ; Он, СМ; Донг, MQ (1 февраля 2013 г.). «pNovo +: de novo пептидное секвенирование с использованием комплементарных тандемных масс-спектров HCD и ETD». Журнал протеомных исследований. 12 (2): 615–25. Дои:10.1021 / pr3006843. PMID 23272783.
- ^ Jeong, K; Ким, S; Певзнер, П.А. (15 августа 2013 г.). «UniNovo: универсальный инструмент для секвенирования пептидов de novo». Биоинформатика. 29 (16): 1953–62. Дои:10.1093 / биоинформатика / btt338. ЧВК 3722526. PMID 23766417.
- ^ Ма, Бин (30 июня 2015 г.). "Novor: программное обеспечение для секвенирования пептидов de Novo в реальном времени". Журнал Американского общества масс-спектрометрии. 26 (11): 1885–1894. Bibcode:2015JASMS..26.1885M. Дои:10.1007 / s13361-015-1204-0. ЧВК 4604512. PMID 26122521.
- ^ Певцов, С .; Федулова, И .; Mirzaei, H .; Бак, С .; Чжан, X. (2006). «Оценка эффективности существующих Де Ново Последовательность действий Алгоритмы ». Журнал протеомных исследований. 5 (11): 3018–3028. Дои:10.1021 / pr060222h. PMID 17081053.

