ГВОЗДЬ-MS - NAIL-MS
ГВОЗДЬ-MS (Короче для нуклеиновая кислота изотоп маркировка в сочетании масс-спектрометрии ) представляет собой метод, основанный на масс-спектрометрии, используемый для исследования нуклеиновых кислот и их модификации. Это позволяет использовать различные схемы экспериментов для изучения основного механизма биологии РНК. in vivo. Например, динамическое поведение нуклеиновых кислот в живых клетках, особенно Модификации РНК, можно проследить более подробно.[1]
Теория
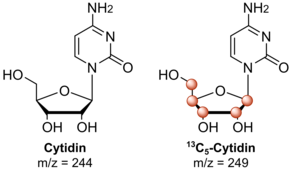
NAIL-MS используется для изучения механизмов модификации РНК. Таким образом, клетки в культуре сначала кормят питательными веществами, меченными стабильными изотопами, и клетки включают их в свои биомолекулы. После очистки нуклеиновых кислот чаще всего РНК, анализ проводится масс-спектрометрией. Масс-спектрометрия - это аналитический метод измерения отношение массы к заряду из ионы. Пары химически идентичных нуклеозидов с различным стабильным изотопным составом можно различать в масс-спектрометре по разнице масс. Без ярлыка нуклеозиды поэтому можно отличить от их стабильного изотопа, помеченного изотопологи. Для большинства подходов NAIL-MS критически важно, чтобы меченые нуклеозиды были более 2 Да тяжелее немаркированных. Это потому, что 1,1% встречающихся в природе атомов углерода 13Изотопы C. В случае нуклеозидов это приводит к увеличению массы на 1 Да в ~ 10% нуклеозидов. Этот сигнал может помешать окончательной оценке измерения.
NAIL-MS можно использовать для исследования динамики модификации РНК путем изменения меченых питательных веществ соответствующей питательной среды во время эксперимента. Кроме того, популяции клеток можно сравнивать напрямую друг с другом без влияния ошибки очистки. Кроме того, его можно использовать для производства биосинтетических изотопологов большинства нуклеозидов, которые необходимы для количественной оценки масс-спектрометрией и даже для открытия еще неизвестных модификаций РНК.[2][3][4]
Общая процедура

Обычно клетки культивируют в немеченой или стабильной (нерадиоактивной) среде, меченной изотопом. Например, среда может содержать глюкозу, помеченную шестью углерод-13 атомы (13В) вместо нормального углерод-12 (12C). Клетки, растущие в этой среде, будут, в зависимости от модельного организма, включать тяжелую глюкозу во все свои молекулы РНК. После этого все нуклеотиды на 5 Да тяжелее их немеченых изотопологов из-за полного углеродного мечения рибозы. После культивирования и соответствующей маркировки клеток их обычно собирают, используя фенол / хлороформ / изотиоцианат гуанидиния. Возможны и иногда необходимы другие методы экстракции (например, для дрожжей). Затем РНК выделяется Фенол-хлороформная экстракция и iso-Пропанол осадки. Дальнейшая очистка конкретных видов РНК (например, рРНК, тРНК) обычно выполняется с помощью эксклюзионная хроматография (SEC), но доступны и другие подходы. Для большинства приложений конечный продукт необходимо ферментативно расщепить до нуклеозидов перед анализом с помощью ЖХ-МС. Следовательно, ферменты пищеварения, такие как бензоназа, NP1 и CIP используются.[5][6] Обычно тройной квадруполь в режиме MRM используется для измерений.
Маркировка ячеек
Как достигается маркировка молекул РНК, зависит от модельного организма. За Кишечная палочка (бактерии ) можно использовать минимальную среду M9 и дополнить ее вариантами необходимых солей, меченными стабильными изотопами. Это позволяет маркировать 13C-углерод, 15N-азот, 34S-сера и 2Н-водород.[7] В S.cerevisiae (дрожжи ) в настоящее время есть две возможности: во-первых, использование коммерчески доступной полной питательной среды, которая позволяет маркировать 13C-углерод и / или 15N-азот и, во-вторых, использование минимальной среды YNB, которая должна быть дополнена несколькими аминокислоты и глюкоза которые могут быть добавлены в виде вариантов, меченных стабильными изотопами, для достижения 13C-углерод, 15N-азот и 2H-водородное маркирование РНК.[8]
При маркировке в модельных организмах, таких как Кишечная палочка и S.cerevisiae достаточно простое, стабильное мечение изотопа в культура клеток намного сложнее, поскольку состав питательной среды более сложен. Ни добавление глюкозы, меченной стабильным изотопом, ни добавление меченых стабильным изотопом вариантов простых предшественников биосинтеза нуклеозидов, таких как глутамин и / или аспартат приводят к определенному увеличению массы более чем на 2 Да. Хотя подходящие соединения для полного мечения в культуре клеток уже найдены, эти результаты еще не опубликованы.
Приложения
С NAIL-MS возможны различные планы экспериментов.
Производство СИЛИС
NAIL-MS можно использовать для производства внутренних стандартов, меченных стабильными изотопами (ISTD). Следовательно, клетки выращивают в среде, которая приводит к полному мечению всех нуклеозидов. Очищенную смесь нуклеозидов затем можно использовать в качестве ISTD, который необходим для точного абсолютного количественного определения нуклеозидов с помощью масс-спектрометрии. Эта смесь меченых нуклеозидов также называется SILIS (внутренний стандарт, меченный стабильным изотопом).[9] Преимущество этого подхода состоит в том, что все модификации, присутствующие в организме, могут быть биосинтезированы в виде меченых соединений. Производство SILIS производилось еще до появления термина NAIL-MS.
Сравнительные эксперименты
Сравнительный эксперимент NAIL-MS очень похож на СИЛАК экспериментируйте, но для РНК вместо белков. Сначала культивируют две популяции соответствующих клеток. Одна из популяций клеток питается питательной средой, содержащей немеченые питательные вещества, тогда как вторая популяция питается питательной средой, содержащей меченые стабильными изотопами питательные вещества. Затем клетки включают соответствующие изотопологи в свои молекулы РНК. Одна из популяций клеток служит контрольной группой, тогда как другая является предметом сопутствующих исследований (например, напряжение нокаута, стресс). После сбора двух популяций клеток их смешивают и совместно обрабатывают, чтобы исключить систематическую ошибку очистки. Благодаря разным массам включенных в нуклеозиды питательных веществ возможна дифференциация двух популяций клеток с помощью масс-спектрометрии.
Pulse-Chase эксперименты
При запуске эксперимента с отслеживанием импульсов среда переключается с среды (1) на среду (2). Эти две среды должны различаться только по содержанию изотопов. Таким образом, можно различать молекулы РНК, уже существующие до начала эксперимента (= молекулы РНК, выращенные в среде (1)), и молекулы РНК, которые вновь транскрибируются после начала эксперимента (= молекулы РНК, выращенные в среде (2)). Это позволяет детально изучить динамику модификации. in vivo. Добавление меченого метионина либо в среду (1), либо в среду (2) позволяет отслеживать процессы метилирования. Другие метаболиты, меченные изотопами, потенциально допускают дальнейший анализ модификации.
В целом NAIL-MS позволяет исследовать динамику модификации РНК с помощью масс-спектрометрии. С помощью этого метода ферментативное деметилирование наблюдалось при нескольких повреждениях РНК внутри живых бактерий.[4][7]
Открытие новых модификаций РНК
Для обнаружения не охарактеризованных модификаций клетки выращивают в немеченых или же 13C ‑ маркированный или же 15N ‑ помечен или же 2H ‑ маркированный или же 34Среда с меткой S. Неизвестные сигналы, возникающие во время масс-спектрометрии, затем исследуются во всех культурах с разными метками. Если время удерживания неизвестных соединений с соответствующим образом различающимся м / з Значения перекрываются, формула суммы соединения может быть постулирована путем вычисления разницы масс перекрывающегося сигнала в культурах с различной меткой. С помощью этого метода можно было обнаружить несколько новых модификаций РНК. Этот экспериментальный дизайн также был первоначальной идеей, положившей начало концепции NAIL-MS.
Олигонуклеотид NAIL-MS
NAIL-MS также можно применять для анализа олигонуклеотидов с помощью масс-спектрометрии. Это полезно, когда необходимо сохранить информацию о последовательности.[10]
Рекомендации
- ^ Райхле, Валентин Ф .; Кайзер, Штеффен; Хейсс, Матиас; Хагельскэмп, Феликс; Борланд, Кайла; Келлнер, Стефани (1 марта 2019 г.). «Превосходя пределы статического анализа модификации РНК с динамическим NAIL-MS». Методы (Сан-Диего, Калифорния).. 156: 91–101. Дои:10.1016 / j.ymeth.2018.10.025. ISSN 1095-9130. PMID 30395967.
- ^ Келлнер, Стефани; Нойман, Дженнифер; Розенкранц, Дэвид; Лебедева, Светлана; Кеттинг, Рене Ф .; Цишлер, Ганс; Шнайдер, Дирк; Хельм, Марк (4 апреля 2014 г.). «Профилирование модификаций РНК с помощью мультиплексного мечения стабильных изотопов». Chemical Communications (Кембридж, Англия). 50 (26): 3516–3518. Дои:10.1039 / c3cc49114e. ISSN 1364-548X. PMID 24567952.
- ^ Даль Магро, Кристина; Келлер, Патрик; Коттер, Анника; Вернер, Стефан; Дуарте, Виктор; Маршан, Вирджиния; Игнарский, Майкл; Фрейвальд, Аня; Мюллер, Роман-Ульрих; Дитрих, Кристоф; Моторин, Юрий (25 июня 2018 г.). «Значительно увеличившееся химическое разнообразие модификаций РНК, содержащих тиоацетальную структуру». Angewandte Chemie International Edition на английском языке. 57 (26): 7893–7897. Дои:10.1002 / anie.201713188. ISSN 1521-3773. PMID 29624844.
- ^ а б Райхле, Валентин Ф .; Петров, Димитар П .; Вебер, Верена; Юнг, Кирстен; Келлнер, Стефани (6 декабря 2019 г.). «NAIL-MS обнаруживает восстановление 2-метилтиоцитидина с помощью AlkB в E. coli». Nature Communications. 10 (1): 5600. Bibcode:2019НатКо..10.5600р. Дои:10.1038 / s41467-019-13565-9. ISSN 2041-1723. ЧВК 6898146. PMID 31811240.
- ^ Quinlivan, Eoin P .; Грегори, Джесси Ф. (15 февраля 2008 г.). «Расщепление ДНК до дезоксирибонуклеозида: упрощенная одноэтапная процедура». Аналитическая биохимия. 373 (2): 383–385. Дои:10.1016 / j.ab.2007.09.031. ISSN 0003-2697. ЧВК 2239294. PMID 18028864.
- ^ Крейн, П. Ф. (1990). «Подготовка и ферментативный гидролиз ДНК и РНК для масс-спектрометрии». Методы в энзимологии. 193: 782–790. Дои:10.1016 / 0076-6879 (90) 93450-у. ISSN 0076-6879. PMID 1706062.
- ^ а б Райхле, Валентин Ф .; Вебер, Верена; Келлнер, Стефани (18 декабря 2018 г.). «NAIL-MS в E. coli определяет источник и судьбу метилирования тРНК». Chembiochem: Европейский журнал химической биологии. 19 (24): 2575–2583. Дои:10.1002 / cbic.201800525. ISSN 1439-7633. ЧВК 6582434. PMID 30328661.
- ^ Хейсс, Матиас; Райхле, Валентин Ф .; Келлнер, Стефани (2 сентября 2017 г.). «Наблюдение за судьбой тРНК и ее модификаций с помощью масс-спектрометрии с мечением изотопов нуклеиновых кислот: NAIL-MS». РНК Биология. 14 (9): 1260–1268. Дои:10.1080/15476286.2017.1325063. ISSN 1555-8584. ЧВК 5699550. PMID 28488916.
- ^ Келлнер, Стефани; Очел, Антония; Тюринг, Катрин; Спенкуч, Феликс; Нойман, Дженнифер; Шарма, Санни; Энтиан, Карл-Дитер; Шнайдер, Дирк; Хельм, Марк (16 августа 2014 г.). «Абсолютное и относительное количественное определение модификаций РНК через биосинтетические изотопомеры». Исследования нуклеиновых кислот. 42 (18): e142. Дои:10.1093 / нар / gku733. ISSN 1362-4962. ЧВК 4191383. PMID 25129236.
- ^ Хагельскэмп, Феликс; Борланд, Кайла; Рамос, Джиллиан; Хендрик, Алан Дж .; Фу, Драгони; Келлнер, Стефани (21 февраля 2020 г.). «Широко применимая олигонуклеотидная масс-спектрометрия для анализа писателей и стирателей РНК in vitro». Исследования нуклеиновых кислот. 48 (7): e41. Дои:10.1093 / nar / gkaa091. ISSN 1362-4962. ЧВК 7144906. PMID 32083657.
