Анализ метилирования Illumina - Illumina Methylation Assay
В Иллюмина Анализ метилирования с использованием Инфиниум I платформа использует технологию BeadChip[требуется разъяснение ] для создания всеобъемлющего геном -широкое профилирование человека Метилирование ДНК. Похожий на бисульфитное секвенирование и пиросеквенирование, этот метод количественно метилирование уровни на различных места в пределах геном. Этот анализ используется для зондов метилирования на чипе Illumina Infinium HumanMethylation27 BeadChip (далее - 27k [метилирование]). Зонды на 27k целевых регионах массива человеческий геном для измерения уровней метилирования на 27 578 Динуклеотиды CpG в 14 495 генах.[1] В Infinium Human Метилирование 450 BeadChip массив нацелен на> 450 000 сайтов метилирования.[2]
Фон
Метилирование ДНК играет важную роль в эпигенетический регулирование хроматин структура, которая в последнее десятилетие была признана важной в регулировании экспрессия гена, развитие и генетический импринтинг у позвоночных.[1] Было показано, что изменения в структуре и уровне метилирования способствуют рак и различные болезни развития.[3] Например, гиперметилирование на промоторных CpG-островках ген-супрессор опухоли, что, в свою очередь, приводит к заглушить, часто ассоциируется с опухоль.[3] Крупномасштабное измерение паттернов метилирования ДНК из широкого выбора гены может помочь нам лучше понять взаимосвязь между эпигенетическими изменениями и генезом различных заболеваний и лучше понять роль, которую эпигенетика играет в тканеспецифических дифференциация.

Материал
Чип метилирования Illumina 27k содержит 27 578 отдельных сайтов CpG, распределенных по 14 495 генам.[1] Эти гены включают гены RefSeq из базы данных NCBI CCDS, гены рака, которые демонстрируют различные паттерны метилирования во время своего развития, и промоторы микроРНК.[4] Маркеры, включенные в чип, приведены в таблице 1.[4]
Метод
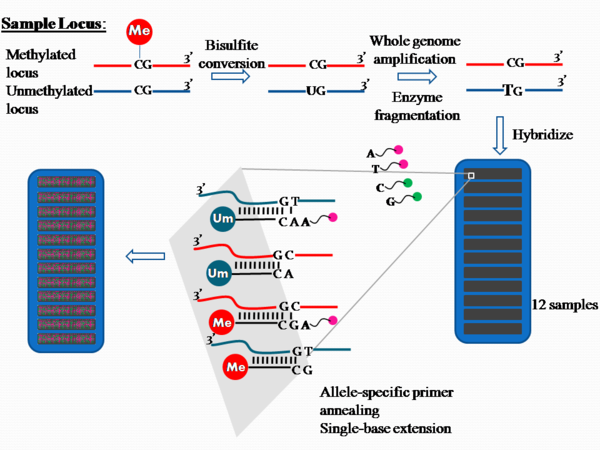
Для технологии химического анализа Infinium I процесс показан на рисунке 1.
Бисульфитная обработка
Приблизительно 1 мкг геномной ДНК используется в превращении бисульфита для преобразования неметилированного цитозин в урацил. Продукт содержит непревращенный цитозин там, где они были ранее метилированы, но цитозин преобразован в урацил, если они ранее не были метилированы.
Амплификация цельной геномной ДНК
Обработанная бисульфитом ДНК подвергается полногеномная амплификация (WGA) с помощью случайного праймирования гексамеров и Phi29 ДНК-полимераза, который имеет корректирующую деятельность, в результате чего коэффициент ошибок в 100 раз ниже, чем Полимераза Taq. Затем продукты ферментативно фрагментируются,[1] очищены от дНТФ, праймеров и ферментов и нанесены на чип.[5]
Гибридизация и расширение на одной базе
На микросхеме есть два типа шариков для каждого CpG (или "CG", как показано на рисунке 1) сайта на локус. Каждый тестируемый локус отличается по типу гранул.[1] Оба типа бусинок прикреплены к одноцепочечной 50-мерной ДНК. олигонуклеотиды которые отличаются последовательностью только на свободном конце; этот тип зонда известен как аллель-специфический олигонуклеотид. Один из типов бусинок будет соответствовать метилированному цитозиновому локусу, а другой - неметилированному цитозиновому локусу, который был преобразован в урацил во время обработки бисульфитом, а затем амплифицирован в виде тимина во время амплификации всего генома. денатурируются в одноцепочечные и гибридизуются с чипом посредством аллель-специфичных отжиг либо к зонду, специфичному для метилирования, либо к зонду неметилирования. За гибридизацией следует расширение на одно основание с гаптен меченые дидезоксинуклеотиды. DdCTP и ddGTP помечены биотин в то время как ddATP и ddUTP помечены 2,4-динитрофенол (DNP).[6]
Флуоресцентное окрашивание и сканирование чипа
После включения этих меченных гаптеном ддНТФ, многослойный иммуногистохимический Анализы выполняются путем повторения циклов окрашивания комбинацией антител для дифференциации двух типов.[6] После окрашивания чип сканируется, чтобы показать интенсивности неметилированных и метилированных гранул. (Фигура 2).[1] Необработанные данные анализируются запатентованным программным обеспечением, и рассчитываются отношения интенсивности флуоресценции между двумя типами гранул. Для данного индивида в данном локусе значение отношения 0 равно неметилированию локуса (т.е., гомозиготный неметилированный); соотношение 1 равно общему метилированию (т.е., гомозиготный метилированный); а значение 0,5 означает, что одна копия метилирована, а другая нет (т.е., гетерозиготность) в диплоидном геноме человека.[1][4]

Анализ данных метилирования
Отсканированный микрочип изображения данных метилирования далее анализируются системой, которая нормализует необработанные данные для уменьшения влияния экспериментальной вариации, фоновой и средней нормализации, а также выполняет стандартные статистические тесты результатов.[7] Затем данные можно объединить в несколько типов фигур для визуализации и анализа. Диаграммы разброса используются для корреляции данных метилирования; гистограммы для визуализации относительных уровней метилирования на каждом тестируемом сайте; тепловые карты для кластеризации данных для сравнения профиля метилирования на тестируемых сайтах. На рисунке 2 показаны различные типы полученных результатов.
Преимущества и недостатки
Преимущества
- Нет ПЦР требуется, что означает отсутствие избирательного смещения в сторону более коротких фрагментов.[4]
- Возможность исследования до 12 образцов на чип обеспечивает высокую производительность обработки.[4]
- Позволяет интегрировать данные между другими платформами, такими как экспрессия гена и микроРНК профилирование.[4]
- Метод рассматривает ~ 2 сайта CpG на каждый островок CpG, обеспечивая охват паттернов метилирования по всему геному.
Недостатки
- Не все гены аннотированы в NCBI База данных была включена в дизайн этого анализа, который охватывает 14 495 генов из 17 052 генов, имеющихся на сегодняшний день (сборка человека 36.3).[8]
- По данным Staaf et al. (2008),[9] «Похоже, что анализ Infinium II показал смещение интенсивности красителя между двумя каналами, используемыми для обнаружения флуоресценции. Более того, это смещение не было устранено даже после того, как данные прошли через алгоритмы нормализации, используемые в программном обеспечении BeadStudio ». Эта проблема, хотя и действительна для анализа метилирования GoldenGate, не имеет отношения к чипам HumanMethylation27k, где оба зонда в паре расширяются и флуоресцируют в одном канале.[9]
Смотрите также
- MethDB База данных метилирования ДНК
внешняя ссылка
- Станция эпигенетического метилирования
- Обзоры природы:Сбор метилирования ДНК в Обзоры природы
- Протоколы анализа ДНК / метилирования
- Illumina, Метилирование инфиниума
- OMICtools: учебный справочник по анализу данных массива метилирования ДНК.
Рекомендации
- ^ а б c d е ж грамм час Вайзенбергер, диджей. и другие. (2008) Всесторонний анализ метилирования ДНК на платформе анализа Illumina Infinium. [1]
- ^ https://support.illumina.com/content/dam/illumina-marketing/documents/products/product_information_sheets/product_info_hm450.pdf
- ^ а б Метилирование ДНК в развитии и заболеваниях человека. Гопалакришнан С., Ван Эмбург Б.О., Робертсон К.Д. Мутационные исследования. 1 декабря 2008 г .; 647 (1-2): 30-8. Epub 2008 20 августа. Обзор.
- ^ а б c d е ж Illumina: Анализ метилирования ДНК: http://www.illumina.com/downloads/DNAMethylationAnalysis_DataSheet.pdf[постоянная мертвая ссылка ]
- ^ Gunderson, KL. и другие. Масштабируемый анализ генотипирования SNP по всему геному с использованием технологии микрочипов. Природа Генетика 37, 549 - 554 (2005).
- ^ а б Стимерс, Ф.Дж. и другие. Полногеномное генотипирование с помощью анализа расширения одного основания. Природные методы, Vol. 3, № 1, 31 - 33 января (2006).
- ^ Руководство пользователя модуля метилирования BeadStudio. Версия 3. Illumina Systems & Software. 2006 г.
- ^ NCBI, проект Consensus CDS (CCDS)
- ^ а б Staaf, J. et al. Нормализация полногеномных данных SNP Illumina Infinium улучшает оценки числа копий и отношения аллельной интенсивности. BMC Bioinformatics 2008, 9:409. [2]
