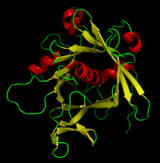
Ленточная диаграмма - Ribbon diagram
Ленточные диаграммы, также известный как Диаграммы Ричардсона, находятся 3D схематические изображения структура белка и являются одним из наиболее распространенных методов изображения белков, используемых сегодня. Лента показывает общий путь и организацию белкового остова в 3D и служит визуальной рамкой, на которой можно повесить детали полной атомной структуры, такие как шары для атомов кислорода, связанных с активным участком миоглобина в соседнем изображение. Ленточные диаграммы генерируются путем интерполяции гладкой кривой через полипептид позвоночник. α-спирали показаны в виде спиральных лент или толстых трубок, β-тяжи в виде стрелок и неповторяющихся витков или петель в виде линий или тонких трубок. Направление полипептидная цепь показан локально стрелками и может обозначаться в целом шкалой цвета по длине ленты.[1]
Ленточные диаграммы просты, но эффективны, они отражают визуальные основы молекулярной структуры (скручивание, складывание и разворачивание). Этот метод успешно изобразил общую организацию белковых структур, отражая их трехмерную природу и позволяя лучше понять эти сложные объекты как опытным структурным биологам, так и другим ученым, студентам,[2] и широкая публика.

История
Первые ленточные диаграммы, нарисованные от руки Джейн С. Ричардсон в 1980 г. (под влиянием более ранних отдельных иллюстраций)[3], были первыми систематическими схемами трехмерной структуры белка.[3][4] Они были созданы, чтобы проиллюстрировать классификацию белковых структур для статьи в Достижения в химии белков[5] (теперь доступны в аннотированной форме на сайте Анатакс ). Эти рисунки были начерчены пером на кальке поверх распечатки Cα след атомных координат и заштрихованный цветным карандашом или пастелью[6]; они сохранили позиции, сгладили магистральный путь и включили небольшие локальные сдвиги, чтобы устранить неоднозначность внешнего вида.[4] Помимо рисунка ленты триозоизомеразы справа, изображены другие нарисованные от руки примеры преальбумин, флаводоксин, и Cu, Zn супероксиддисмутаза.
В 1982 г. Артур М. Леск и сотрудники впервые включили автоматическое создание ленточных диаграмм с помощью вычислительной реализации, которая использует Банк данных белков файлы в качестве входных.[7] Это концептуально простой алгоритм поместиться кубический многочлен B-шлиц кривые к пептидным плоскостям. Большинство современных графических систем предоставляют либо B-сплайны, либо Шлицы Hermite как базовый примитив чертежа. Один тип реализации сплайна проходит через каждую направляющую точку Cα, создавая точную, но прерывистую кривую. И нарисованные от руки, и большинство компьютерных лент (например, показанные здесь) сглаживаются примерно по четырем последовательным направляющим точкам (обычно по средней точке пептида), чтобы получить более визуально приятное и понятное изображение. Чтобы задать правильный радиус винтовой спирали при сохранении гладких β-нитей, шлицы могут быть модифицированы смещениями, пропорциональными локальной кривизне, как впервые разработал Майк Карсон для его программы Ribbons.[8] и позже адаптированный другим программным обеспечением для молекулярной графики, таким как программа Mage с открытым исходным кодом для кинемаг графика[9] который создал изображение ленты вверху справа (другие примеры: Тример 1XK8 и ДНК-полимераза ).
С момента их создания и по настоящее время ленточные диаграммы были единственным наиболее распространенным представлением структуры белка и обычным выбором изображения обложки для журнала или учебника.
Текущие компьютерные программы
Одной из популярных программ, используемых для рисования ленточных диаграмм, является Молскрипт. Molscript использует Шлицы Hermite для создания координат витков, витков, нитей и спиралей. Кривая проходит через все свои контрольные точки (атомы Cα), ориентируясь по векторам направления. Программа построена на основе традиционной молекулярной графики. Артур М. Леск, Карл Хардман и Джон Пристл.[10] Jmol это программа просмотра на основе Java с открытым исходным кодом для просмотра молекулярных структур в сети; он включает упрощенную «мультяшную» версию лент. Другие графические программы, такие как DeepView (пример: уреаза ) и МолМол (пример: SH2 домен ) также создают изображения лент. Король[11] является преемником Mage на основе Java (примеры: α-гемолизин вид сверху и вид сбоку ).
UCSF Химера это мощная программа молекулярного моделирования, которая также включает визуализации такие как ленты, примечательные особенно способностью сочетать их с контурными формами из крио-электронная микроскопия данные.[12] PyMOL, к Уоррен ДеЛано,[13] популярная и гибкая программа молекулярной графики (на основе Python ), который работает в интерактивном режиме, а также создает 2D-изображения презентационного качества для ленточных диаграмм и многих других представлений.
Функции
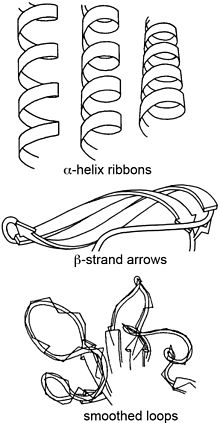
| Вторичная структура[4][5] | |
|---|---|
| α-спирали | Цилиндрические спиральные ленты, плоскость которых примерно повторяет плоскость пептидов. |
| β-нити | Стрелки толщиной примерно в четверть их ширины показывают направление и изгиб нити от аминогруппы к карбоксильному концу. β-листы считаются едиными, потому что соседние нити скручиваются в унисон. |
| Петли и прочее | |
| Неповторяющиеся петли | Круглые веревки, которые толще на переднем плане и тоньше к спине, следуя сглаженной траектории следа Cα. |
| Переходы между петлями и спиралями | Круглая веревка, которая постепенно превращается в тонкую спиральную ленту. |
| Другие свойства | |
| Полипептидное направление, NH2 и COOH termini | Маленькие стрелки на одном или обоих концах или буквах. Для β-нитей достаточно направления стрелки. Сегодня направление полипептидной цепи часто указывается с помощью цветовой шкалы. |
| Дисульфидные связи | Переплетенный символ СС или зигзаг, похожий на стилизованный удар молнии. |
| Протезные группы или ингибиторы | Фигурки из палочек или мяч и клюшка. |
| Металлы | Сферы. |
| Затенение и цвет | Затенение или цвет добавляют диаграмме объемности. Как правило, элементы спереди наиболее контрастны, а элементы сзади - минимальны. |
Смотрите также
Рекомендации
- ^ Смит, Томас Дж. (27 октября 2005 г.). «Отображение и анализ атомных структур на Macintosh». Центр науки о растениях Данфорта. Архивировано из оригинал 28 марта 2002 г.
- ^ Richardson, D.C .; Ричардсон, Дж. С. (январь 2002 г.). «Обучение молекулярной 3-D грамотности». Биохимия и молекулярная биология образование. 30 (1): 21–26. Дои:10.1002 / bmb.2002.494030010005.
- ^ а б Ричардсон, Джейн С. (2000), «Ранние ленточные рисунки белков», Структурная биология природы, 7 (8): 624–625, Дои:10.1038/77912, PMID 10932243.
- ^ а б c Ричардсон, Джейн С. (1985), «Схематические изображения белковых структур», Методы в энзимологии, Методы в энзимологии, 115: 359–380, Дои:10.1016/0076-6879(85)15026-3, ISBN 978-0-12-182015-2, PMID 3853075.
- ^ а б Ричардсон, Джейн С. (1981), «Анатомия и таксономия белковых структур», Достижения в химии белков, Достижения в химии белков, 34: 167–339, Дои:10.1016 / S0065-3233 (08) 60520-3, ISBN 978-0-12-034234-1, PMID 7020376.
- ^ «Мать ленточных диаграмм» Science отмечает 50-летие компании Duke ». Истории герцога. 2018-10-19. Получено 2020-06-09.
- ^ Леск, Артур М .; Хардман, Карл Д. (1982), "Компьютерные схематические диаграммы белковых структур", Наука, 216 (4545): 539–540, Bibcode:1982Научный ... 216..539L, Дои:10.1126 / science.7071602, PMID 7071602.
- ^ Carson, M .; Багг, К. Э. (1986), "Алгоритм для ленточных моделей белков", Журнал молекулярной графики, 4 (2): 121–122, Дои:10.1016/0263-7855(86)80010-8.
- ^ Richardson, D.C .; Ричардсон, Дж. С. (январь 1992 г.), «Кинемаг: инструмент для научного общения», Белковая наука, 1 (1): 3–9, Дои:10.1002 / pro.5560010102, ЧВК 2142077, PMID 1304880
- ^ MolScript v2.1: О программе
- ^ Chen, V. B .; Дэвис, И. В .; Ричардсон, Д. К. (2009), «KING (Kinemage, Next Generation): универсальная интерактивная программа молекулярной и научной визуализации», Белковая наука, 18 (11): 2403–2409, Дои:10.1002 / pro.250, ЧВК 2788294, PMID 19768809
- ^ Годдард, Томас Д .; Хуанг, Конрад С.; Феррин, Томас Э. (2005), «Расширения программного обеспечения для UCSF Chimera для интерактивной визуализации больших молекулярных сборок», Структура, 13 (3): 473–482, Дои:10.1016 / j.str.2005.01.006, PMID 15766548.
- ^ Brunger, Axel T .; Уэллс, Джеймс А. (2009), «Уоррен Л. ДеЛано, 21 июня 1972 г. - 3 ноября 2009 г.», Структурная и молекулярная биология природы, 16 (12): 1202–1203, Дои:10.1038 / nsmb1209-1202, PMID 19956203.