H4K91ac - H4K91ac
H4K91ac является эпигенетический модификация белка упаковки ДНК гистон H4. Это знак, обозначающий ацетилирование на 91-м лизин остаток белка гистона H4. Этому знаку не приписываются никакие известные заболевания, но он может быть связан с меланома.
Номенклатура
H4K91ac указывает на ацетилирование лизин 91 на субъединице белка гистона H4:[1]
| Сокр. | Смысл |
| H4 | Семейство гистонов H4 |
| K | стандартное сокращение для лизина |
| 91 | положение аминокислотный остаток (считая от N-конца) |
| ac | ацетильная группа |
Модификации гистонов
Геномная ДНК эукариотический клетки окружены особыми белковыми молекулами, известными как гистоны. Комплексы, образованные петлей ДНК, известны как хроматин. Основной структурной единицей хроматина является нуклеосома: он состоит из основного октамера гистонов (H2A, H2B, H3 и H4), а также линкерного гистона и около 180 пар оснований ДНК. Эти гистоны ядра богаты остатками лизина и аргинина. Карбоксильный (С) конец этих гистонов способствует взаимодействию гистонов с гистонами, а также взаимодействиям гистонов с ДНК. Амино (N) -концевые заряженные хвосты являются местом расположения посттрансляционные модификации, например, тот, что видели в H3K36me3.[2][3]
Гистон H4
Модификации H4 не так хорошо известны, как H3, и H4 имеют меньше вариаций, что может объяснить их важную функцию.[4]
H4K91ac
По состоянию на 15 декабря 2019 года этому знаку не приписываются никакие заболевания.[5] несмотря на то что Домен гомологии плэкстрина Нацеленный бромодомен (PHIP) специфически связывает H4K91ac, который может вовлекать PHIP в прогрессирование меланомы.[6] Он находится в сайт начала транскрипции (TSS) активных и сбалансированных генов.[5]
Гистонацетилтрансфераза KAT2A это конкретный читатель.[5]
Ацетилирование и деацетилирование лизина
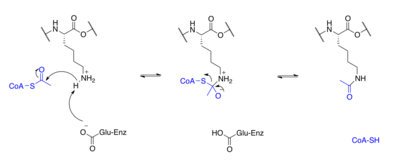
Белки обычно ацетилируются на лизин остатков, и эта реакция зависит от ацетил-кофермент А в качестве донора ацетильной группы. В ацетилирование и деацетилирование гистонов, гистоновые белки ацетилируются и деацетилируются по остаткам лизина в N-концевом хвосте как часть генная регуляция. Обычно эти реакции катализируются ферменты с гистонацетилтрансфераза (HAT) или гистоновая деацетилаза (HDAC), хотя HAT и HDAC также могут изменять статус ацетилирования негистоновых белков.[7]
Регуляция факторов транскрипции, эффекторных белков, молекулярные шапероны, а белки цитоскелета путем ацетилирования и деацетилирования являются важным посттрансляционным регуляторным механизмом.[8] Эти регуляторные механизмы аналогичны фосфорилированию и дефосфорилированию под действием киназы и фосфатазы. Не только состояние ацетилирования белка может изменять его активность, но и эта посттрансляционная модификация может также пересекаться с фосфорилирование, метилирование, убиквитинирование, сумоилирование и другие для динамического контроля клеточной сигнализации.[9][10][11]
Эпигенетические последствия
Посттрансляционная модификация гистоновых хвостов с помощью комплексов модификации гистонов или комплексов ремоделирования хроматина интерпретируется клеткой и приводит к сложному комбинаторному выходу транскрипции. Считается, что гистоновый код диктует экспрессию генов за счет сложного взаимодействия между гистонами в определенной области.[12] Текущее понимание и интерпретация гистонов происходит из двух крупномасштабных проектов: КОДИРОВАТЬ и эпигеномная дорожная карта.[13] Целью эпигеномного исследования было изучить эпигенетические изменения по всему геному. Это привело к состояниям хроматина, которые определяют области генома путем группирования взаимодействий различных белков и / или модификаций гистонов вместе. Состояния хроматина исследовали в клетках дрозофилы, глядя на место связывания белков в геноме. Использование ChIP-секвенирование выявили участки в геноме, характеризующиеся различной полосатостью.[14] Различные стадии развития были профилированы и у Drosophila, акцент был сделан на релевантности модификации гистонов.[15] Анализ полученных данных привел к определению состояний хроматина на основе модификаций гистонов.[16]
Геном человека был аннотирован состояниями хроматина. Эти аннотированные состояния могут использоваться как новые способы аннотирования генома независимо от базовой последовательности генома. Эта независимость от последовательности ДНК обеспечивает эпигенетический характер модификаций гистонов. Состояния хроматина также полезны для идентификации регуляторных элементов, не имеющих определенной последовательности, таких как усилители. Этот дополнительный уровень аннотации позволяет глубже понять регуляцию клеточно-специфических генов.[17]
Методы
Ацетилирование гистоновой метки можно обнаружить разными способами:
1. Иммунопреципитация хроматина Последовательность действий (ChIP-секвенирование ) измеряет степень обогащения ДНК после связывания с целевым белком и иммунопреципитированный. Это приводит к хорошей оптимизации и используется in vivo для выявления связывания ДНК с белками, происходящего в клетках. ChIP-Seq можно использовать для идентификации и количественного определения различных фрагментов ДНК для различных модификаций гистонов вдоль геномной области.[18]
2. Секвенирование микрококковой нуклеазы (MNase-seq ) используется для исследования областей, которые связаны с хорошо расположенными нуклеосомами. Для определения положения нуклеосом используется фермент микрококковой нуклеазы. Видно, что хорошо расположенные нуклеосомы имеют обогащенные последовательности.[19]
3. Анализ для секвенирования доступного транспозазе хроматина (ATAC-seq ) используется для поиска участков, свободных от нуклеосом (открытый хроматин). Использует гиперактивный Транспозон Tn5 чтобы выделить локализацию нуклеосом.[20][21][22]
Смотрите также
Рекомендации
- ^ Хуанг, Суминг; Литт, Майкл Д .; Энн Блейки, К. (30 ноября 2015 г.). Экспрессия и регуляция эпигенетических генов. С. 21–38. ISBN 9780127999586.
- ^ Ruthenburg AJ, Li H, Patel DJ, Allis CD (декабрь 2007 г.). «Многовалентное взаимодействие модификаций хроматина за счет связанных связывающих модулей». Обзоры природы. Молекулярная клеточная биология. 8 (12): 983–94. Дои:10.1038 / nrm2298. ЧВК 4690530. PMID 18037899.
- ^ Кузаридес Т. (февраль 2007 г.). «Модификации хроматина и их функции». Клетка. 128 (4): 693–705. Дои:10.1016 / j.cell.2007.02.005. PMID 17320507.
- ^ «Гистоновые белки и модификации». Epigenie. Получено 15 декабря 2019.
- ^ а б c "Пост-трансляционная модификация H4K91ac - HIstome". Получено 14 декабря 2019.
- ^ Де Семир, Дэвид; Безрооково, Владимир; Носрати, Мехди; Dar, Altaf A .; Ву, Клейтон; Шен, Юлия; Рикен, Кристофер; Венкатасубраманиан, Минакши; Миллер, Джеймс Р .; Деспре, Пьер-Ив; Макаллистер, Шон; Сорочану, Лилиана; Дебс, Роберт Дж .; Саломонис, Натан; Шадендорф, Дирк; Кливер, Джеймс Э .; Кашани-Сабет, Мохаммед (2018). «PHIP как терапевтическая мишень для отрицательных по водителю подтипов меланомы, рака груди и легких». Труды Национальной академии наук. 115 (25): E5766 – E5775. Дои:10.1073 / pnas.1804779115. ЧВК 6016792. PMID 29866840.
- ^ Садоул К., Бойо С., Пабион М., Хочбин С. (2008). «Регулирование белкового обмена ацетилтрансферазами и деацетилазами». Биохимия. 90 (2): 306–12. Дои:10.1016 / j.biochi.2007.06.009. PMID 17681659.
- ^ Glozak MA, Sengupta N, Zhang X, Seto E (2005). «Ацетилирование и деацетилирование негистоновых белков». Ген. 363: 15–23. Дои:10.1016 / j.gene.2005.09.010. PMID 16289629.
- ^ Ян XJ, Сето Э (2008). «Ацетилирование лизина: кодифицированное перекрестное взаимодействие с другими посттрансляционными модификациями». Мол. Клетка. 31 (4): 449–61. Дои:10.1016 / j.molcel.2008.07.002. ЧВК 2551738. PMID 18722172.
- ^ Эдде Б., Денуле П., де Нешо Б., Кулакофф А., Бервальд-Неттер Ю., Грос Ф (1989). «Посттрансляционные модификации тубулина в культивируемых нейронах мозга мышей и астроглии». Биол. Клетка. 65 (2): 109–117. Дои:10.1016 / 0248-4900 (89) 90018-х. PMID 2736326.
- ^ Марута Х, Грир К., Розенбаум Дж. Л. (1986). «Ацетилирование альфа-тубулина и его связь со сборкой и разборкой микротрубочек». J. Cell Biol. 103 (2): 571–579. Дои:10.1083 / jcb.103.2.571. ЧВК 2113826. PMID 3733880.
- ^ Jenuwein T, Allis CD (август 2001 г.). «Перевод гистонового кода». Наука. 293 (5532): 1074–80. Дои:10.1126 / science.1063127. PMID 11498575.
- ^ Бирни Э, Стаматояннопулос Ж.А., Датта А, Гиго Р., Джингерас Т.Р., Маргулиес Э.Х. и др. (Консорциум проекта ENCODE) (июнь 2007 г.). «Идентификация и анализ функциональных элементов в 1% генома человека в рамках пилотного проекта ENCODE». Природа. 447 (7146): 799–816. Bibcode:2007Натура.447..799Б. Дои:10.1038 / природа05874. ЧВК 2212820. PMID 17571346.
- ^ Филион Дж. Дж., Ван Беммель Дж. Г., Брауншвейг Ю., Талхаут В., Кинд Дж., Уорд Л. Д., Бругман В., де Кастро И. Дж., Керховен Р. М., Бассемейкер Г. Дж., Ван Стинсель Б. «Систематическое картирование расположения белков выявляет пять основных типов хроматина в клетках дрозофилы». Клетка. 143 (2): 212–24. Дои:10.1016 / j.cell.2010.09.009. ЧВК 3119929. PMID 20888037.
- ^ Рой С., Эрнст Дж., Харченко П.В., Херадпур П., Негре Н., Итон М.Л. и др. (Консорциум modENCODE) (декабрь 2010 г.). «Идентификация функциональных элементов и регуляторных цепей с помощью Drosophila modENCODE». Наука. 330 (6012): 1787–97. Bibcode:2010Научный ... 330.1787R. Дои:10.1126 / science.1198374. ЧВК 3192495. PMID 21177974.
- ^ Харченко П.В., Алексеенко А.А., Шварц Ю.Б., Минода А., Риддл Н.С., Эрнст Дж. И др. (Март 2011 г.). «Комплексный анализ хроматина у Drosophila melanogaster». Природа. 471 (7339): 480–5. Bibcode:2011Натура.471..480K. Дои:10.1038 / природа09725. ЧВК 3109908. PMID 21179089.
- ^ Kundaje A, Meuleman W., Ernst J, Bilenky M, Yen A, Heravi-Moussavi A, Kheradpour P, Zhang Z, et al. (Консорциум Roadmap Epigenomics) (февраль 2015 г.). «Интегративный анализ 111 эталонных эпигеномов человека». Природа. 518 (7539): 317–30. Bibcode:2015Натура.518..317.. Дои:10.1038 / природа14248. ЧВК 4530010. PMID 25693563.
- ^ «IP-секвенирование всего генома хроматина (ChIP-Seq)» (PDF). Иллюмина. Получено 23 октября 2019.
- ^ «MAINE-Seq / Mnase-Seq». иллюмина. Получено 23 октября 2019.
- ^ Буэнростро, Джейсон Д .; Ву, Пекин; Chang, Howard Y .; Гринлиф, Уильям Дж. (2015). «ATAC-seq: метод определения доступности хроматина для всего генома». Текущие протоколы в молекулярной биологии. 109: 21.29.1–21.29.9. Дои:10.1002 / 0471142727.mb2129s109. ISBN 9780471142720. ЧВК 4374986. PMID 25559105.
- ^ Schep, Alicia N .; Буэнростро, Джейсон Д .; Денни, Сара К .; Шварц, Катя; Шерлок, Гэвин; Гринлиф, Уильям Дж. (2015). «Структурированные отпечатки пальцев нуклеосом позволяют с высоким разрешением картировать архитектуру хроматина в регуляторных областях». Геномные исследования. 25 (11): 1757–1770. Дои:10.1101 / гр.192294.115. ISSN 1088-9051. ЧВК 4617971. PMID 26314830.
- ^ Песня, Л .; Кроуфорд, Г. Э. (2010). «DNase-seq: метод высокого разрешения для картирования активных регуляторных элементов генов в геноме из клеток млекопитающих». Протоколы Колд-Спринг-Харбор. 2010 (2): pdb.prot5384. Дои:10.1101 / pdb.prot5384. ISSN 1559-6095. ЧВК 3627383. PMID 20150147.
