Иммунопреципитация метилированной ДНК - Methylated DNA immunoprecipitation
Иммунопреципитация метилированной ДНК (MeDIP или mDIP) - крупномасштабный (хромосома - или же геном широко) метод очистки в молекулярной биологии, который используется для обогащения метилированные последовательности ДНК. Он состоит из выделения метилированных фрагментов ДНК через антитело поднял против 5-метилцитозин (5 мкС). Этот метод был впервые описан Вебером М. и другие.[1] в 2005 г. и помог проложить путь к жизнеспособным метилом -уровневая оценка, поскольку очищенная фракция метилированной ДНК может быть использована для высокопроизводительных методов обнаружения ДНК, таких как высокое разрешение ДНК-микрочипы (MeDIP-чип ) или секвенирование следующего поколения (MeDIP-seq). Тем не менее, понимание метилома остается рудиментарным; его изучение осложняется тем, что, как и другие эпигенетический Свойства, шаблоны различаются от типа ячейки к типу ячейки.
Фон
Метилирование ДНК, ссылаясь на обратимое метилирование 5-го положения цитозина посредством метилтрансферазы, является основным эпигенетический модификация в многоклеточных организмах.[2] У млекопитающих эта модификация в основном происходит в CpG сайты, которые, в свою очередь, имеют тенденцию группироваться в регионах, называемых Острова CpG.[3] Существует небольшая часть островков CpG, которые могут перекрываться или находиться в непосредственной близости от промоторных областей сайтов начала транскрипции. Изменение также может произойти на других сайтах,[4] но метилирование в любом из этих сайтов может подавлять экспрессию гена, вмешиваясь в связывание факторы транскрипции или изменение хроматин структура до репрессивного состояния.[5]
Исследования патологий в значительной степени способствовали пониманию роли метилирования ДНК. В настоящее время основной исследовательский интерес заключается в изучении таких заболеваний, как: рак для идентификации участков ДНК, подвергшихся обширным изменениям метилирования. Гены, содержащиеся в этих областях, представляют функциональный интерес, поскольку они могут предложить механистическое объяснение основных генетических причин заболевания. Например, паттерн патологического метилирования раковых клеток[6][7][8] первоначально было показано, что это механизм, посредством которого подавитель опухолей -подобные гены заглушаются,[9] хотя позже было замечено, что поражается гораздо более широкий спектр типов генов.[10][11][12]
Прочие технологии
Существует два подхода к анализу метилирования: технологии типирования и профилирования. Технологии ввода нацелены на небольшое количество локусов во многих выборках и предполагают использование таких методов, как ПЦР, рестрикционные ферменты, и масс-спектрометрии. Технологии профилирования, такие как MeDIP, ориентированы на геном - или же метилом - оценка уровня метилирования по всему миру; Это включает в себя геномное сканирование рестрикционных ориентиров (RLGS),[13] и преобразование бисульфита -основанные методы, основанные на обработке ДНК бисульфит преобразовать неметилированный цитозин остатки урацил.[14][15][16][17]
Ограничения других технологий
Другие методы картирования и профилирования метилома были эффективными, но не без ограничений, которые могут влиять на разрешение, уровень пропускной способности или экспериментальные вариации. Например, RLGS ограничен количеством сайтов рестрикции в геноме, которые могут быть мишенями для рестрикционного фермента; обычно можно оценить максимум ~ 4100 ориентиров.[18] Бисульфитное секвенирование Методы, основанные на методах, несмотря на возможное разрешение одного нуклеотида, имеют недостаток: превращение неметилированного цитозина в урацил может быть нестабильным.[19] Кроме того, когда превращение бисульфита сочетается с ДНК-микрочипы Для обнаружения сайтов, превращенных в бисульфит, проблемой является снижение сложности последовательности ДНК. Микроматрицы, способные всесторонне профилировать весь геном, становится трудным в разработке, поскольку доступно меньше уникальных зондов.[20]
Методы
В следующих разделах описывается метод MeDIP в сочетании с гибридизацией массива с высоким разрешением или секвенированием с высокой пропускной способностью. Каждый метод обнаружения ДНК также кратко описывает пост-лабораторную обработку и анализ. В зависимости от технологии, используемой для идентификации метилированных последовательностей, требуется различная постобработка исходных данных. Это аналогично данным, полученным с помощью ЧИП-чип и ChIP-seq.
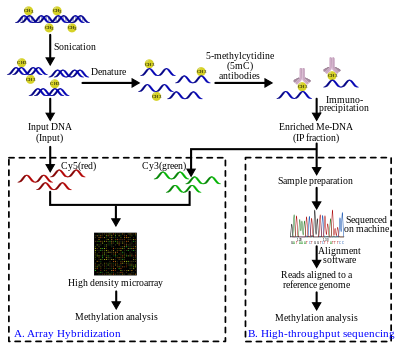
Иммунопреципитация метилированной ДНК (MeDIP)
Геномная ДНК извлекается (Извлечение ДНК ) из клеток и очищены. Затем очищенную ДНК подвергают обработке ультразвуком, чтобы разделить ее на случайные фрагменты. Этот процесс обработки ультразвуком быстрый, простой и позволяет избежать рестрикционный фермент предубеждения. Полученные фрагменты имеют длину от 300 до 1000 пар оснований (п.о.), хотя обычно они составляют от 400 до 600 п.н.[21] Короткая длина этих фрагментов важна для получения адекватного разрешения, повышения эффективности последующего этапа иммунопреципитации и уменьшения влияния длины фрагмента или смещения. Кроме того, размер фрагмента влияет на связывание 5-метилцитидинового (5mC) антитела, поскольку для эффективного связывания антителу требуется больше, чем один 5mC.[22] Для дальнейшего повышения аффинности связывания антител фрагменты ДНК денатурируют с образованием одноцепочечной ДНК. После денатурации ДНК инкубируют с моноклональный Антитела 5mC. Классический иммунопреципитация Затем применяется техника: магнитные шарики конъюгированы с антимышинымIgG используются для связывания антител против 5mC, а несвязанная ДНК удаляется в надосадочной жидкости. Чтобы очистить ДНК, протеиназа К добавляется для переваривания антител и высвобождения ДНК, которую можно собрать и подготовить для обнаружения ДНК.
Подробнее об экспериментальных этапах см.[1][19][23][24]
MeDIP и гибридизация на основе массивов (MeDIP-чип)
Часть входящей ДНК, полученной после этапа обработки ультразвуком, помечена цианин -5 (Cy5; красный) дезоксицитозинтрифосфат, в то время как метилированная ДНК, обогащенная после стадии иммунопреципитации, помечена цианин -3 (Cy3; зеленый). Меченые образцы ДНК совместно гибридизуют на 2-канальном геномном микрочипе высокой плотности для проверки наличия и относительных количеств. Целью этого сравнения является идентификация последовательностей, которые показывают значительные различия в уровнях гибридизации, тем самым подтверждая, что интересующая последовательность обогащена. Идентификация последовательностей MeDIP на основе массива ограничивается конструкцией массива. В результате разрешение ограничено датчиками в конструкции массива. При обработке сигналов требуются дополнительные стандартные шаги для исправления таких проблем гибридизации, как шум, как в случае с большинством технологий массивов.
Видеть [23][24][25] Больше подробностей.
MeDIP и высокопроизводительное секвенирование (MeDIP-seq)
Подход MeDIP-seq, то есть соединение MeDIP с технологиями короткого чтения следующего поколения, такими как 454 пиросеквенирование или Иллюмина (Солекса), была впервые описана Дауном и другие. в 2008.[20] Высокопроизводительное секвенирование метилированных фрагментов ДНК дает большое количество коротких прочтений (36-50 п.н.[26] или 400 п.н.,[27] в зависимости от технологии). Короткие чтения выравниваются по эталонному геному с помощью программного обеспечения для выравнивания, такого как Mapping and Assembly with Quality (Maq ), который использует Байесовский подход, наряду с базовыми качествами и качествами отображения, для моделирования вероятностей ошибок для выравниваний.[28] Затем считывания могут быть расширены для представления фрагментов от ~ 400 до 700 п.н. из стадии обработки ультразвуком. Охват этих расширенных чтений можно использовать для оценки уровня метилирования региона. А браузер генома такие как Ансамбль также может использоваться для визуализации данных.
Подтверждение подхода к оценке качества и точности данных может быть выполнено с помощью количественная ПЦР. Это делается путем сравнения последовательности из образца MeDIP с неметилированной контрольной последовательностью. Затем образцы запускаются на гель и сравниваются интенсивности полос.[19] Относительная интенсивность служит ориентиром для поиска обогащения. Результаты также можно сравнить с результатами чипа MeDIP, чтобы определить необходимое покрытие.
Последующий биоинформатический анализ

Оценки уровня метилирования ДНК могут быть затруднены из-за различной плотности метилированных сайтов CpG в геноме при наблюдении за данными, генерируемыми MeDIP. Это может быть проблематичным для анализа областей с низким содержанием CpG (с более низкой плотностью). Одной из причин этой проблемы с плотностью является ее влияние на эффективность иммунопреципитации. В своем исследовании Даун и другие.[20] разработали инструмент для оценки абсолютных уровней метилирования на основе данных, полученных с помощью MeDIP, путем моделирования плотности метилированных сайтов CpG. Этот инструмент называется Байесовский инструмент для анализа метилирования (Бэтмен). В исследовании сообщается о покрытии ~ 90% всех сайтов CpG в промоторах, ген-кодирующих областях, островках и регуляторных элементах, где можно оценить уровни метилирования; это почти в 20 раз лучшее покрытие, чем любые предыдущие методы.
Исследования с использованием MeDIP-seq или MeDIP-chip представляют собой общегеномные подходы, которые имеют общую цель - получение функционального картирования метилома. После идентификации участков метилирования ДНК можно применить ряд биоинформатических анализов, чтобы ответить на определенные биологические вопросы. Один очевидный шаг - исследовать гены, содержащиеся в этих регионах, и исследовать функциональное значение их репрессии. Например, подавление генов-супрессоров опухолей при раке может быть связано с метилированием ДНК.[29] Выявив мутационные события, ведущие к гиперметилированию и последующей репрессии известных генов-супрессоров опухолей, можно более конкретно охарактеризовать факторы, способствующие возникновению причины заболевания. Альтернативно, можно идентифицировать гены, которые, как известно, обычно метилированы, но в результате некоторого события мутации больше не заглушаются.
Кроме того, можно попытаться исследовать и определить, был ли затронут какой-либо эпигенетический регулятор, такой как ДНК-метилтрансфераза (DNMT);[21] в этих случаях обогащение может быть более ограниченным.
Было показано, что анализ генов (например, с использованием таких инструментов, как DAVID и GoSeq) сильно смещен при применении к данным метилирования с высокой пропускной способностью (например, MeDIP-seq и MeDIP-ChIP); Было высказано предположение, что это можно исправить, используя перестановки меток образца или используя статистическую модель для контроля различий в количестве зондов CpG / сайтов CpG, нацеленных на каждый ген.[30]
Ограничения MeDIP
Ограничения, которые следует учитывать при использовании MeDIP, являются типичными экспериментальными факторами. Это включает качество и перекрестную реактивность антител 5mC, используемых в процедуре. Кроме того, методы обнаружения ДНК (т.е. матричная гибридизация и высокопроизводительное секвенирование) обычно имеют хорошо установленные ограничения. В частности, для процедур на основе массивов, как упоминалось выше, анализируемые последовательности ограничиваются конкретным используемым дизайном массива.
Применяются наиболее типичные ограничения для высокопроизводительного секвенирования следующего поколения. Проблема точности выравнивания повторяющихся областей в геноме приведет к менее точному анализу метилирования в этих областях. Кроме того, как упоминалось выше, короткие чтения (например, 36-50 б.п. из Анализатор генома Illumina ) представляют собой часть срезанного фрагмента при выравнивании по геному; следовательно, точный сайт метилирования может попадать в любое место в пределах окна, которое зависит от размера фрагмента.[19] В этом отношении бисульфитное секвенирование имеет гораздо более высокое разрешение (вплоть до одного сайта CpG; уровень одного нуклеотида). Однако этот уровень разрешения может не требоваться для большинства приложений, так как было показано, что статус метилирования сайтов CpG в пределах <1000 п.н. значительно коррелирует.[20]
Приложения MeDIP
- Вебер и другие. 2005[1] определили, что неактивная Х-хромосома у женщин гиперметилирована на уровне всей хромосомы с использованием MeDIP в сочетании с микрочипом.
- Кешет и другие. 2006[31] провели исследование клеток рака толстой кишки и простаты с помощью MeDIP-чипа. Результатом является полногеномный анализ генов, лежащих в гиперметилированных областях, а также делается вывод о том, что существует поучительный механизм метилирования de novo в раковых клетках.
- Чжан и другие. 2006[24] с помощью MeDIP-чипа получили метиломное картирование с высоким разрешением у Arabidopsis.
- Новак и другие. 2006[32] использовали подход MeDIP-chip для исследования рака груди человека на молчание, связанное с метилированием, и наблюдали инактивацию кластера генов HOXA
Смотрите также
- Эпигенетика
- Иммунопреципитация
- Метилом
- Геномное сканирование рестрикционных ориентиров
- Бисульфитное секвенирование
Рекомендации
- ^ а б c Вебер М., Дэвис Дж. Дж., Виттиг Д. и др. (Август 2005 г.). «Хромосомный и промотор-специфический анализ выявляет участки дифференциального метилирования ДНК в нормальных и трансформированных клетках человека». Nat. Genet. 37 (8): 853–62. Дои:10,1038 / ng1598. PMID 16007088.
- ^ Птица А (январь 2002 г.). «Паттерны метилирования ДНК и эпигенетическая память». Genes Dev. 16 (1): 6–21. Дои:10.1101 / gad.947102. PMID 11782440.
- ^ Гардинер-Гарден М., Фроммер М. (июль 1987 г.). «Острова CpG в геномах позвоночных». J. Mol. Биол. 196 (2): 261–82. Дои:10.1016/0022-2836(87)90689-9. PMID 3656447.
- ^ Кларк С.Дж., Харрисон Дж., Фроммер М. (май 1995 г.). «Метилирование CpNpG в клетках млекопитающих». Nat. Genet. 10 (1): 20–7. Дои:10.1038 / ng0595-20. PMID 7647784.
- ^ Jaenisch R, Bird A (март 2003 г.). «Эпигенетическая регуляция экспрессии генов: как геном объединяет внутренние и внешние сигналы». Nat. Genet. 33 (Прил.): 245–54. Дои:10.1038 / ng1089. PMID 12610534.
- ^ Робертсон К.Д., Вольф А.П. (октябрь 2000 г.). «Метилирование ДНК в здоровье и болезни». Nat. Преподобный Жене. 1 (1): 11–9. Дои:10.1038/35049533. PMID 11262868.
- ^ Байлин С.Б., Герман Дж. Г. (2000). «Гиперметилирование ДНК в онкогенезе: эпигенетика присоединяется к генетике». Тенденции Genet. 16 (4): 268–274. Дои:10.1016 / S0168-9525 (99) 01971-X. PMID 10729832.
- ^ Джонс PA, Laird PW (февраль 1999 г.). «Эпигенетика рака достигает совершеннолетия». Nat. Genet. 21 (2): 163–7. Дои:10.1038/5947. PMID 9988266.
- ^ Джонс П.А., Бейлин С.Б. (июнь 2002 г.). «Фундаментальная роль эпигенетических событий в раке». Nat. Преподобный Жене. 3 (6): 415–28. Дои:10.1038 / nrg816. PMID 12042769.
- ^ Костелло Дж. Ф., Фрювальд М. С., Смираглия Д. Д. и др. (Февраль 2000 г.). «Аберрантное метилирование CpG-островков имеет неслучайные и специфичные для опухолевого типа паттерны». Nat. Genet. 24 (2): 132–8. Дои:10.1038/72785. PMID 10655057.
- ^ Зардо Г., Тиирикайнен М.И., Хонг С. и др. (Ноябрь 2002 г.). «Комплексные геномные и эпигеномные анализы позволяют точно определить инактивацию двуаллельных генов в опухолях». Nat. Genet. 32 (3): 453–8. Дои:10,1038 / ng1007. PMID 12355068.
- ^ Yu L, Liu C, Vandeusen J, et al. (Март 2005 г.). «Глобальная оценка метилирования промотора на мышиной модели рака определяет ID4 как предполагаемый ген-супрессор опухоли при лейкемии человека». Nat. Genet. 37 (3): 265–74. Дои:10,1038 / ng1521. PMID 15723065.
- ^ Хатада И., Хаяшизаки Ю., Хироцунэ С., Комацубара Х., Мукаи Т. (ноябрь 1991 г.). «Метод геномного сканирования высших организмов с использованием рестрикционных сайтов в качестве ориентиров». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 88 (21): 9523–7. Bibcode:1991PNAS ... 88.9523H. Дои:10.1073 / pnas.88.21.9523. ЧВК 52750. PMID 1946366.
- ^ Ракян В.К., Хильдманн Т., Новик К.Л. и др. (Декабрь 2004 г.). «Профилирование ДНК-метилирования основного комплекса гистосовместимости человека: пилотное исследование для проекта эпигенома человека». ПЛОС Биол. 2 (12): e405. Дои:10.1371 / journal.pbio.0020405. ЧВК 529316. PMID 15550986.

- ^ Гитан Р.С., Ши Х., Чен С.М., Ян П.С., Хуан Т.Х. (январь 2002 г.). "Микроматрица специфических для метилирования олигонуклеотидов: новый потенциал для высокопроизводительного анализа метилирования". Genome Res. 12 (1): 158–64. Дои:10.1101 / гр.202801. ЧВК 155260. PMID 11779841.
- ^ Мейснер А., Гнирке А., Белл Г. В., Рамсахой Б., Ландер Э. С., Яениш Р. (2005). «Сокращенное представление бисульфитного секвенирования для сравнительного анализа метилирования ДНК с высоким разрешением». Нуклеиновые кислоты Res. 33 (18): 5868–77. Дои:10.1093 / нар / gki901. ЧВК 1258174. PMID 16224102.
- ^ Адорьян П., Дистлер Дж., Липшер Э. и др. (Март 2002 г.). «Прогнозирование и обнаружение класса опухоли с помощью анализа метилирования ДНК на основе микрочипов». Нуклеиновые кислоты Res. 30 (5): 21e – 21. Дои:10.1093 / nar / 30.5.e21. ЧВК 101257. PMID 11861926.
- ^ Dai Z, Weichenhan D, Wu YZ и др. (Октябрь 2002 г.). "Библиотека границ AscI для изучения генетических и эпигенетических изменений на островах CpG". Genome Res. 12 (10): 1591–8. Дои:10.1101 / гр.197402. ЧВК 187524. PMID 12368252.
- ^ а б c d Помранинг К.Р., Смит К.М., Фрайтаг М. (март 2009 г.). «Полногеномный высокопроизводительный анализ метилирования ДНК у эукариот». Методы. 47 (3): 142–50. Дои:10.1016 / j.ymeth.2008.09.022. PMID 18950712.
- ^ а б c d Даун Т.А., Ракян В.К., Тернер Д.Д. и др. (Июль 2008 г.). «Байесовская стратегия деконволюции для анализа метиломов ДНК на основе иммунопреципитации». Nat. Биотехнология. 26 (7): 779–85. Дои:10.1038 / nbt1414. ЧВК 2644410. PMID 18612301.
- ^ а б Хасинто Ф.В., Баллестар Э, Эстеллер М (январь 2008 г.). «Иммунопреципитация метил-ДНК (MeDIP): поиск метилома ДНК». Биотехнологии. 44 (1): 35–43. Дои:10.2144/000112708. PMID 18254377.
- ^ Михан Р. Р., Льюис Дж. Д., Берд А. П. (октябрь 1992 г.). «Характеристика MeCP2, ДНК-связывающего белка позвоночных со сродством к метилированной ДНК». Нуклеиновые кислоты Res. 20 (19): 5085–92. Дои:10.1093 / nar / 20.19.5085. ЧВК 334288. PMID 1408825.
- ^ а б Wilson IM, et al. (2005). «Эпигеномика: картирование метилома». Клеточный цикл. 5 (2): 155–8. Дои:10.4161 / cc.5.2.2367. PMID 16397413.
- ^ а б c Чжан X, Язаки Дж., Сундаресан А. и др. (Сентябрь 2006 г.). «Полногеномное картирование с высоким разрешением и функциональный анализ метилирования ДНК арабидопсиса». Клетка. 126 (6): 1189–201. Дои:10.1016 / j.cell.2006.08.003. PMID 16949657.
- ^ Микроматрица метилирования ДНК
- ^ "Illumina | Секвенирование и основанные на массивах решения для генетических исследований".
- ^ «Архивная копия». Архивировано из оригинал на 2008-03-18. Получено 2008-03-18.CS1 maint: заархивированная копия как заголовок (ссылка на сайт)
- ^ Ли Х, Руан Дж, Дурбин Р. (ноябрь 2008 г.). «Картирование считываний коротких последовательностей ДНК и вызов вариантов с использованием показателей качества картирования». Genome Res. 18 (11): 1851–8. Дои:10.1101 / гр.078212.108. ЧВК 2577856. PMID 18714091.
- ^ Эстеллер М (апрель 2007 г.). «Эпигенетическое подавление гена при раке: гиперметилом ДНК». Гм. Мол. Genet. 16 (ТУ №1): Р50–9. Дои:10.1093 / hmg / ddm018. PMID 17613547.
- ^ Гилехер П., Хартнетт Л., Иган Л.Дж., Голден А., Раджа Али Р.А., Сойхе С. (июнь 2013 г.). «Анализ генного набора сильно предвзят, когда применяется к данным метилирования всего генома». Биоинформатика. 29 (15): 1851–7. Дои:10.1093 / биоинформатика / btt311. PMID 23732277.
- ^ Кешет И., Шлезингер Ю., Фаркаш С. и др. (Февраль 2006 г.). «Доказательства поучительного механизма метилирования de novo в раковых клетках». Nat. Genet. 38 (2): 149–53. Дои:10,1038 / ng1719. PMID 16444255.
- ^ Новак П., Дженсен Т., Оширо М.М. и др. (Ноябрь 2006 г.). «Эпигенетическая инактивация кластера генов HOXA при раке груди». Рак Res. 66 (22): 10664–70. Дои:10.1158 / 0008-5472.CAN-06-2761. PMID 17090521.
