Искусственный синтез генов - Artificial gene synthesis

Искусственный синтез генов, или же синтез генов, относится к группе методов, которые используются в синтетическая биология построить и собрать гены из нуклеотидов de novo. В отличие от ДНК Для синтеза в живых клетках искусственный синтез генов не требует матричной ДНК, что позволяет синтезировать практически любую последовательность ДНК в лаборатории. Он состоит из двух основных этапов, первый из которых твердофазный синтез ДНК, иногда известный как Печать ДНК.[1] Это дает фрагменты олигонуклеотидов, которые обычно составляют менее 200 пар оснований. Затем второй этап включает соединение этих олигонуклеотидных фрагментов с использованием различных методов сборки ДНК. Поскольку для искусственного генного синтеза не требуется матричная ДНК, теоретически возможно создать полностью синтетические молекулы ДНК без ограничений по нуклеотидной последовательности или размеру.
Синтез первого полного гена - дрожжей тРНК, был продемонстрирован Хар Гобинд Кхорана и сотрудниками в 1972 году.[2] Синтез первого пептид - и белок -кодирование генов проводилось в лабораториях Герберт Бойер и Александр Маркхэм, соответственно.[3][4] Совсем недавно были разработаны методы искусственного синтеза генов, которые позволят собирать целые хромосомы и геномы. Первый синтетический дрожжи хромосома была синтезирована в 2014 г., и весь функциональный бактериальный хромосомы также были синтезированы.[5] Кроме того, в будущем искусственный синтез генов может использовать новые азотистое основание пары (неестественные пары оснований).[6][7][8]
Стандартные методы синтеза ДНК
Синтез олигонуклеотидов
Олигонуклеотиды химически синтезированы с использованием строительных блоков, называемых нуклеозидами фосфорамидиты. Это могут быть нормальные или модифицированные нуклеозиды, которые имеют защитные группы для предотвращения неправильного взаимодействия их аминов, гидроксильных групп и фосфатных групп. За один раз добавляют один фосфорамидит, снимают защиту с 5'-гидроксильной группы, добавляют новое основание и так далее. Цепь растет в направлении от 3 'к 5', что является обратным по отношению к биосинтезу. В конце все защитные группы удаляются. Тем не менее, поскольку это химический процесс, происходит несколько неправильных взаимодействий, приводящих к появлению дефектных продуктов. Чем длиннее олигонуклеотидная последовательность, которая синтезируется, тем больше дефектов, поэтому этот процесс применим только для получения коротких последовательностей нуклеотиды. Текущий практический предел составляет около 200 б.п. (пар оснований ) для олигонуклеотида с достаточным качеством для непосредственного использования в биологических целях. ВЭЖХ может использоваться для выделения продуктов в правильной последовательности. Между тем, большое количество олигонуклеотидов можно синтезировать параллельно на генные чипы. Для оптимального выполнения последующих процедур синтеза генов их следует готовить индивидуально и в больших масштабах.
Отжиг на основе соединения олигонуклеотидов
Обычно набор индивидуально разработанных олигонуклеотидов изготавливается на автоматических твердофазных синтезаторах, очищается и затем соединяется специальными отжиг и стандарт перевязка или же полимераза реакции. Для повышения специфичности отжига олигонуклеотидов на стадии синтеза используется набор термостабильных ДНК. лигаза и полимераза ферменты. На сегодняшний день описано несколько методов синтеза генов, таких как лигирование фосфорилированных перекрывающихся олигонуклеотидов,[2][3] метод Фока I[4] и модифицированная форма лигазная цепная реакция для синтеза генов. Кроме того, несколько ПЦР Были описаны подходы к сборке.[9] Обычно в них используются олигонуклеотиды длиной 40-50 нуклеотидов, которые перекрывают друг друга. Эти олигонуклеотиды сконструированы так, чтобы покрывать большую часть последовательности обеих цепей, и полноразмерная молекула постепенно генерируется с помощью ПЦР с удлинением перекрытия (OE),[9] термодинамически сбалансированная ПЦР наизнанку (TBIO)[10] или комбинированные подходы.[11] Чаще всего синтезируемые гены имеют размер от 600 до 1200 п.н., хотя гораздо более длинные гены были получены путем соединения ранее собранных фрагментов длиной менее 1000 п.н. В этом диапазоне размеров необходимо протестировать несколько клонов-кандидатов, подтверждающих последовательность клонированного синтетического гена с помощью методов автоматического секвенирования.
Ограничения
Более того, поскольку сборка полноразмерного генного продукта зависит от эффективного и специфического выравнивания длинных одноцепочечных олигонуклеотидов, критические параметры для успеха синтеза включают участки с расширенной последовательностью, содержащие вторичные структуры, вызванные инвертированными повторами, чрезвычайно высоким или низким содержанием GC, или повторяющиеся структуры. Обычно эти сегменты конкретного гена могут быть синтезированы только путем разделения процедуры на несколько последовательных шагов и окончательной сборки более коротких подпоследовательностей, что, в свою очередь, приводит к значительному увеличению времени и трудозатрат, необходимых для его получения. Эксперимент по синтезу генов сильно зависит от качества используемых олигонуклеотидов. Для этих протоколов синтеза генов на основе отжига качество продукта прямо и экспоненциально зависит от правильности используемых олигонуклеотидов. В качестве альтернативы, после выполнения синтеза гена с олигонуклеотами более низкого качества необходимо приложить больше усилий для последующего обеспечения качества во время анализа клонов, который обычно выполняется с помощью трудоемких стандартных процедур клонирования и секвенирования. Еще одна проблема, связанная со всеми текущими методами синтеза генов, - это высокая частота ошибок последовательности из-за использования химически синтезированных олигонуклеотидов. Частота ошибок увеличивается с более длинными олигонуклеотидами, и, как следствие, процент правильного продукта резко снижается по мере использования большего количества олигонуклеотидов. Проблема мутации может быть решена с помощью более коротких олигонуклеотидов, используемых для сборки гена. Однако все методы сборки на основе отжига требуют, чтобы праймеры были смешаны вместе в одной пробирке. В этом случае более короткие перекрытия не всегда позволяют точно и специфично отжигать комплементарные праймеры, что приводит к ингибированию образования полноразмерных продуктов. Дизайн олигонуклеотидов вручную является трудоемкой процедурой и не гарантирует успешный синтез желаемого гена. Для оптимального выполнения почти всех методов, основанных на отжиге, температуры плавления перекрывающихся областей должны быть одинаковыми для всех олигонуклеотидов. Необходимая оптимизация праймеров должна выполняться с использованием специализированных программ конструирования олигонуклеотидов. К настоящему времени представлено несколько решений для автоматизированного конструирования праймеров для синтеза генов.[12][13][14]
Процедуры исправления ошибок
Чтобы преодолеть проблемы, связанные с качеством олигонуклеотидов, было разработано несколько тщательно продуманных стратегий, в которых используются либо отдельно приготовленные рыболовные олигонуклеотиды, либо[15] несоответствие связывающих ферментов семья mutS[16] или специфические эндонуклеазы из бактерий или фагов.[17] Тем не менее, все эти стратегии увеличивают время и затраты на синтез генов, основанный на отжиге химически синтезированных олигонуклеотидов.
Массивно параллельное секвенирование также использовалось в качестве инструмента для скрининга сложных библиотек олигонуклеотидов и обеспечения возможности получения точных молекул. В одном подходе олигонуклеотиды секвенируют на платформе пиросеквенирования 454, и роботизированная система создает изображения и выбирает отдельные шарики, соответствующие точной последовательности.[18] В другом подходе сложная библиотека олигонуклеотидов модифицируется уникальными фланкирующими тегами перед массовым параллельным секвенированием. Затем праймеры, ориентированные на метки, позволяют извлекать молекулы с желаемыми последовательностями с помощью исходящей ПЦР.[19]
Все чаще гены упорядочиваются в наборах, включая функционально связанные гены или несколько вариантов последовательностей одного гена. Практически все разрабатываемые терапевтические белки, такие как моноклональные антитела, оптимизированы путем тестирования многих вариантов генов на улучшенную функцию или экспрессию.
Неестественные пары оснований
В то время как традиционный синтез нуклеиновых кислот использует только 4 пары оснований - аденин, тимин, гуанин и цитозин, синтез олигонуклеотидов в будущем может включать использование неестественных пар оснований, которые являются искусственно созданными и синтезированными азотистыми основаниями, которые не встречаются в природе.
В 2012 году группа американских ученых во главе с Флойдом Ромесбергом, химическим биологом из Научно-исследовательский институт Скриппса в Сан-Диего, Калифорния, опубликовал, что его команда разработала неестественную пару оснований (UBP). Два новых искусственных нуклеотида или Неестественная базовая пара (УБП) были названы d5SICS и dNaM. Технически эти искусственные нуклеотиды несущий гидрофобный азотистые основания, имеют две слитные ароматические кольца которые образуют комплекс (d5SICS – dNaM) или пару оснований в ДНК. В 2014 году та же команда из Исследовательского института Скриппса сообщила, что они синтезировали отрезок кольцевой ДНК, известный как плазмида содержащий естественные пары оснований T-A и C-G вместе с наиболее эффективным UBP, разработанным лабораторией Ромесберга, и вставлял его в клетки общей бактерии Кишечная палочка которые успешно воспроизвели неестественные пары оснований в нескольких поколениях. Это первый известный пример передачи живым организмом расширенного генетического кода последующим поколениям. Частично это было достигнуто путем добавления поддерживающего гена водорослей, который экспрессирует нуклеотидтрифосфат транспортер, который эффективно импортирует трифосфаты как d5SICSTP, так и dNaMTP в Кишечная палочка бактерии. Затем естественные пути репликации бактерий используют их для точного воспроизведения плазмида содержащий d5SICS – dNaM.
Успешное включение третьей пары оснований является значительным прорывом на пути к цели значительного увеличения числа аминокислоты которые могут быть кодированы ДНК, от существующих 20 аминокислот до теоретически возможных 172, тем самым расширяя потенциал живых организмов для производства новых белки.[20] В будущем эти неестественные пары оснований можно будет синтезировать и включить в олигонуклеотиды с помощью методов печати ДНК.
Сборка ДНК
Таким образом, печать ДНК может использоваться для производства частей ДНК, которые определяются как последовательности ДНК, кодирующие конкретную биологическую функцию (например, промоутеры, регуляторные последовательности транскрипции или открытые рамки для чтения ).[21] Однако, поскольку синтез олигонуклеотидов обычно не может точно производить последовательности олигонуклеотидов длиной более нескольких сотен пар оснований, необходимо использовать методы сборки ДНК для сборки этих частей вместе для создания функциональных генов, мультигенных цепей или даже целых синтетических хромосом или геномов. Некоторые методы сборки ДНК определяют только протоколы соединения частей ДНК, в то время как другие методы также определяют правила для формата частей ДНК, совместимых с ними. Эти процессы можно масштабировать, чтобы обеспечить сборку целых хромосом или геномов. В последние годы увеличилось количество различных стандартов сборки ДНК, и по состоянию на 2015 год было разработано 14 различных стандартов сборки, каждый со своими плюсами и минусами.[22] В целом, разработка стандартов сборки ДНК значительно облегчила рабочий процесс синтетической биологии, помогла обмену материалом между исследовательскими группами, а также позволила создавать модульные и повторно используемые части ДНК.[22]
Различные методы сборки ДНК можно разделить на три основные категории - сборка, опосредованная эндонуклеазами, сайт-специфическая рекомбинация и сборка на основе длинного перекрытия.[22] Каждая группа методов имеет свои отличительные особенности, а также свои преимущества и ограничения.
Сборка, опосредованная эндонуклеазой
Эндонуклеазы представляют собой ферменты, которые распознают и расщепляют сегменты нуклеиновых кислот, и их можно использовать для управления сборкой ДНК. Из различных типов рестрикционных ферментов рестрикционные ферменты типа II являются наиболее общедоступными и используются, поскольку их сайты расщепления расположены рядом или в их сайтах узнавания. Следовательно, методы сборки, опосредованные эндонуклеазами, используют это свойство для определения частей ДНК и протоколов сборки.
BioBricks

Стандарт сборки BioBricks был описан и представлен Томом Найтом в 2003 году и с тех пор постоянно обновляется.[23] В настоящее время наиболее часто используемым стандартом BioBricks является стандарт сборки 10 или BBF RFC 10. BioBricks определяет последовательность префиксов и суффиксов, необходимых для совместимости части ДНК с методом сборки BioBricks, что позволяет соединять вместе все части ДНК, которые являются в формате BioBricks.
Префикс содержит сайты рестрикции для EcoRI, NotI и XBaI, а суффикс содержит сайты рестрикции SpeI, NotI и PstI. Вне области префикса и суффикса часть ДНК не должна содержать эти сайты рестрикции. Чтобы соединить две части BioBrick вместе, одна из плазмид расщепляется EcoRI и SpeI, а вторая плазмида расщепляется EcoRI и XbaI. Два выступа EcoRI дополняют друг друга и, таким образом, отжигаются вместе, в то время как SpeI и XbaI также образуют дополнительные выступы, которые также можно лигировать вместе. Поскольку полученная плазмида содержит исходные последовательности префикса и суффикса, ее можно использовать для соединения с другими частями BioBricks.[24] Из-за этого свойства стандарт сборки BioBricks называется идемпотент в природе. Однако между двумя слитыми BioBricks будет также образовываться «рубцовая» последовательность (TACTAG или TACTAGAG). Это предотвращает использование BioBricks для создания слитых белков, поскольку последовательность рубца длиной 6 пар оснований кодирует тирозин и стоп-кодон, что приводит к прекращению трансляции после экспрессии первого домена, в то время как последовательность рубца длиной 8 пар оснований вызывает сдвиг рамки, предотвращая непрерывное считывание кодонов. Чтобы предложить альтернативные рубцовые последовательности, которые, например, дают рубец в 6 пар оснований, или последовательности рубцов, которые не содержат стоп-кодоны, были разработаны другие стандарты сборки, такие как BB-2 Assembly, BglBricks Assembly, Silver Assembly и Freiburg Assembly.[25][26][27][28]
Хотя самый простой метод сборки деталей BioBrick описан выше, существует также несколько других широко используемых методов сборки, которые предлагают несколько преимуществ по сравнению со стандартной сборкой. Сборка из 3 антибиотиков (3A) позволяет выбрать правильную сборку посредством выбора антибиотика, в то время как сборка амплифицированной вставки стремится преодолеть низкую эффективность трансформации, наблюдаемую в сборке 3A.[29][30]
Стандарт сборки BioBrick также послужил вдохновением для использования других типов эндонуклеаз для сборки ДНК. Например, как стандарт iBrick, так и стандарты сборки вектора HomeRun используют самонаводящиеся эндонуклеазы вместо рестрикционных ферментов типа II.[31][32]
Сборка эндонуклеаз рестрикции типа II
Некоторые методы сборки также используют эндонуклеазы рестрикции типа II. Они отличаются от других эндонуклеаз типа II, поскольку они отрезают несколько пар оснований от сайта узнавания. В результате последовательность выступа может быть изменена, чтобы содержать желаемую последовательность. Это обеспечивает методы сборки Типа II с двумя преимуществами: он обеспечивает сборку без образования рубцов и позволяет производить сборку из нескольких частей в одной ванне. Способы сборки, в которых используются эндонуклеазы типа II, включают: Золотые ворота и связанные с ним варианты.
Клонирование Golden Gate

Протокол сборки Golden Gate был определен Engler et al. 2008, чтобы определить метод сборки ДНК, который дал бы окончательную конструкцию без рубцовой последовательности, а также без исходных сайтов рестрикции. Это позволяет белку экспрессироваться без содержания нежелательных белковых последовательностей, которые могут отрицательно повлиять на укладку или экспрессию белка. При использовании рестрикционного фермента BsaI, который производит выступ из 4 пар оснований, для сборки можно использовать до 240 уникальных непалиндромных последовательностей.[33]
Дизайн и сборка плазмид
При клонировании Golden Gate каждый собираемый фрагмент ДНК помещают в плазмиду, фланкируемую обращенными внутрь сайтами рестрикции BsaI, содержащими запрограммированные выступающие последовательности. Для каждого фрагмента ДНК 3'-выступающая последовательность комплементарна 5'-выступающей части следующего нижестоящего фрагмента ДНК. Для первого фрагмента 5'-выступ комплементарен 5'-выступу целевой плазмиды, в то время как 3'-выступ последнего фрагмента комплементарен 3'-выступу целевой плазмиды. Такая конструкция позволяет собрать все фрагменты ДНК в реакции в одном сосуде (где все реагенты смешиваются вместе), при этом все фрагменты расположены в правильной последовательности. Успешно собранные конструкции отбирают путем обнаружения потери функции кассеты для скрининга, которая изначально находилась в целевой плазмиде.[33]
MoClo и золотая коса
Исходная сборка Golden Gate позволяет создать только одну конструкцию в векторе назначения. Чтобы позволить использовать эту конструкцию в последующей реакции в качестве вектора входа, были разработаны стандарты MoClo и Golden Braid.[34]
Стандарт MoClo включает определение нескольких уровней сборки ДНК:
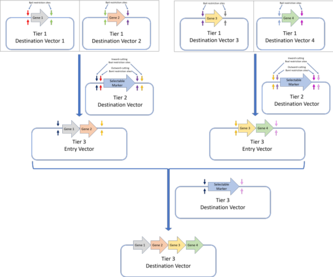
- Уровень 1: Сборка уровня 1 - это стандартная сборка Golden Gate, и гены собираются из их составных частей (частей ДНК, кодирующих такие генетические элементы, как UTR, промоутеры, сайты связывания рибосом или же терминатор последовательности). По бокам от сайта вставки векторов-мишеней уровня 1 находится пара сайтов рестрикции BpiI, разрезанных внутрь. Это позволяет использовать эти плазмиды в качестве векторов входа для векторов назначения второго уровня.Стандарт сборки MoClo позволяет собирать конструкции Golden Gate на последующих уровнях. В приведенном здесь примере четыре гена, собранные с помощью сборки Golden Gate 1 уровня, собраны в мультигенную конструкцию на сборке 2 уровня.Стандарт сборки Golden Braid также основан на первом уровне сборки Golden Gate и собирает последующие уровни с помощью попарного протокола. Четыре вектора назначения уровня 1 (собранные с помощью сборки Golden Gate) собираются в два вектора назначения уровня 2, которые затем используются в качестве векторов входа уровня 3 для вектора назначения уровня 3. Используются чередующиеся рестрикционные ферменты (BpiI для уровня 2 и BsaI для уровня 3).Стандарты сборки MoClo и Golden Braid являются производными от исходного стандарта сборки Golden Gate.
- Уровень 2: Сборка уровня 2 включает дальнейшую сборку генов, собранных в сборке уровня 1, в мультигенные конструкции. Если есть необходимость в дальнейшей сборке более высокого уровня, можно добавить сайты рестрикции BsaI с внутренним разрезом, чтобы фланкировать сайты вставки. Эти векторы затем можно использовать в качестве входных векторов для конструкций более высокого уровня.
Каждый уровень сборки чередует использование сайтов рестрикции BsaI и BpiI, чтобы минимизировать количество запрещенных сайтов, а последовательная сборка для каждого уровня достигается с помощью дизайна плазмиды Golden Gate. В целом, стандарт MoClo позволяет собрать конструкцию, содержащую несколько единиц транскрипции, все из которых собраны из разных частей ДНК, с помощью серии реакций Golden Gate в одном сосуде. Однако одним из недостатков стандарта MoClo является то, что он требует использования «фиктивных частей» без биологической функции, если для окончательной конструкции требуется менее четырех составных частей.[35] Стандарт Golden Braid, с другой стороны, ввел стандарт попарной сборки Golden Gate.
Стандарт Golden Braid использует ту же многоуровневую сборку, что и MoClo, но каждый уровень включает сборку только двух фрагментов ДНК, то есть попарный подход. Следовательно, на каждом уровне пары генов клонируются в целевой фрагмент в желаемой последовательности, а затем они собираются по два за раз в последовательных уровнях. Как и MoClo, стандарт Golden Braid чередует рестрикционные ферменты BsaI и BpiI на каждом уровне.
Разработка методов сборки Golden Gate и его вариантов позволила исследователям разработать наборы инструментов для ускорения рабочего процесса синтетической биологии. Например, EcoFlex был разработан как набор инструментов для E. Coli который использует стандарт MoClo для своих частей ДНК, в то время как аналогичный набор инструментов был также разработан для разработки Chlamydomonas reinhardtii mircoalgae.[36][37]
Сайт-специфическая рекомбинация
В сайт-специфической рекомбинации используется фаг. интегрирует вместо ферментов рестрикции, что устраняет необходимость наличия сайтов рестрикции во фрагментах ДНК. Вместо этого интегразы используют уникальные сайты присоединения (att) и катализируют перестройку ДНК между целевым фрагментом и вектором-получателем. Система клонирования Invitrogen Gateway была изобретена в конце 1990-х и использует две запатентованные смеси ферментов: клоназу BP и клоназу LR. Смесь клоназ BP катализирует рекомбинацию между сайтами attB и attP, генерируя гибридные сайты attL и attR, в то время как смесь клоназ LR катализирует рекомбинацию сайтов attL и attR с образованием сайтов attB и attP. Поскольку каждая смесь ферментов распознает только определенные сайты att, рекомбинация высокоспецифична, и фрагменты могут быть собраны в желаемой последовательности.[38]
Векторный дизайн и сборка
Поскольку клонирование шлюза является запатентованной технологией, все реакции шлюза должны выполняться с помощью комплекта шлюза, предоставляемого производителем. Реакцию можно разделить на две стадии. Первый этап включает сборку входных клонов, содержащих интересующий фрагмент ДНК, а второй этап включает вставку этого интересующего фрагмента в целевой клон.
- Входные клоны должны быть созданы с использованием поставляемых «донорных» векторов, содержащих кассету Gateway, фланкированную сайтами attP. Кассета Gateway содержит бактериальный суицидный ген (например, ccdB ), что позволит выжить и отобрать успешно рекомбинированные исходные клоны. Пара сайтов attB добавляется для фланкирования интересующего фрагмента ДНК, и это позволит рекомбинировать с сайтами attP при добавлении смеси клоназ ВР. Производятся входные клоны, и интересующий фрагмент фланкируется сайтами attL.
- Вектор назначения также поставляется с кассетой шлюза, но вместо этого он окружен парой сайтов attR. Смешивание этой целевой плазмиды с входными клонами и смесью LR-клоназ позволит рекомбинации между сайтами attR и attL. Создается целевой клон с успешно вставленным интересующим фрагментом. Летальный ген вставляется в исходный вектор, и бактерии, трансформированные этой плазмидой, погибнут. Таким образом, можно легко выбрать желаемый вектор.
Самые ранние итерации метода клонирования шлюза позволяли использовать только один клон записи для каждого созданного клона назначения. Однако дальнейшие исследования показали, что могут быть сгенерированы еще четыре ортогональные последовательности att, позволяющие собрать до четырех различных фрагментов ДНК, и этот процесс теперь известен как технология Multisite Gateway.[39]
Помимо клонирования шлюза, были также разработаны некоммерческие методы с использованием других интеграций. Например, метод рекомбинационной сборки сериновой интегразы (SIRA) использует интегразу ϕC31, в то время как метод тандемной сборки на основе сайт-специфической рекомбинации (SSRTA) использует метод Streptomyces фаг φBT1 интегразы.[40][41] Другие методы, такие как система сборки вектора HomeRun (HVAS), основаны на системе клонирования Gateway и дополнительно включают эндоуклазы самонаведения для разработки протокола, который потенциально может поддерживать промышленный синтез синтетических конструкций ДНК.[31]

Сборка с длинным перекрытием
В последние годы было разработано множество методов сборки с длинным перекрытием. Один из наиболее часто используемых методов, метод сборки Гибсона, был разработан в 2009 году и обеспечивает метод сборки ДНК в одном горшке, который не требует использования рестрикционных ферментов или интеграз.[42] Другие аналогичные методы сборки на основе перекрытия включают клонирование с расширением круговой полимеразы (CPEC), независимое от последовательностей и лигаз клонирование (SLIC) и экстракт клонирования с бесшовным лигированием (SLiCE).[43][44][45] Несмотря на наличие множества методов сборки внахлест, метод сборки Гибсона по-прежнему остается наиболее популярным.[46] Помимо методов, перечисленных выше, другие исследователи основывались на концепциях, используемых в сборке Гибсона и других методах сборки, для разработки новых стратегий сборки, таких как модульная сборка, направленная на перекрытие, с линкерами (MODAL) или стандарт сборки Biopart для идемпотентного клонирования (BASIC). ) метод.[47][48]
Сборка Гибсона
Метод сборки Гибсона - относительно простой метод сборки ДНК, требующий всего нескольких дополнительных реагентов: 5 'T5 экзонуклеаза, Phusion ДНК-полимераза, и Taq ДНК-лигаза. Фрагменты ДНК, которые должны быть собраны, синтезируются так, чтобы иметь перекрывающиеся 5'- и 3'-концы в том порядке, в котором они должны быть собраны. Эти реагенты смешиваются вместе с фрагментами ДНК, которые необходимо собрать, при 50 ° C, и происходят следующие реакции:
- Экзонуклеаза Т5 отщепляет ДНК с 5'-конца каждого фрагмента, обнажая 3'-выступающие части на каждом фрагменте ДНК.
- Комплементарные выступы на соседних фрагментах ДНК отжигаются посредством комплементарного спаривания оснований.
- ДНК-полимераза Phusion заполняет все пробелы в местах отжига фрагментов.
- ДНК-лигаза Taq восстанавливает порезы на обеих цепях ДНК.
Поскольку экзонуклеаза Т5 термолабильна, она инактивируется при 50 ° C после первого жевания.Таким образом, продукт стабилен, а фрагменты собраны в желаемом порядке. Этот протокол с одним горшком может точно собрать до 5 различных фрагментов, в то время как несколько коммерческих поставщиков имеют наборы для точной сборки до 15 различных фрагментов в двухэтапной реакции.[49] Однако, хотя протокол сборки Гибсона является быстрым и использует относительно небольшое количество реагентов, он требует индивидуального синтеза ДНК, поскольку каждый фрагмент должен быть сконструирован таким образом, чтобы содержать перекрывающиеся последовательности с соседними фрагментами и амплифицировать с помощью ПЦР. Эта зависимость от ПЦР может также повлиять на точность реакции при использовании длинных фрагментов, фрагментов с высоким содержанием GC или повторяющихся последовательностей.[48]

МОДАЛЬНЫЙ
Стратегия MODAL определяет перекрывающиеся последовательности, известные как «линкеры», чтобы уменьшить количество настроек, которые необходимо выполнить с каждым фрагментом ДНК. Линкеры были разработаны с использованием R2oDNA Designer программное обеспечение и области перекрытия были разработаны так, чтобы иметь длину 45 п.н., чтобы быть совместимыми с сборкой Гибсона и другими способами сборки с перекрытием. Чтобы прикрепить эти линкеры к собираемым частям, проводят ПЦР с использованием специфичных для частей праймеров, содержащих последовательности префикса 15 п.н. Затем линкеры присоединяются к последовательностям адаптера посредством второй реакции ПЦР. Чтобы расположить фрагменты ДНК, один и тот же линкер будет присоединен к суффиксу желаемого восходящего фрагмента и к префиксу желаемых нисходящих фрагментов. После присоединения линкеров сборка Гибсона, CPEC или другие способы сборки с перекрытием могут быть использованы для сборки фрагментов ДНК в желаемом порядке.
БАЗОВЫЙ
Стратегия сборки BASIC была разработана в 2015 году и направлена на устранение ограничений предыдущих методов сборки, включая шесть ключевых концепций из них: стандартные детали многократного использования; одноуровневый формат (все детали одного формата и собираются по одному и тому же процессу); идемпотентное клонирование; параллельная (составная) сборка ДНК; независимость от размера; автоматичность.[48]
Части ДНК и дизайн линкера
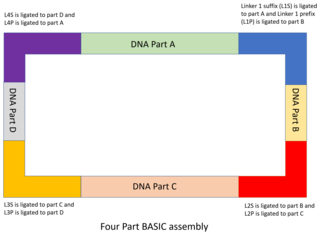
Части ДНК сконструированы и клонированы в плазмиды для хранения, при этом часть окружена встроенным префиксом (яP) и интегрированный суффикс (яS) последовательность. В яP и яПоследовательности S содержат обращенные внутрь сайты рестрикции BsaI, которые содержат выступы, комплементарные линкерам BASIC.[48] Как и в MODAL, 7 стандартных линкеров, используемых в BASIC, были разработаны с помощью программного обеспечения R2oDNA Designer и проверены, чтобы убедиться, что они не содержат последовательностей, гомологичных геномам шасси, и что они не содержат нежелательных последовательностей, таких как последовательности вторичной структуры, сайты рестрикции. или сайты связывания рибосом. Каждая линкерная последовательность разделяется на две половины, каждая с выступом из 4 пар оснований, комплементарным сайту рестрикции BsaI, двухцепочечной последовательностью 12 пар оснований и разделяющей перекрывающуюся последовательность 21 пар оснований с другой половиной. Половина, которая будет связываться с вышестоящей частью ДНК, известна как суффиксная линкерная часть (например, L1S), а половина, которая связывается с нижней частью, известна как префиксная линкерная часть (например, L1P). Эти линкеры составляют основу сборки частей ДНК вместе.
Помимо управления порядком сборки, стандартные линкеры BASIC также могут быть изменены для выполнения других функций. Чтобы обеспечить идемпотентную сборку, линкеры были также разработаны с дополнительным метилированным яP и яПоследовательности S вставлены для защиты их от распознавания BsaI. Это метилирование теряется после трансформации и репликации плазмиды in vivo, и плазмиды можно экстрагировать, очищать и использовать для дальнейших реакций.
Поскольку линкерные последовательности относительно длинные (45 п.н. для стандартного линкера), есть возможность включить функциональные последовательности ДНК для уменьшения количества частей ДНК, необходимых во время сборки. Стандарт сборки BASIC предоставляет несколько линкеров, встроенных в RBS разной силы. Подобно тому, чтобы облегчить конструирование слитых белков, содержащих несколько белковых доменов, были также разработаны несколько слитых линкеров, обеспечивающих полное считывание конструкции ДНК. Эти слитые линкеры кодируют полипептид глицина и серина из 15 аминокислот, который является идеальным линкерным пептидом для слитых белков с несколькими доменами.
сборка
Сборка окончательной конструкции состоит из трех основных этапов.
- Сначала части ДНК вырезаются из запасающей плазмиды, давая фрагмент ДНК с выступами BsaI на 3 'и 5' концах.
- Затем каждая линкерная часть присоединяется к соответствующей части ДНК путем инкубации с ДНК-лигазой Т4. Каждая часть ДНК будет иметь суффикс и префикс линкерной части из двух разных линкеров, чтобы определять порядок сборки. Например, первая часть последовательности будет иметь L1P и L2S, а вторая часть будет иметь присоединенные L2P и L3S. Детали компоновщика можно изменить, чтобы изменить последовательность сборки.
- Наконец, части с прикрепленными линкерами собирают в плазмиду путем инкубации при 50 ° C. Выступающие части линкеров P и S размером 21 п.н. отжигаются, и конечная конструкция может быть трансформирована в клетки бактерий для клонирования. Ремонт однонитевых зарубок in vivo после трансформации получают стабильную конечную конструкцию, клонированную в плазмиды.
Приложения
Поскольку методы печати ДНК и сборки ДНК позволили коммерческому синтезу генов прогрессивно и экспоненциально дешеветь за последние годы,[50] искусственный синтез генов представляет собой мощный и гибкий инженерный инструмент для создания и конструирования новых последовательностей ДНК и функций белков. Помимо синтетической биологии, в различных областях исследований, таких как экспрессия гетерологичного гена, вакцина разработка, генная терапия и молекулярная инженерия, значительно выиграют от наличия быстрых и дешевых методов синтеза ДНК для кодирования белков и пептидов.[51] Методы, используемые для печати и сборки ДНК, даже позволили использовать ДНК в качестве носитель информации.
Синтез бактериальных геномов
Синтия и Лаборатория микоплазм
28 июня 2007 г. Институт Дж. Крейга Вентера опубликовал статью в Science Express, заявив, что они успешно трансплантировали естественную ДНК из Mycoplasma mycoides бактерия в Mycoplasma capricolum клетки, создавая бактерию, которая вела себя как М. mycoides.[52]
6 октября 2007 г. Крейг Вентер объявил в интервью британскому Хранитель газете, что та же команда синтезировала модифицированную версию сингла хромосома из Mycoplasma genitalium искусственно. Хромосома была изменена, чтобы удалить все гены, в которых тесты на живых бактериях показали ненужность. Следующий запланированный шаг в этом минимальный проект генома заключается в трансплантации синтезированного минимального генома в бактериальную клетку с удалением ее старой ДНК; получившаяся бактерия будет называться Лаборатория микоплазм. На следующий день канадец биоэтика группа, ETC Group сделали заявление через своего представителя, Пэт Муни, говоря, что «творением» Вентера было «шасси, на котором можно построить почти все». Синтезированный геном еще не был пересажен в рабочую клетку.[53]
21 мая 2010 г. Наука сообщил, что группа Вентера успешно синтезировала геном бактерии Mycoplasma mycoides из компьютерной записи и трансплантировал синтезированный геном в существующую клетку Mycoplasma capricolum бактерия, у которой была удалена ДНК. «Синтетическая» бактерия была жизнеспособной, т. Е. Способной воспроизводиться миллиарды раз. Изначально команда планировала использовать М. genitalium бактерии, с которыми они раньше работали, но перешли на М. mycoides потому что последняя бактерия растет намного быстрее, что привело к более быстрым экспериментам.[54] Вентер описывает его как «первый вид… родителями которого стали компьютеры».[55] Трансформированная бактерия получила название "Synthia "ETC. Представитель Venter отказался подтвердить какие-либо открытия на момент написания этой статьи.
Синтетические дрожжи 2.0
В рамках проекта Synthetic Yeast 2.0 различные исследовательские группы по всему миру приняли участие в проекте по синтезу синтетических геномов дрожжей и, с помощью этого процесса, оптимизации генома модельного организма. Saccharomyces cerevisae.[56] В проекте Yeast 2.0 использовались различные методы сборки ДНК, которые обсуждались выше, и в марте 2014 г. Джеф Боке из Медицинского центра Лангоне при Нью-Йоркском университете, выяснилось, что его команда синтезировала хромосому III S. cerevisae.[57][58] Процедура включала замену генов в исходной хромосоме синтетическими версиями, а готовая синтетическая хромосома затем была интегрирована в дрожжевую клетку. Это потребовало разработки и создания 273 871 пары оснований ДНК - меньше, чем 316 667 пар в исходной хромосоме. В марте 2017 года синтез 6 из 16 хромосом был завершен, а синтез остальных все еще продолжается.[59]
Смотрите также
Примечания
- ^ Штейн Р. (7 мая 2015 г.). "ДНК" печатает "большое благо для исследований, но некоторые вызывают опасения". Все учтено. Национальное общественное радио.
- ^ а б Khorana HG, Agarwal KL, Büchi H, Caruthers MH, Gupta NK, Kleppe K и др. (Декабрь 1972 г.). «Исследования полинуклеотидов. 103. Полный синтез структурного гена рибонуклеиновой кислоты переноса аланина из дрожжей». Журнал молекулярной биологии. 72 (2): 209–17. Дои:10.1016/0022-2836(72)90146-5. PMID 4571075.
- ^ а б Итакура К., Хиросе Т., Креа Р., Риггс А.Д., Хейнекер Х.Л., Боливар Ф., Бойер Х.В. (декабрь 1977 г.). «Экспрессия в Escherichia coli химически синтезированного гена гормона соматостатина». Наука. 198 (4321): 1056–63. Bibcode:1977Научный ... 198.1056I. Дои:10.1126 / science.412251. PMID 412251.
- ^ а б Edge MD, Green AR, Heathcliffe GR, Meacock PA, Schuch W., Scanlon DB и др. (Август 1981 г.). «Полный синтез гена интерферона лейкоцитов человека». Природа. 292 (5825): 756–62. Bibcode:1981Натура.292..756E. Дои:10.1038 / 292756a0. PMID 6167861. S2CID 4330168.
- ^ Шукман Д. (27.03.2014). "Прогресс синтетической ДНК приветствуется". Новости BBC. Получено 2020-04-11.
- ^ Кимото М., Ямашиге Р., Мацунага К., Йокояма С., Хирао И. (май 2013 г.). «Генерация высокоаффинных ДНК-аптамеров с использованием расширенного генетического алфавита». Природа Биотехнологии. 31 (5): 453–7. Дои:10.1038 / nbt.2556. PMID 23563318. S2CID 23329867.
- ^ Малышев Д.А., Дхами К., Quach HT, Lavergne T, Ordoukhanian P, Torkamani A, Romesberg FE (июль 2012 г.). «Эффективная и независимая от последовательности репликация ДНК, содержащей третью пару оснований, устанавливает функциональный шестибуквенный генетический алфавит». Труды Национальной академии наук Соединенных Штатов Америки. 109 (30): 12005–10. Bibcode:2012ПНАС..10912005М. Дои:10.1073 / pnas.1205176109. ЧВК 3409741. PMID 22773812.
- ^ Малышев Д.А., Дхами К., Лавергн Т., Чен Т., Дай Н., Фостер Дж. М. и др. (Май 2014 г.). «Полусинтетический организм с расширенным генетическим алфавитом». Природа. 509 (7500): 385–8. Bibcode:2014Натура.509..385M. Дои:10.1038 / природа13314. ЧВК 4058825. PMID 24805238.
- ^ а б Fuhrmann M, Oertel W, Hegemann P (август 1999). «Синтетический ген, кодирующий зеленый флуоресцентный белок (GFP), является универсальным репортером Chlamydomonas reinhardtii». Журнал растений. 19 (3): 353–61. Дои:10.1046 / j.1365-313X.1999.00526.x. PMID 10476082.
- ^ Mandecki W, Bolling TJ (август 1988 г.). «ФокИ метод генного синтеза». Ген. 68 (1): 101–7. Дои:10.1016/0378-1119(88)90603-8. PMID 3265397.
- ^ Стеммер В.П., Крамери А., Ха К.Д., Бреннан Т.М., Хейнекер Х.Л. (октябрь 1995 г.). «Одностадийная сборка гена и всей плазмиды из большого количества олигодезоксирибонуклеотидов». Ген. 164 (1): 49–53. Дои:10.1016/0378-1119(95)00511-4. PMID 7590320.
- ^ Гао X, Йо П, Кейт А., Раган Т.Дж., Харрис Т.К. (ноябрь 2003 г.). «Термодинамически сбалансированный синтез генов на основе ПЦР наизнанку (TBIO): новый метод конструирования праймеров для высокоточной сборки более длинных последовательностей генов». Исследования нуклеиновых кислот. 31 (22): 143e – 143. Дои:10.1093 / nar / gng143. ЧВК 275580. PMID 14602936.
- ^ Янг Л., Донг Кью (апрель 2004 г.). «Двухэтапный метод тотального синтеза генов». Исследования нуклеиновых кислот. 32 (7): e59. Дои:10.1093 / nar / gnh058. ЧВК 407838. PMID 15087491.
- ^ Хиллсон, штат Нью-Джерси, Розенгартен, Д. Д. Кислинг (январь 2012 г.). "Программное обеспечение для автоматизации проектирования сборки ДНК j5". Синтетическая биология ACS. 1 (1): 14–21. Дои:10.1021 / sb2000116. PMID 23651006.
- ^ Гувер Д.М., Лубковски Дж. (Май 2002 г.). «DNAWorks: автоматизированный метод конструирования олигонуклеотидов для синтеза генов на основе ПЦР». Исследования нуклеиновых кислот. 30 (10): 43e – 43. Дои:10.1093 / nar / 30.10.e43. ЧВК 115297. PMID 12000848.
- ^ Вильялобос А., Несс Дж. Э., Густафссон К., Миншалл Дж., Говиндараджан С. (июнь 2006 г.). «Конструктор генов: инструмент синтетической биологии для создания искусственных сегментов ДНК». BMC Bioinformatics. 7: 285. Дои:10.1186/1471-2105-7-285. ЧВК 1523223. PMID 16756672.
- ^ Тиан Дж, Гун Х, Шэн Н, Чжоу Х, Гулари Э, Гао Х, Церковь G (декабрь 2004 г.). «Точный мультиплексный синтез генов из программируемых микрочипов ДНК» (PDF). Природа. 432 (7020): 1050–4. Bibcode:2004 Натур.432.1050Т. Дои:10.1038 / природа03151. HDL:2027.42/62677. PMID 15616567. S2CID 4373350.
- ^ Matzas M, Stähler PF, Kefer N, Siebelt N, Boisguérin V, Leonard JT и др. (Декабрь 2010 г.). «Синтез генов высокой точности путем извлечения ДНК с подтвержденной последовательностью, идентифицированной с помощью высокопроизводительного пиросеквенирования». Природа Биотехнологии. 28 (12): 1291–4. Дои:10.1038 / nbt.1710. ЧВК 3579223. PMID 21113166.
- ^ Шварц Дж. Дж., Ли С., Шендур Дж. (Сентябрь 2012 г.). «Точный синтез генов с извлечением по меткам молекул ДНК с подтвержденной последовательностью». Методы природы. 9 (9): 913–5. Дои:10.1038 / nmeth.2137. ЧВК 3433648. PMID 22886093.
- ^ Вайдман К. (2017-12-06). «Расширяя генетический алфавит». Блог. Гарвардский университет. Получено 2020-04-17.
- ^ "Справка: синтетическая биология - parts.igem.org". parts.igem.org. Получено 2020-04-11.
- ^ а б c Казини А, Сторч М., Болдуин Г.С., Эллис Т. (сентябрь 2015 г.). «Кубики и чертежи: методы и стандарты сборки ДНК». Обзоры природы. Молекулярная клеточная биология. 16 (9): 568–76. Дои:10.1038 / nrm4014. HDL:10044/1/31281. PMID 26081612. S2CID 3502437.
- ^ Рыцарь Т (2003). «Идемпотентный векторный дизайн для стандартной сборки биокирпичей». HDL:1721.1/21168. Цитировать журнал требует
| журнал =(помощь) - ^ Рёкке Дж., Корвальд Э., Пар Дж., Ойос О, Лале Р. (2014). «Стандарты и методы сборки BioBrick и соответствующие программные инструменты». В Valla S, Lale R (ред.). Методы клонирования и сборки ДНК. Методы молекулярной биологии. 1116. Клифтон, Нью-Джерси, стр. 1–24. Дои:10.1007/978-1-62703-764-8_1. ISBN 978-1-62703-763-1. PMID 24395353.
- ^ Knight T (19 ноября 2008 г.). «Проект стандарта на биологические части биокирпича BB-2». HDL:1721.1/45139. Цитировать журнал требует
| журнал =(помощь) - ^ Андерсон Дж. К., Дьюбер Дж. Э., Легия М., Ву ГК, Голер Дж. А., Аркин А. П., Кислинг Дж. Д. (январь 2010 г.). «BglBricks: гибкий стандарт сборки биологических деталей». Журнал биологической инженерии. 4 (1): 1. Дои:10.1186/1754-1611-4-1. ЧВК 2822740. PMID 20205762.
- ^ Филипс I, Сильвер П (20 апреля 2006 г.). «Новая стратегия сборки биокирпича, разработанная для легкой инженерии белков». HDL:1721.1/32535. Цитировать журнал требует
| журнал =(помощь) - ^ Грюнберг Р., Арндт К., Мюллер К. (18 апреля 2009 г.). «Стандарт сборки биокирпича Fusion Protein (Фрайбург)». HDL:1721.1/45140. Цитировать журнал требует
| журнал =(помощь) - ^ Шетти Р., Лизаразо М., Реттберг Р., Найт Т.Ф. (2011). «Сборка стандартных биологических частей BioBrick с использованием трех антибиотиков». Методы в энзимологии. 498: 311–26. Дои:10.1016 / B978-0-12-385120-8.00013-9. HDL:1721.1/65066. ISBN 9780123851208. PMID 21601683.
- ^ Speer MA, Ричард Т.Л. (декабрь 2011 г.). «Усиленная сборка вставок: оптимизированный подход к стандартной сборке генетических схем BioBrickTM». Журнал биологической инженерии. 5 (1): 17. Дои:10.1186/1754-1611-5-17. ЧВК 3287150. PMID 22176971.
- ^ а б Ли М.В., Шукла Д., Родос Б.Х., Лалл А., Шу Дж., Мориарити Б.С., Ларгаэспада Д.А. (24.06.2014). «Система сборки вектора HomeRun: гибкая и стандартизированная система клонирования для сборки многомодульных конструкций ДНК». PLOS ONE. 9 (6): e100948. Bibcode:2014PLoSO ... 9j0948L. Дои:10.1371 / journal.pone.0100948. ЧВК 4069157. PMID 24959875.
- ^ Лю Дж.К., Чен У.Х., Рен С.Х., Чжао ГП, Ван Дж. (2014-10-20). «iBrick: новый стандарт итеративной сборки биологических частей с помощью самонаводящихся эндонуклеаз». PLOS ONE. 9 (10): e110852. Bibcode:2014PLoSO ... 9k0852L. Дои:10.1371 / journal.pone.0110852. ЧВК 4203835. PMID 25329380.
- ^ а б Энглер К., Кандзия Р., Мариллоннет С. (2008-11-05). "Одноразовый, одноэтапный прецизионный метод клонирования с высокой производительностью". PLOS ONE. 3 (11): e3647. Bibcode:2008PLoSO ... 3.3647E. Дои:10.1371 / journal.pone.0003647. ЧВК 2574415. PMID 18985154.
- ^ Вебер Э, Энглер С., Грюцнер Р., Вернер С., Мариллоннет С. (февраль 2011 г.). «Модульная система клонирования для стандартизированной сборки мультигенных конструкций». PLOS ONE. 6 (2): e16765. Bibcode:2011PLoSO ... 616765W. Дои:10.1371 / journal.pone.0016765. ЧВК 3041749. PMID 21364738.
- ^ Кляйн К.А., Эмде Л., Куиджперс А., Собецко П. (2019-10-17). «MoCloFlex: модульная, но гибкая система клонирования». Границы биоинженерии и биотехнологии. 7: 271. Дои:10.3389 / fbioe.2019.00271. ЧВК 6843054. PMID 31750294.
- ^ Мур SJ, Lai HE, Kelwick RJ, Chee SM, Bell DJ, Polizzi KM, Freemont PS (октябрь 2016 г.). «EcoFlex: Многофункциональный набор MoClo для синтетической биологии E. coli». Синтетическая биология ACS. 5 (10): 1059–1069. Дои:10.1021 / acssynbio.6b00031. PMID 27096716.
- ^ Crozet P, Navarro FJ, Willmund F, Mehrshahi P, Bakowski K, Lauersen KJ и др. (Сентябрь 2018 г.). "Рождение фотосинтетического каркаса: набор инструментов MoClo, позволяющий использовать синтетическую биологию в микроводорослях Chlamydomonas reinhardtii". Синтетическая биология ACS. 7 (9): 2074–2086. Дои:10.1021 / acssynbio.8b00251. PMID 30165733.
- ^ Рис-Хойес Дж. С., Уолхаут А. Дж. (Январь 2018 г.). "Шлюз рекомбинационного клонирования". Протоколы Колд-Спринг-Харбор. 2018 (1): pdb.top094912. Дои:10.1101 / pdb.top094912. ЧВК 5935001. PMID 29295908.
- ^ Sasaki Y, Sone T, Yoshida S, Yahata K, Hotta J, Chesnut JD и др. (Февраль 2004 г.). «Доказательства высокой специфичности и эффективности множественных сигналов рекомбинации при смешанном клонировании ДНК с помощью системы Multisite Gateway». Журнал биотехнологии. 107 (3): 233–43. Дои:10.1016 / j.jbiotec.2003.10.001. PMID 14736459.
- ^ Colloms SD, Merrick CA, Olorunniji FJ, Stark WM, Smith MC, Osbourn A и др. (Февраль 2014). «Быстрая сборка и модификация метаболических путей с использованием сайт-специфической рекомбинации серин-интегразы». Исследования нуклеиновых кислот. 42 (4): e23. Дои:10.1093 / nar / gkt1101. ЧВК 3936721. PMID 24225316.
- ^ Чжан Л., Чжао Г, Дин Х (2011-11-03). «Тандемная сборка кластера биосинтетических генов эпотилона посредством сайт-специфической рекомбинации in vitro». Научные отчеты. 1 (1): 141. Bibcode:2011НатСР ... 1Е.141З. Дои:10.1038 / srep00141. ЧВК 3216622. PMID 22355658.
- ^ Гибсон Д.Г., Янг Л., Чуанг Р.Й., Вентер Дж.С., Хатчисон, Калифорния, Смит, HO (май 2009 г.). «Ферментативная сборка молекул ДНК до нескольких сотен килобаз». Методы природы. 6 (5): 343–5. Дои:10.1038 / nmeth.1318. PMID 19363495. S2CID 1351008.
- ^ Цюань Дж., Тиан Дж. (Июль 2009 г.). «Клонирование с расширением круговой полимеразы сложных библиотек генов и путей». PLOS ONE. 4 (7): e6441. Bibcode:2009PLoSO ... 4.6441Q. Дои:10.1371 / journal.pone.0006441. ЧВК 2713398. PMID 19649325.
- ^ Ли М.З., Элледж С.Дж. (март 2007 г.). «Использование гомологичной рекомбинации in vitro для создания рекомбинантной ДНК с помощью SLIC». Методы природы. 4 (3): 251–6. Дои:10.1038 / nmeth1010. PMID 17293868. S2CID 30893882.
- ^ Чжан Ю., Верлинг Ю., Эдельманн В. (апрель 2012 г.). «SLiCE: новый метод клонирования ДНК на основе экстракта бактериальных клеток». Исследования нуклеиновых кислот. 40 (8): e55. Дои:10.1093 / нар / gkr1288. ЧВК 3333860. PMID 22241772.
- ^ «Как Gibson Assembly® меняет синтетическую биологию». Биолаборатории Новой Англии. Получено 2020-04-14.
- ^ Казини А., Макдональд Дж. Т., Де Йонге Дж., Христодулу Дж., Фримонт П. С., Болдуин Г. С., Эллис Т. (январь 2014 г.). «Конструирование ДНК в одном горшке для синтетической биологии: модульная сборка, направленная на перекрытие, с линкерами (MODAL)». Исследования нуклеиновых кислот. 42 (1): e7. Дои:10.1093 / nar / gkt915. ЧВК 3874208. PMID 24153110.
- ^ а б c d Сторч М., Казини А., Макроу Б., Флеминг Т., Трюитт Х., Эллис Т., Болдуин Г.С. (июль 2015 г.). «BASIC: новый стандарт сборки биодеталей для идемпотентного клонирования, обеспечивающий точную сборку одноуровневой ДНК для синтетической биологии». Синтетическая биология ACS. 4 (7): 781–7. Дои:10.1021 / sb500356d. PMID 25746445.
- ^ "Протокол сборки Гибсона". Addgene. Получено 2020-04-14.
- ^ Эль Каруи М., Ойос-Флайт М., Флетчер Л. (2019). «Будущие тенденции в синтетической биологии - отчет». Границы биоинженерии и биотехнологии. 7: 175. Дои:10.3389 / fbioe.2019.00175. ЧВК 6692427. PMID 31448268.
- ^ Косури С., Church GM (май 2014 г.). «Масштабный синтез ДНК de novo: технологии и приложения» (PDF). Методы природы. 11 (5): 499–507. Дои:10.1038 / nmeth.2918. ЧВК 7098426. PMID 24781323.
- ^ Лартиг С., Гласс Дж. И., Альперович Н., Пипер Р., Пармар П. П., Хатчисон С. А. и др. (Август 2007 г.). «Трансплантация генома бактерий: смена одного вида на другой». Наука. 317 (5838): 632–8. Bibcode:2007Научный ... 317..632L. CiteSeerX 10.1.1.395.4374. Дои:10.1126 / science.1144622. PMID 17600181. S2CID 83956478.
- ^ Пилкингтон Э (06.10.2009). «Я создаю искусственную жизнь, - заявляет генный пионер США». Лондон: Гардиан. В архиве из оригинала 28 мая 2010 г.. Получено 2010-05-22.
- ^ Пенниси, Э. (21 мая 2010 г.). «Синтетический геном приносит новую жизнь бактериям» (PDF). Наука. 328 (5981): 958–9. Дои:10.1126 / science.328.5981.958. PMID 20488994. В архиве (PDF) из оригинала 25 мая 2010 г.. Получено 2010-05-21.
- ^ «Как ученые создали искусственную жизнь'". Новости BBC. 2010-05-20. В архиве с оригинала от 1 июня 2013 г.. Получено 2010-05-21.
- ^ «Дрожжи 2.0». Коллекция Nature Communications. Springer Nature Limited. Получено 2020-04-17.
- ^ Шукман Д. (27 марта 2014 г.). «Ученые приветствуют развитие синтетических хромосом». Новости BBC. Получено 2014-03-28.
- ^ Annaluru N, Muller H, Mitchell LA, Ramalingam S, Stracquadanio G, Richardson SM и др. (Апрель 2014 г.). «Полный синтез функционального конструктора эукариотической хромосомы». Наука. 344 (6179): 55–8. Bibcode:2014Наука ... 344 ... 55А. Дои:10.1126 / science.1249252. ЧВК 4033833. PMID 24674868.
- ^ Специальный выпуск СИНТЕТИЧЕСКИЙ ГЕНОМ ДРОЖЖЕЙ Наука 10 марта 2017 г., том 355, номер 6329