Реплисом - Replisome
Эта статья нужны дополнительные цитаты для проверка. (Июль 2018 г.) (Узнайте, как и когда удалить этот шаблон сообщения) |

В ответственный это сложный молекулярная машина который выполняет репликация из ДНК. Реплисома сначала раскручивает двухцепочечную ДНК на две одноцепочечные. Для каждой из полученных одиночных прядей новый дополнительный синтезируется последовательность ДНК. Конечный результат - образование двух новых последовательностей двухцепочечной ДНК, которые являются точными копиями исходной последовательности двухцепочечной ДНК.[1]
По структуре реплисома состоит из двух репликативных полимераза комплексы, один из которых синтезирует ведущая нить, а другой синтезирует отстающая прядь. Реплисома состоит из ряда белки включая геликаза, RFC, PCNA, гираза /топоизомераза, SSB /RPA, прима, ДНК-полимераза III, РНКаза H, и лигаза.
Обзор процесса репликации прокариотической ДНК
За прокариоты, каждое деление нуклеоид (область, содержащая генетический материал, не являющийся ядром) требует двух реплисом для двунаправленная репликация. Две реплисомы продолжают репликацию на обоих вилки в середине клетки. Наконец, когда сайт терминации реплицируется, две реплисомы отделяются от ДНК. Реплисома остается в фиксированном месте в середине клетки и прикрепляется к мембрана, и матричная ДНК пронизывает его. ДНК подается через неподвижную пару реплисом, расположенных на клеточной мембране.
Обзор процесса репликации эукариотической ДНК
За эукариоты, многочисленные пузыри репликации формируются в истоках репликации на протяжении всего хромосома. Как и в случае с прокариотами, требуются две реплисомы, по одной на каждой репликационной вилке, расположенной на конце репликационного пузыря. Из-за значительных различий в размерах хромосом и связанных с ними сложностей высококонденсированных хромосом различные аспекты процесса репликации ДНК у эукариот, включая терминальные фазы, менее хорошо изучены, чем у прокариот.
Проблемы репликации ДНК
Реплисома - это система, в которой различные факторы работают вместе, чтобы решить структурные и химические проблемы репликации ДНК. Размер и структура хромосом у разных организмов различны, но поскольку молекулы ДНК являются резервуаром генетической информации для всех форм жизни, многие проблемы репликации и решения одинаковы для разных организмов. В результате факторы репликации, которые решают эти проблемы, являются высоко консервативными с точки зрения структуры, химического состава, функциональности или последовательности. Общие структурные и химические проблемы включают следующее:
- Эффективная сборка реплисом в ориджинах репликации (комплексы распознавания ориджина или специфические последовательности ориджина репликации у некоторых организмов)
- Разделение дуплекса на ведущие и отстающие нити шаблона (геликасы )
- Защита ведущих и отстающих нитей от повреждений после разделения дуплекса (факторы SSB и RPA)
- Праймирование ведущей и отстающей цепей матрицы (праймаза или ДНК-полимераза альфа)
- Обеспечение процессивность (факторы нагрузки зажима, белки зажима кольцевой формы, белки связывания цепи)
- Высокоточная репликация ДНК (ДНК-полимераза III, ДНК-полимераза дельта, ДНК-полимераза эпсилон. Все они имеют низкую частоту ошибок из-за своей структуры и химического состава).
- Исправление ошибок (ошибки распознавания активных сайтов репликативной полимеразы; от 3 'до 5' экзонуклеаза домены репликативных полимераз исправляют ошибки)
- Синхронизированная полимеризация ведущих и отстающих цепей, несмотря на антипараллельную структуру (структура репликационной вилки, димеризация репликативных полимераз)
- Удаление праймера (ДНК-полимераза I, РНКаза H, лоскут эндонуклеазы Такие как FEN1 или другие факторы репарации ДНК)
- Образование фосфодиэфирных связей в зазорах между Фрагменты Окадзаки (лигаза)
В общем, проблемы репликации ДНК включают структуру молекул, химию молекул и, с точки зрения системы, лежащие в основе отношения между структурой и химией.
Решение проблем репликации ДНК
Многие структурные и химические проблемы, связанные с репликацией ДНК, решаются с помощью молекулярного механизма, который хорошо сохраняется у организмов. В этом разделе обсуждается, как реплисомные факторы решают структурные и химические проблемы репликации ДНК.
Реплисомная сборка
Репликация ДНК начинается в сайтах, называемых источниками репликации. У организмов с небольшими геномами и простой структурой хромосом, таких как бактерии, на каждой хромосоме может быть только несколько источников репликации. У организмов с большими геномами и сложной структурой хромосом, таких как люди, могут быть сотни или даже тысячи источников репликации, распределенных по множеству хромосом.
Структура ДНК изменяется во времени, пространстве и последовательности, и считается, что эти вариации, помимо их роли в экспрессии генов, также играют активную роль в сборке реплисом во время синтеза ДНК. Сборка реплисом в ориджине репликации примерно разделяется на три фазы.
Для прокариот:
- Формирование пререпликационного комплекса. DnaA связывается с комплекс распознавания происхождения и разделяет дуплекс. Это привлекает DnaB геликаза и DnaC, которые поддерживают пузырек репликации.
- Формирование предпускового комплекса. SSB связывается с одной нитью, а затем гамма (коэффициент нагрузки зажима) связывается с SSB.
- Формирование инициирующего комплекса. Гамма вносит скользящий зажим (бета) и привлекает ДНК-полимеразу III.
Для эукариот:
- Формирование пререпликационного комплекса. MCM факторы связаны с комплекс распознавания происхождения и разделите дуплекс, образуя репликационный пузырь.
- Формирование предпускового комплекса. Репликационный белок А (RPA) связывается с одноцепочечной ДНК, а затем RFC (фактор нагрузки зажима) связывается с RPA.
- Формирование инициирующего комплекса. RFC размещает скользящий зажим (PCNA ) и привлекает ДНК-полимеразы, такие как альфа (α), дельта (δ), эпсилон (ε).
И для прокариот, и для эукариот следующую стадию обычно называют «удлинением», и именно на этой стадии происходит большая часть синтеза ДНК.
Разделение дуплекса
ДНК представляет собой дуплекс, образованный двумя антипараллельными цепями. Следующий Meselson-Stahl, процесс репликации ДНК является полуконсервативным, посредством чего во время репликации исходный дуплекс ДНК разделяется на две дочерние цепи (называемые матрицами ведущей и отстающей). Каждая дочерняя цепь становится частью нового дуплекса ДНК. Факторы, обычно называемые геликасами, раскручивают дуплекс.
Геликасы
Геликаза представляет собой фермент, который разрывает водородные связи между парами оснований в середине дуплекса ДНК. Его структура, похожая на пончик, обвивает ДНК и разделяет нити перед синтезом ДНК. У эукариот комплекс Mcm2-7 действует как геликаза, хотя не совсем ясно, какие субъединицы необходимы для активности геликазы.[2] Эта геликаза перемещается в том же направлении, что и ДНК-полимераза (от 3 'до 5' относительно цепи-матрицы). У прокариотических организмов геликазы лучше идентифицируются и включают dnaB, который перемещается с 5 'на 3' на нити, противоположной ДНК-полимеразе.
Размотка суперспиралей и декатенация
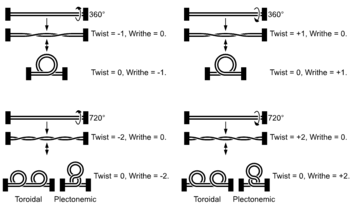
Когда геликаза раскручивает двойную спираль, топологические изменения, вызванные вращательным движением геликазы, приводят к образованию суперспирали перед геликазой (аналогично тому, что происходит, когда вы скручиваете кусок нити).
Гираза и топоизомеразы
Gyrase (форма топоизомераза ) расслабляет и устраняет суперспирализацию, вызванную геликазой. Он делает это, разрезая нити ДНК, позволяя ей вращаться и высвобождая суперспираль, а затем снова соединяя нити. Гираза чаще всего находится перед вилкой репликации, где образуются суперспирали.
Защита ведущих и отстающих прядей

Одноцепочечная ДНК очень нестабильна и может образовывать водородные связи с собой, которые называются «шпильками» (или одна цепь может неправильно связываться с другой одной цепью). Чтобы противодействовать этой нестабильности, одноцепочечные связывающие белки (SSB у прокариот и Репликационный белок А у эукариот) связываются с открытыми основаниями, чтобы предотвратить неправильное лигирование.
Если рассматривать каждую прядь как «динамическую, эластичную струну», структурный потенциал неправильного лигирования должен быть очевиден.
| Отстающая нить без связывающих белков. |
|---|
|
Расширенная схема раскрывает основную химию проблемы: возможность образования водородной связи между неродственными парами оснований.
| Схематическое изображение недавно разделенных цепей ДНК без белков, связывающих цепь. |
|---|
|
Связывающие белки стабилизируют одиночную нить и защищают нить от повреждений, вызванных нелицензированными химическими реакциями.
| Отстающая нить покрыта связывающими белками (*), предотвращающими неправильное лигирование. |
|---|
|
Комбинация одной цепи и ее связывающих белков служит лучшим субстратом для репликативных полимераз, чем голая одиночная цепь (связывающие белки обеспечивают дополнительную термодинамическую движущую силу для реакции полимеризации). Белки, связывающие нити, удаляются репликативными полимеразами.
Грунтование ведущих и отстающих прядей
Как со структурной, так и с химической точки зрения, отдельная цепь ДНК сама по себе (и связанные с ней связывающие белки с одной цепью) не подходят для полимеризации. Это связано с тем, что химические реакции, катализируемые репликативными полимеразами, требуют свободного 3 'ОН, чтобы инициировать удлинение нуклеотидной цепи. С точки зрения структуры, конформация активных сайтов репликативной полимеразы (которая в значительной степени связана с присущей репликативной полимераз точностью) означает, что эти факторы не могут начать удлинение цепи без ранее существовавшей цепи нуклеотидов, потому что никакая известная репликативная полимераза не может начать удлинение цепи. de novo.
Праймирующие ферменты (ДНК-зависимые РНК-полимеразы ), решить эту проблему путем создания праймера РНК на ведущей и отстающей цепях. Ведущую цепь праймируют один раз, а отстающей цепи праймируют примерно через каждые 1000 (+/- 200) пар оснований (один праймер для каждого фрагмента Окадзаки на отстающей цепи). Каждый праймер РНК имеет длину примерно 10 оснований.
| Однонить ДНК с белками, связывающими нить (*), и праймером РНК, добавленными примирующими ферментами (UAGCUAUAUAUA). |
|---|
|
Интерфейс в точке (A *) содержит свободный 3 'ОН, который химически подходит для реакции, катализируемой репликативными полимеразами, а конфигурация «выступа» структурно подходит для удлинения цепи репликативной полимеразой. Таким образом, репликативные полимеразы могут начинать удлинение цепи в точке (A *).
Primase
У прокариот прима создает праймер РНК в начале недавно разделенных ведущей и отстающей цепей.
ДНК-полимераза альфа
У эукариот ДНК-полимераза альфа создает праймер РНК в начале недавно разделенных ведущей и отстающей цепей, и, в отличие от примазы, ДНК-полимераза альфа также синтезирует короткую цепь дезоксинуклеотидов после создания праймера.
Обеспечение процессивности и синхронизации
Процессивность относится как к скорости, так и к непрерывности репликации ДНК, а высокая процессивность является требованием для своевременной репликации. Высокая процессивность частично обеспечивается кольцевыми белками, называемыми «зажимами», которые помогают репликативным полимеразам оставаться связанными с ведущими и отстающими цепями. Есть и другие переменные: с химической точки зрения белки, связывающие цепи, стимулируют полимеризацию и обеспечивают дополнительную термодинамическую энергию для реакции. С системной точки зрения структура и химический состав многих факторов реплисомы (таких как особенности ААА + АТФазы отдельных субъединиц нагрузки зажима вместе со спиральной конформацией, которую они принимают), а также связи между факторами нагрузки зажима и другими дополнительными факторами, также увеличивает процессивность.
К этому моменту, согласно исследованиям Курияна и др.,[3] из-за своей роли в рекрутировании и связывании других факторов, таких как прайм-ферменты и репликативные полимеразы, зажимы-загрузчики и скользящие зажимы лежат в основе механизма реплисом. Исследования показали, что нагрузка зажима и факторы скользящего зажима абсолютно необходимы для воспроизведения, что объясняет высокую степень структурной консервации, наблюдаемую для нагрузки зажима и факторов скользящего зажима. Эта архитектурная и структурная консервация наблюдается у таких разнообразных организмов, как бактерии, фаги, дрожжи и люди. То, что такая значительная степень структурной консервативности наблюдается без гомологии последовательностей, еще больше подчеркивает важность этих структурных решений для проблем репликации.
Зажимный погрузчик
Загрузчик зажима - это общий термин, который относится к факторам репликации, называемым гамма (прокариоты) или RFC (эукариоты). Комбинация матричной ДНК и праймерной РНК обозначается как «А-форма ДНК 'и считается, что белки репликации с зажимной загрузкой (спиральные гетеропентамеры) хотят связываться с ДНК А-формы из-за ее формы (структура большой / малой бороздки) и химического состава (паттерны водородная связь доноры и акцепторы).[3][4] Таким образом, загрузочные белки зажима связываются с праймированной областью цепи, что вызывает гидролиз АТФ и обеспечивает энергию для открытия зажима и прикрепления его к цепи.[3][4]
Скользящий зажим

Скользящий зажим - это общий термин, который относится к кольцевым факторам репликации, называемым бета (прокариоты) или PCNA (эукариоты). Зажимные белки привлекают и связывают репликативные полимеразы, такие как ДНК-полимераза III, для увеличения количества времени, в течение которого репликативная полимераза остается связанной с цепью. С химической точки зрения, зажим имеет слегка положительный заряд в центре, который почти идеально соответствует слегка отрицательному заряду цепи ДНК.
У некоторых организмов зажим представляет собой димер, а у других организмов зажим представляет собой тример. Несмотря на это, консервативная кольцевая архитектура позволяет зажиму закрывать прядь.
Димеризация репликативных полимераз
Репликативные полимеразы образуют асимметричный димер на вилке репликации, связываясь с субъединицами фактора загрузки зажима. Эта асимметричная конформация способна одновременно реплицировать ведущую и отстающую нити, и совокупность факторов, которая включает репликативные полимеразы, обычно называется холоэнзим. Однако остаются серьезные проблемы: ведущие и отстающие нити антипараллельны. Это означает, что синтез нуклеотидов на ведущей цепи естественным образом происходит в направлении от 5 'к 3'. Однако отстающая цепь проходит в противоположном направлении, и это представляет собой серьезную проблему, поскольку никакие известные репликативные полимеразы не могут синтезировать ДНК в направлении от 3 'до 5'.
Димеризация репликативных полимераз решает проблемы, связанные с эффективной синхронизацией синтеза ведущей и отстающей цепи на репликационной вилке, но тесное пространственно-структурное связывание репликативных полимераз, решая сложную проблему синхронизации, создает еще одну проблему: димеризация Репликативные полимеразы на репликационной вилке означают, что синтез нуклеотидов для обеих цепей должен происходить в одном и том же пространственном положении, несмотря на тот факт, что отстающая цепь должна синтезироваться в обратном направлении относительно ведущей цепи. Синтез отстающей цепи происходит после того, как геликаза размотала достаточное количество отстающей цепи, и это «достаточное количество отстающей цепи» полимеризуется в дискретных нуклеотидных цепях, называемых фрагментами Окадзаки.
Учтите следующее: хеликаза непрерывно раскручивает родительский дуплекс, но отстающая нить должна полимеризоваться в противоположном направлении. Это означает, что, в то время как полимеризация ведущей цепи продолжается, полимеризация отстающей цепи происходит только после того, как достаточная часть отстающей цепи будет размотана геликазой. На этом этапе репликативная полимераза отстающей цепи связывается с зажимом и праймером, чтобы начать полимеризацию. Во время синтеза отстающей цепи репликативная полимераза отправляет отстающую цепь обратно к репликационной вилке. Репликативная полимераза диссоциирует, когда достигает праймера РНК. Геликаза продолжает раскручивать родительский дуплекс, праймирующий фермент прикрепляет другой праймер, и репликативная полимераза повторно связывается с зажимом и праймером, когда разматывается достаточное количество отстающей цепи.
В совокупности синтез ведущей и отстающей нити называется «полунепрерывным».
Репликация ДНК с высокой точностью
Прокариотические и эукариотические организмы используют множество репликативных полимераз, некоторые из которых хорошо охарактеризованы:
- ДНК-полимераза III
- ДНК-полимераза дельта
- ДНК-полимераза эпсилон
ДНК-полимераза III
Эта полимераза синтезирует ведущую и отстающую цепь ДНК у прокариот.
ДНК-полимераза дельта
Эта полимераза синтезирует запаздывающую цепь ДНК у эукариот.[5] (Предполагается, что с ДНК-полимеразой эпсилон образуется асимметричный димер.)[6]
ДНК-полимераза эпсилон
Эта полимераза синтезирует ведущую цепь ДНК у эукариот.[7] (Предполагается, что с дельта ДНК-полимеразой образуется асимметричный димер.)[5]
Корректура и исправление ошибок
Хотя и редко, но полимеризация с неправильным спариванием оснований все же происходит во время удлинения цепи. (Структура и химический состав репликативных полимераз означают, что ошибки маловероятны, но они все же возникают.) Многие репликативные полимеразы содержат механизм «исправления ошибок» в виде экзонуклеазного домена от 3 'до 5', который способен удалять пары оснований из открытый 3 'конец растущей цепочки. Коррекция ошибок возможна, потому что ошибки пары оснований искажают положение ионов магния в субблоке полимеризации, а структурно-химическое искажение блока полимеризации эффективно тормозит процесс полимеризации, замедляя реакцию.[8] Впоследствии химическая реакция в экзонуклеазной единице берет верх и удаляет нуклеотиды с открытого 3'-конца растущей цепи.[9] После устранения ошибки структура и химический состав блока полимеризации возвращаются к норме, и репликация ДНК продолжается. Работая вместе таким образом, активный сайт полимеризации можно рассматривать как «корректор», поскольку он обнаруживает несоответствия, а экзонуклеаза является «редактором», поскольку он исправляет ошибки.
Ошибки пар оснований искажают активный сайт полимеразы на 4-6 нуклеотидов, что означает, что в зависимости от типа несоответствия существует до шести шансов на исправление ошибки.[8] Функции распознавания ошибок и исправления ошибок в сочетании с присущей им точностью, обусловленной структурой и химическим составом репликативных полимераз, вносят вклад в частоту ошибок, составляющую примерно 1 несоответствие пары оснований из 10.8 до 1010 пар оснований.
| Схематическое изображение правильных пар оснований, за которыми следуют 8 возможных несовпадений пар оснований.[10] |
|---|
|
Ошибки можно разделить на три категории: несовпадения пурин-пурин, несовпадения пиримидин-пиримидин и несовпадения пиримидин-пурин. Химический состав каждого несоответствия меняется, как и поведение репликативной полимеразы в отношении ее активности восприятия несоответствия.
Воспроизведение бактериофаг Т4 ДНК при заражении Кишечная палочка хорошо изученная система репликации ДНК. В период экспоненциального увеличения ДНК при 37 ° C скорость удлинения составляет 749 нуклеотидов в секунду.[11] В скорость мутации при репликации 1,7 мутации на 108 пар оснований.[12] Таким образом, репликация ДНК в этой системе происходит очень быстро и с высокой точностью.
Удаление праймера и лигирование порезов
После синтеза ведущей и отстающей цепи возникают две проблемы: РНК остается в дуплексе, и между каждым фрагментом Окадзаки в отстающем дуплексе есть зазоры. Эти проблемы решаются с помощью различных ферментов репарации ДНК, которые различаются в зависимости от организма, включая ДНК-полимеразу I, ДНК-полимеразу бета, РНКазу H, лигазу и ДНК2. Этот процесс хорошо охарактеризован у прокариот и гораздо менее хорошо охарактеризован у многих эукариот.
В общем, ферменты репарации ДНК дополняют фрагменты Окадзаки различными способами, включая: вырезание пар оснований и активность экзонуклеазы от 5 'до 3', которая удаляет химически нестабильные рибонуклеотиды из отстающего дуплекса и заменяет их стабильными дезоксинуклеотидами. Этот процесс называется «созреванием фрагментов Окадзаки», и лигаза (см. Ниже) завершает последний этап процесса созревания.
| Дуплекс РНК-ДНК с рибонуклеотидами, добавленными праймирующим ферментом (-), и дезоксинуклеотидами, добавленными репликативной полимеразой (+). |
|---|
|
Удаление праймера и лигирование разрывов можно рассматривать как процессы репарации ДНК, в результате которых образуется химически стабильный, безошибочный дуплекс. К этому моменту, что касается химии дуплекса РНК-ДНК, в дополнение к присутствию урацила в дуплексе, присутствие рибозы (которая имеет реактивный 2'-ОН) имеет тенденцию делать дуплекс гораздо менее химически стабильным. чем дуплекс, содержащий только дезоксирибозу (которая имеет нереактивный 2 'H).
ДНК-полимераза I
ДНК-полимераза I - это фермент, восстанавливающий ДНК.
РНКаза H
РНКаза H - это фермент, который удаляет РНК из дуплекса РНК-ДНК.
Лигаза
После того, как факторы репарации ДНК заменяют рибонуклеотиды праймера дезоксинуклеотидами, в сахарофосфатном скелете остается один разрыв между каждым фрагментом Окадзаки в отстающем дуплексе. Фермент под названием ДНК-лигаза соединяет разрыв в основной цепи, образуя фосфодиэфирную связь между каждым разрывом, который разделяет фрагменты Окадзаки. Структурные и химические аспекты этого процесса, обычно называемого «ник-переводом», выходят за рамки данной статьи.
| Схематическое изображение нового дуплекса дочерней ДНК с отстающей цепью показано ниже вместе с сахарно-фосфатным остовом. |
|---|
|
| Готовый дуплекс: |
|---|
|
Репликационный стресс
Репликационный стресс может привести к остановке репликационной вилки. Один тип репликативного стресса возникает в результате повреждения ДНК, например: межнитевые поперечные связи (ICL). ICL может блокировать развитие репликативной вилки из-за нарушения разделения цепей ДНК. В клетках позвоночных репликация ICL-содержащих хроматин шаблон запускает набор более 90 Ремонт ДНК и геном Факторы обслуживания.[13] Эти факторы включают белки, которые выполняют последовательные разрезы и гомологичная рекомбинация.
История
Кэтрин Лемон и Алан Гроссман показали, как Bacillus subtilis что реплисомы не движутся, как поезда по рельсам, но ДНК фактически проходит через неподвижную пару реплисом, расположенных на клеточной мембране. В своем эксперименте реплисомы в Б. subtilis каждый был помечен зеленым флуоресцентным белком, и местоположение комплекса отслеживали в реплицирующихся клетках с использованием флуоресцентная микроскопия. Если бы реплисомы двигались, как поезд по рельсам, белок полимераза-GFP находился бы в разных положениях в каждой клетке. Вместо этого, однако, в каждой реплицирующейся клетке реплисомы наблюдались как отдельные флуоресцентные фокусы, расположенные в средней клетке или рядом с ней. Клеточная ДНК, окрашенная синим флуоресцентным красителем (DAPI), явно занимала большую часть цитоплазматического пространства.[14]
Рекомендации
- ^ Yao, Nina Y .; О'Доннелл, Майк (2010). "SnapShot: Replisome". Клетка. Elsevier BV. 141 (6): 1088–1088.e1. Дои:10.1016 / j.cell.2010.05.042. ISSN 0092-8674. ЧВК 4007198. PMID 20550941.
- ^ Бохман М.Л., Швача А. (июль 2008 г.). «Комплекс Mcm2-7 обладает геликазной активностью in vitro». Мол. Клетка. 31 (2): 287–93. Дои:10.1016 / j.molcel.2008.05.020. PMID 18657510.
- ^ а б c Келч Б.А., Макино Д.Л., О'Доннелл М., Куриян Дж. (2012). «АТФазы с зажимом-загрузчиком и эволюция механизма репликации ДНК». BMC Biol. 10: 34. Дои:10.1186/1741-7007-10-34. ЧВК 3331839. PMID 22520345.
- ^ а б Боумен Г.Д., О'Доннелл М., Курьян Дж. (Июнь 2004 г.). «Структурный анализ эукариотического скользящего загрузочного комплекса зажим-зажим ДНК». Природа. 429 (6993): 724–30. Дои:10.1038 / природа02585. PMID 15201901.
- ^ а б Swan MK, Johnson RE, Prakash L, Prakash S, Aggarwal AK (сентябрь 2009 г.). «Структурные основы высокоточного синтеза ДНК дрожжевой ДНК-полимеразой дельта». Nat. Struct. Мол. Биол. 16 (9): 979–86. Дои:10.1038 / nsmb.1663. ЧВК 3055789. PMID 19718023.
- ^ Миябе И., Кункель Т.А., Карр А.М. (декабрь 2011 г.). «Основные роли ДНК-полимераз эпсилон и дельта в эукариотической репликационной вилке сохраняются эволюционно». PLOS Genet. 7 (12): e1002407. Дои:10.1371 / journal.pgen.1002407. ЧВК 3228825. PMID 22144917.
- ^ Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (июль 2007 г.). «Дрожжевая ДНК-полимераза эпсилон участвует в репликации ведущей цепи ДНК». Наука. 317 (5834): 127–30. Дои:10.1126 / science.1144067. ЧВК 2233713. PMID 17615360.
- ^ а б Джонсон С.Дж., Биз Л.С. (март 2004 г.). «Структуры ошибок несовпадения репликации, наблюдаемые в ДНК-полимеразе». Клетка. 116 (6): 803–16. Дои:10.1016 / S0092-8674 (04) 00252-1. PMID 15035983.
- ^ Jiricny J (март 2004 г.). «Неверная ДНК-полимераза поймана с поличным» (PDF). Мол. Клетка. 13 (6): 768–9. Дои:10.1016 / S1097-2765 (04) 00149-2. PMID 15053870.
- ^ «Мутагенез и репарация ДНК». ATDBio Ltd.
- ^ Маккарти Д., Миннер С., Бернштейн Х, Бернштейн С. (1976). «Скорость удлинения ДНК и распределение точек роста фага Т4 дикого типа и янтарного мутанта с задержкой ДНК». J. Mol. Биол. 106 (4): 963–81. Дои:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Дрейк Дж. В. (1970) Молекулярная основа мутации. Холден-Дэй, Сан-Франциско ISBN 0816224501 ISBN 978-0816224500
- ^ Räschle M, Smeenk G, Hansen RK, Temu T, Oka Y, Hein MY, Nagaraj N, Long DT, Walter JC, Hofmann K, Storchova Z, Cox J, Bekker-Jensen S, Mailand N, Mann M (2015). «Ремонт ДНК. Протеомика выявляет динамическую сборку репарационных комплексов во время обхода перекрестных связей ДНК». Наука. 348 (6234): 1253671. Дои:10.1126 / science.1253671. ЧВК 5331883. PMID 25931565.
- ^ Фостер JB, Slonczewski J (2010). Микробиология: развивающаяся наука (Второе изд.). Нью-Йорк: W. W. Norton & Company. ISBN 978-0-393-93447-2.
дальнейшее чтение
- Померанц RT, О'Доннелл М (апрель 2007 г.). «Реплисомная механика: взгляд на двойную ДНК-полимеразную машину». Тенденции Microbiol. 15 (4): 156–64. Дои:10.1016 / j.tim.2007.02.007. PMID 17350265.
внешняя ссылка
- ДНК + реплисома в Национальной медицинской библиотеке США Рубрики медицинской тематики (MeSH)
