Репликация эукариотической ДНК - Eukaryotic DNA replication - Wikipedia
Репликация эукариотической ДНК консервативный механизм, ограничивающий Репликация ДНК до одного раза за клеточный цикл. Репликация ДНК эукариот хромосомный ДНК имеет центральное значение для дублирования клетка и необходим для поддержания эукариотической геном.
Репликация ДНК - это действие ДНК-полимеразы синтез цепи ДНК, комплементарной исходной цепи матрицы. Чтобы синтезировать ДНК, двухцепочечная ДНК разматывается ДНК геликасы впереди полимераз, образуя вилку репликации, содержащую два однонитевых шаблона. Процессы репликации позволяют копировать одну двойную спираль ДНК в две спирали ДНК, которые делятся на дочерние клетки на митоз. Основные ферментативные функции, выполняемые в репликационной вилке, хорошо сохраняются от прокариоты к эукариоты, но репликационный аппарат в репликации эукариотической ДНК представляет собой гораздо более крупный комплекс, координирующий многие белки в месте репликации, формируя ответственный.[1]
Реплисома отвечает за копирование всего геномный ДНК в каждой пролиферативной клетке. Этот процесс позволяет с высокой точностью передавать наследственную / генетическую информацию от родительской клетки к дочерней клетке и, таким образом, необходим для всех организмов. Большая часть клеточный цикл построен на обеспечении того, чтобы репликация ДНК происходила без ошибок.[1]
В грамм1 фаза клеточного цикла запускаются многие процессы регуляции репликации ДНК. У эукариот подавляющее большинство Синтез ДНК происходит во время S фаза клеточного цикла, и весь геном должен быть размотан и продублирован, чтобы сформировать две дочерние копии. В течение грамм2, любые поврежденные ДНК или ошибки репликации исправляются. Наконец, одна копия генома отделяется от каждой дочерней клетки в митозе или M-фазе.[2] Каждая из этих дочерних копий содержит одну цепь родительской дуплексной ДНК и одну возникающую антипараллельную цепь.
Этот механизм сохраняется от прокариот до эукариот и известен как полуконсервативный Репликация ДНК. Процесс полуконсервативной репликации для сайта репликации ДНК представляет собой вилкообразную структуру ДНК, вилку репликации, где спираль ДНК открывается или раскручивается, обнажая неспаренную ДНК. нуклеотиды для распознавания и спаривания оснований для включения свободных нуклеотидов в двухцепочечную ДНК.[3]
Посвящение
Инициирование репликации эукариотической ДНК - это первая стадия синтеза ДНК, на которой двойная спираль ДНК разматывается и на ведущей цепи происходит начальное событие праймирования ДНК-полимеразой α. Событие прайминга на отстающей нити устанавливает вилку репликации. Праймирование спирали ДНК заключается в синтезе праймера РНК, позволяющего синтез ДНК ДНК-полимеразой α. Праймирование происходит один раз в начале на ведущей нити и в начале каждого фрагмента Окадзаки на отстающей нити.
Репликация ДНК начинается с определенных последовательностей, называемых истоки репликации, а эукариотические клетки имеют несколько источников репликации. Чтобы инициировать репликацию ДНК, несколько репликативных белков собираются и отделяются от этих репликативных источников.[4] Отдельные факторы, описанные ниже, работают вместе, чтобы направлять формирование пререпликационный комплекс (pre-RC), ключевое промежуточное звено в процессе инициации репликации.
Ассоциация комплекс распознавания происхождения (ORC) с источником репликации набирает белок 6 цикла деления клетки (Cdc6), чтобы сформировать платформу для загрузки комплекс поддержания минихромосом (Mcm 2-7) белки, чему способствует лицензирование хроматина и белок фактора репликации ДНК 1 (Cdt1). ORC, Cdc6 и Cdt1 вместе необходимы для стабильной ассоциации комплекса Mcm2-7 с репликативными ориджинами во время G1 фаза клеточного цикла.[5]
Пререпликативный комплекс
Эукариотические источники репликации контролируют образование ряда белковых комплексов, которые приводят к сборке двух двунаправленных вилок репликации ДНК. Эти события инициированы формированием пререпликационный комплекс (pre-RC) в истоках репликации. Этот процесс происходит в G1 стадия клеточного цикла. Формирование пре-RC включает упорядоченную сборку многих факторов репликации, включая комплекс распознавания ориджина (ORC), белок Cdc6, белок Cdt1 и поддерживающие белки минихромосомы (Mcm2-7).[6][7] Как только пре-RC образуется, активация комплекса запускается двумя киназы, циклин-зависимая киназа 2 (CDK) и Dbf4-зависимая киназа (DDK), которые помогают преобразовать пре-RC в комплекс инициации до инициации репликации ДНК. Этот переход включает упорядоченную сборку дополнительных факторов репликации для раскручивания ДНК и накопления множественных полимераз эукариотической ДНК вокруг раскрученной ДНК. Центральным в вопросе о том, как двунаправленные репликационные вилки устанавливаются в ориджинах репликации, является механизм, с помощью которого ORC рекрутирует два встречных комплекса Mcm2-7 в каждую точку начала репликации, чтобы сформировать пререпликационный комплекс.[8][9][10]
Комплекс распознавания происхождения
Первым шагом в сборке пререпликационного комплекса (pre-RC) является связывание комплекс распознавания происхождения (ORC) к источнику репликации. В позднем митозе белок Cdc6 присоединяется к связанному ORC с последующим связыванием комплекса Cdt1-Mcm2-7.[11] ORC, Cdc6 и Cdt1 все необходимы для загрузки комплекса из шести белков минихромосомы (Mcm 2-7) на ДНК. ORC представляет собой шести-субъединичный белковый комплекс Orc1p-6, который выбирает сайты репликации на ДНК для инициации репликации, а связывание ORC с хроматином регулируется через клеточный цикл.[6][12] Как правило, функция и размер субъединиц ORC сохраняются во многих геномах эукариот, с той разницей, что их сайты связывания ДНК расходятся.
Наиболее изученным комплексом распознавания происхождения является комплекс Saccharomyces cerevisiae или дрожжи, которые, как известно, связываются с автономно реплицирующаяся последовательность (ARS).[13] В С. cerevisiae ORC специфически взаимодействует как с элементами A, так и с B1 дрожжевых источников репликации, охватывая область 30 пар оснований.[14] Связывание с этими последовательностями требует АТФ.[6][14]
Атомная структура С. cerevisiae Был определен ORC, связанный с ДНК ARS.[14] Orc1, Orc2, Orc3, Orc4 и Orc5 окружают элемент A посредством двух типов взаимодействий, неспецифичных по основанию и специфичных по основанию, которые изгибают ДНК в элементе A. Все пять субъединиц контактируют с сахарно-фосфатным остовом в нескольких точках элемента A, образуя плотный захват без специфичности основания. Orc1 и Orc2 контактируют с малой бороздкой элемента A, в то время как домен крылатой спирали Orc4 контактирует с метильными группами инварианта Ts в большой бороздке элемента A через спираль вставки (IH). Отсутствие этого ИГ у многоклеточных животных.[14] частично объясняет отсутствие специфичности последовательности в ORC человека.[15] ДНК ARS также изгибается в элементе B1 за счет взаимодействия с Orc2, Orc5 и Orc6.[14] Изгибание исходной ДНК с помощью ORC, по-видимому, эволюционно консервативно, предполагая, что это может быть необходимо для механизма загрузки комплекса Mcm2-7.[14][16]
Когда ORC связывается с ДНК в точках начала репликации, он служит каркасом для сборки других ключевых факторов инициации пререпликативного комплекса.[17] Эта пререпликативная сложная сборка во время G1 стадия клеточного цикла требуется до активации репликации ДНК во время S-фазы.[18] Удаление хотя бы части комплекса (Orc1) из хромосомы на метафаза является частью регуляции ORC млекопитающих, чтобы гарантировать, что образование пререпликативного комплекса до завершения метафазы устранено.[19]
Cdc6 белок
Связывание цикл деления клеток 6 (Cdc6) к комплексу распознавания ориджина (ORC) является важным этапом сборки пре-репликационного комплекса (pre-RC) в ориджинах репликации. Cdc6 связывается с ORC на ДНК АТФ-зависимым образом, что вызывает изменение паттерна связывания ориджина, что требует Orc1 АТФаза.[20] Cdc6 требует ORC для связывания с хроматином и, в свою очередь, необходим для гептамера Cdt1-Mcm2-7.[11] связываться с хроматином.[21] Комплекс ORC-Cdc6 образует кольцевую структуру и аналогичен другим АТФ-зависимым белковым машинам. Уровни и активность Cdc6 регулируют частоту, с которой источники репликации используются в течение клеточного цикла.
Cdt1 белок
В лицензирование хроматина и фактор репликации ДНК 1 (Cdt1) белок необходим для лицензирования хроматина для репликации ДНК.[22][23] В С. cerevisiae, Cdt1 способствует загрузке комплекса Mcm2-7 по одному на хромосому, стабилизируя левостороннюю структуру с открытым кольцом одиночного гексамера Mcm2-7.[11][24][25] Cdt1 ассоциируется с Конечная точка C Cdc6, чтобы совместно способствовать ассоциации белков Mcm с хроматином.[26] Крио-ЭМ структура комплекса OCCM (ORC-Cdc6-Cdt1-MCM) показывает, что Cdt1-CTD взаимодействует с Mcm6-WHD.[27] У многоклеточных животных активность Cdt1 во время клеточного цикла строго регулируется его ассоциацией с белком близнецы, который ингибирует активность Cdt1 во время фазы S, чтобы предотвратить повторную репликацию ДНК и предотвращает ее убиквитинирование и последующие протеолиз.[28]
Белковый комплекс для поддержания минихромосом
Белки поддержания минихромосомы (Mcm) были названы в честь генетического скрининга мутантов инициации репликации ДНК в С. cerevisiae которые влияют на стабильность плазмид специфическим для ARS образом.[29] Mcm2, Mcm3, Mcm4, Mcm5, Mcm6 и Mcm7 образуют гексамерный комплекс, который имеет структуру с разомкнутым кольцом с зазором между Mcm2 и Mcm5.[11] Сборка белков Mcm на хроматин требует скоординированной функции комплекса распознавания источника (ORC), Cdc6 и Cdt1.[30] Как только белки Mcm загружены в хроматин, ORC и Cdc6 могут быть удалены из хроматина без предотвращения последующей репликации ДНК. Это наблюдение предполагает, что основная роль пререпликационного комплекса заключается в правильной загрузке белков Mcm.[31]

Белки Mcm на хроматине образуют двойной гексамер «голова к голове» с двумя кольцами, слегка наклоненными, скрученными и смещенными по центру, чтобы создать перегиб в центральном канале, где связанная ДНК захватывается на границе двух колец.[32][33] Каждое гексамерное кольцо Mcm2-7 сначала служит каркасом для сборки реплисомы, а затем ядром каталитической геликазы CMG (Cdc45-MCM-GINS), которая является основным компонентом реплисомы. Каждый белок Mcm тесно связан со всеми другими, но уникальные последовательности, отличающие каждый из типов субъединиц, сохраняются у эукариот. Все эукариоты имеют ровно шесть аналогов белка Mcm, каждый из которых попадает в один из существующих классов (Mcm2-7), что указывает на то, что каждый белок Mcm выполняет уникальную и важную функцию.[34][9]
Белки поддержания минихромосом необходимы для активности ДНК-геликазы. Инактивация любого из шести белков Mcm во время S-фазы предотвращает дальнейшее развитие репликационной вилки, предполагая, что геликаза не может рециклироваться и должна быть собрана в точках начала репликации.[35] Наряду с активностью геликазы комплекса поддерживающего белка минихромосомы, комплекс также имеет связанную с ним активность АТФазы.[36] Мутация в любом из шести белков Mcm снижает консервативные сайты связывания АТФ, что указывает на то, что гидролиз АТФ является скоординированным событием, в котором участвуют все шесть субъединиц комплекса Mcm.[37] Исследования показали, что внутри белкового комплекса Mcm находятся определенные каталитические пары белков Mcm, которые действуют вместе для координации гидролиза АТФ. Например, Mcm3 но нет Mcm6 может активировать активность Mcm6. Эти исследования, подтвержденные крио-ЭМ-структурами комплексов Mcm2-7,[11][32] предполагают, что комплекс Mcm представляет собой гексамер с Mcm3 следующий на Mcm7, Mcm2 следующий на Mcm6, и Mcm4 следующий на Mcm5. Оба члена каталитической пары вносят свой вклад в конформацию, которая позволяет связывать и гидролизу АТФ, а смесь активных и неактивных субъединиц создает скоординированную активность АТФазы, которая позволяет белковому комплексу Mcm завершить связывание и гидролиз АТФ в целом.[38]
В ядерная локализация поддерживающих белков минихромосом регулируется в почкующихся дрожжевых клетках.[39][40] Белки Mcm присутствуют в ядро в G1 стадии и фазы S клеточного цикла, но экспортируются в цитоплазма во время G2 этап и фаза М. Полный и интактный комплекс Mcm из шести субъединиц необходим для проникновения в ядро клетки.[41] В С. cerevisiae, ядерный экспорт способствует активности циклин-зависимой киназы (CDK). Белки Mcm, которые связаны с хроматином, защищены от механизма экспорта CDK из-за отсутствия доступа к CDK.[42]
Комплекс инициации
Во время G1 На стадии клеточного цикла факторы инициации репликации, комплекс распознавания ориджина (ORC), Cdc6, Cdt1 и белковый комплекс поддержания минихромосомы (Mcm) последовательно связываются с ДНК с образованием пререпликационного комплекса (pre-RC). При переходе G1 стадии в S-фазу клеточного цикла, S-фазу специфичную циклин-зависимый белок киназа (CDK) и Cdc7 / Dbf4 киназа (DDK) преобразует pre-RC в активную вилку репликации. Во время этого преобразования пре-RC разбирается с потерей Cdc6, создавая комплекс инициации. Помимо связывания белков Mcm, цикл деления клеток 45 Белок (Cdc45) также необходим для инициации репликации ДНК.[43][44] Исследования показали, что Mcm является критическим для загрузки Cdc45 на хроматин, и этот комплекс, содержащий как Mcm, так и Cdc45, образуется в начале S-фазы клеточного цикла.[45][46] Cdc45 нацелен на белковый комплекс Mcm, который загружен на хроматин, как компонент пре-RC в ориджине репликации во время G1 стадия клеточного цикла.[47]
Cdc45 белок
Цикл деления клеток 45 Белок (Cdc45) является критическим компонентом превращения пререпликативного комплекса в комплекс инициации. Белок Cdc45 собирается в точках репликации до инициации и необходим для начала репликации в Saccharomyces cerevisiae, и играет важную роль при удлинении. Таким образом, Cdc45 играет центральную роль как в фазах инициации, так и в фазах удлинения репликации хромосомной ДНК.[48]
Cdc45 ассоциирует с хроматином после начала инициации в конце G1 стадии и во время фазы S клеточного цикла. Cdc45 физически ассоциируется с Mcm5 и демонстрирует генетические взаимодействия с пятью из шести членов семейства генов Mcm и ORC2 ген.[49][47] Загрузка Cdc45 на хроматин имеет решающее значение для загрузки других различных белков репликации, включая ДНК-полимераза α, ДНК-полимераза ε, репликационный белок А (RPA) и ядерный антиген пролиферирующих клеток (PCNA) на хроматин.[46][50][51][52]В пределах Xenopus Система без ядра, было продемонстрировано, что Cdc45 необходим для раскручивания плазмидной ДНК.[52] В Xenopus Безъядерная система также демонстрирует, что раскручивание ДНК и прочное связывание RPA с хроматином происходит только в присутствии Cdc45.[46]
Связывание Cdc45 с хроматином зависит от активности киназы Clb-Cdc28, а также от функциональных Cdc6 и Mcm2, что свидетельствует о том, что Cdc45 связывается с пре-RC после активации S-фазы циклин-зависимых киназ (CDK). Как показывает время и зависимость от CDK, связывание Cdc45 с хроматином является критическим для инициации репликации ДНК. Во время S фазы Cdc45 физически взаимодействует с белками Mcm на хроматине; однако диссоциация Cdc45 от хроматина происходит медленнее, чем диссоциация Mcm, что указывает на то, что белки высвобождаются с помощью разных механизмов.[34]
Джинс
Шесть поддерживающих белков минихромосом и Cdc45 необходимы во время инициации и удлинения для движения репликационных вилок и для раскручивания ДНК. GINS важны для взаимодействия Mcm и Cdc45 в источниках репликации во время инициации, а затем в ответвлениях репликации ДНК по мере развития реплисомы.[53][54] Комплекс GINS состоит из четырех небольших белков Sld5 (Cdc105), Psf1 (Cdc101), Psf2 (Cdc102) и Psf3 (Cdc103), GINS представляет собой «go, ichi, ni, san», что означает «5, 1, 2, 3». ' на японском языке.[55] Cdc45, Mcm2-7 и GINS вместе образуют геликазу CMG,[56] репликативная геликаза реплисомы. Хотя комплекс Mcm2-7 сам по себе имеет слабую геликазную активность [57] Cdc45 и GINS необходимы для устойчивой активности геликазы[58][59]
Mcm10
Mcm10 необходим для репликации хромосом и взаимодействует с минихромосомной поддерживающей геликазой 2-7, которая загружается в неактивной форме в ориджинах репликации ДНК. [60] [61] Mcm10 также шапероны каталитическая ДНК-полимераза α и помогает стабилизировать полимеразу в репликационных вилках.[62][63]
Киназы DDK и CDK
В начале S-фазы пререпликативный комплекс должен быть активирован двумя киназами, специфичными для S-фазы, чтобы сформировать комплекс инициации в ориджине репликации. Одна киназа - это киназа Cdc7-Dbf4, называемая Dbf4-зависимой киназой (DDK), а другая - циклин-зависимая киназа (CDK).[64] Анализ связывания хроматина Cdc45 в дрожжах и Xenopus показали, что последующим событием действия CDK является загрузка Cdc45 на хроматин.[44][45] Cdc6 предполагалось, что это цель действия CDK из-за ассоциации между Cdc6 и CDK, а также CDK-зависимой фосфорилирование из Cdc6. Считается, что CDK-зависимое фосфорилирование Cdc6 необходимо для перехода в S-фазу.[65]
Как каталитические субъединицы DDK, Cdc7, так и активаторный белок, Dbf4, консервативны у эукариот и необходимы для начала S-фазы клеточного цикла.[66][67] И DDK, и Cdc7 необходимы для загрузки Cdc45 на точки начала репликации хроматина. Мишенью для связывания киназы DDK является комплекс Mcm, возможно, Mcm2.[68][66] DDK нацелен на комплекс Mcm, и его фосфорилирование приводит к возможной активации активности геликазы Mcm.[69]
Белки Dpb11, Sld3 и Sld2
Sld3, Sld2 и Dpb11 взаимодействуют со многими белками репликации. Sld3 и Cdc45 образуют комплекс, который связан с пре-RC в ранних источниках репликации даже в G1.1 фазы и с более поздними источниками репликации в S фазе взаимно Mcm-зависимым образом.[70][71] Dpb11 и Sld2 взаимодействуют с полимеразой ɛ, и эксперименты по перекрестному связыванию показали, что Dpb11 и полимераза ɛ совместно преципитируют в S-фазе и связываются с источниками репликации.[72][73]
Sld3 и Sld2 фосфорилируются CDK, что позволяет двум репликативным белкам связываться с Dpb11. Dpb11 имел две пары доменов С-конца BRCA1 (BRCT), которые известны как фосфопептид-связывающие домены.[74] N-концевая пара доменов BRCT связывается с фосфорилированным Sld3, а C-концевая пара связывается с фосфорилированным Sld2. Оба этих взаимодействия важны для CDK-зависимой активации почкования ДНК у дрожжей.[75]
Dpb11 также взаимодействует с GINS и участвует в этапах инициации и удлинения репликации хромосомной ДНК.[54][76][77] GINS - один из белков репликации, обнаруженных в ответвлениях репликации, и образует комплекс с Cdc45 и Mcm.
Эти зависимые от фосфорилирования взаимодействия между Dpb11, Sld2 и Sld3 важны для CDK-зависимой активации репликации ДНК, и при использовании сшивающих реагентов в некоторых экспериментах был идентифицирован хрупкий комплекс, называемый комплексом предварительной нагрузки (pre-LC). . Этот комплекс содержит Pol ɛ, GINS, Sld2 и Dpb11. Было обнаружено, что пре-LC формируется перед любой ассоциацией с ориджинами CDK-зависимым и DDK-зависимым образом, и активность CDK регулирует инициацию репликации ДНК посредством образования пре-LC.[78]
Удлинение

Образование пререпликативного комплекса (пре-RC) отмечает потенциальные сайты для инициации репликации ДНК. В соответствии с комплексом поддержания минихромосомы, окружающим двухцепочечную ДНК, образование пре-RC не приводит к немедленному раскручиванию исходной ДНК или привлечению ДНК-полимераз. Вместо этого пре-RC, который формируется во время G1 клеточного цикла активируется только для раскручивания ДНК и инициации репликации после того, как клетки перейдут из G1 к S-фазе клеточного цикла.[2]
Как только инициирующий комплекс образуется и клетки переходят в S-фазу, комплекс становится реплисомой. Комплекс реплисом эукариот отвечает за координацию репликации ДНК. Репликация на ведущей и отстающей цепях осуществляется ДНК-полимеразой ε и ДНК-полимеразой δ. Многие факторы реплисомы, включая Claspin, And1, загрузчик зажима фактора репликации C и комплекс защиты вилки, ответственны за регуляцию функций полимеразы и координацию синтеза ДНК с раскручиванием цепи матрицы комплексом Cdc45-Mcm-GINS. По мере разматывания ДНК число скручивания уменьшается. Чтобы компенсировать это, корчиться число увеличивается, привнося положительные суперспирали в ДНК. Эти суперспирали остановили бы репликацию ДНК, если бы они не были удалены. Топоизомеразы отвечают за удаление этих суперспиралей перед вилкой репликации.
Реплисома отвечает за копирование всей геномной ДНК в каждой пролиферативной клетке. Реакции спаривания оснований и образования цепей, образующие дочернюю спираль, катализируются ДНК-полимеразами.[79] Эти ферменты перемещаются по одноцепочечной ДНК и позволяют удлинить зарождающуюся цепь ДНК, «считывая» цепочку-матрицу и обеспечивая включение надлежащего пурин азотистые основания, аденин и гуанин, и пиримидин азотистые основания, тимин и цитозин. Активировано бесплатно дезоксирибонуклеотиды существуют в клетке как дезоксирибонуклеотидтрифосфаты (дНТФ). Эти свободные нуклеотиды добавляются к открытой 3'-гидроксильной группе на последнем включенном нуклеотиде. В этой реакции из свободного dNTP высвобождается пирофосфат, генерируя энергию для реакции полимеризации и обнажая 5'-монофосфат, который затем ковалентно связывается с 3'-кислородом. Кроме того, неправильно вставленные нуклеотиды могут быть удалены и заменены правильными нуклеотидами в энергетически выгодной реакции. Это свойство жизненно важно для правильной вычитки и исправления ошибок, возникающих во время репликации ДНК.
Вилка репликации
Репликационная вилка - это соединение между недавно разделенными цепями матрицы, известными как ведущая и отстающая цепи, и двухцепочечной ДНК. Поскольку дуплексная ДНК антипараллельна, репликация ДНК происходит в противоположных направлениях между двумя новыми цепями в репликационной вилке, но все ДНК-полимеразы синтезируют ДНК в направлении от 5 'до 3' относительно вновь синтезированной цепи. Дальнейшая координация требуется во время репликации ДНК. Две репликативные полимеразы синтезируют ДНК в противоположных ориентациях. Полимераза ε непрерывно синтезирует ДНК на «ведущей» цепи ДНК, поскольку она указывает в том же направлении, что и раскручивание ДНК реплисомой. Напротив, полимераза δ синтезирует ДНК на «отстающей» цепи, которая является противоположной цепью матрицы ДНК, фрагментированным или прерывистым образом.
Прерывистые участки продуктов репликации ДНК на отстающей цепи известны как фрагменты Окадзаки и составляют от 100 до 200 оснований в длину на эукариотических вилках репликации. Отстающая цепь обычно содержит более длинные участки одноцепочечной ДНК, покрытой одноцепочечными связывающими белками, которые помогают стабилизировать одноцепочечные матрицы, предотвращая образование вторичной структуры. У эукариот эти одноцепочечные связывающие белки представляют собой гетеротримерный комплекс, известный как репликационный белок А (РПА).[80]
Каждому фрагменту Окадзаки предшествует праймер РНК, который в процессе синтеза замещается последовательностью следующего фрагмента Окадзаки. РНКаза H распознает гибриды ДНК: РНК, которые создаются с помощью праймеров РНК, и отвечает за их удаление из реплицированной цепи, оставляя после себя соединение праймер: матрица. ДНК-полимераза α распознает эти сайты и удлиняет разрывы, оставшиеся после удаления праймера. В эукариотических клетках небольшое количество сегмента ДНК непосредственно перед праймером РНК также смещается, создавая лоскутную структуру. Затем этот лоскут расщепляется эндонуклеазами. В репликационной вилке разрыв в ДНК после удаления лоскута закрывается ДНК-лигаза I, который восстанавливает щели, оставшиеся между 3'-ОН и 5'-фосфатом вновь синтезированной цепи.[81] Из-за относительно короткой природы эукариотического фрагмента Окадзаки, синтез репликации ДНК, происходящий прерывисто на отстающей цепи, менее эффективен и требует больше времени, чем синтез ведущей цепи. Синтез ДНК завершается после удаления всех праймеров РНК и заделки зазубрин.

Ведущая прядь
Во время репликации ДНК реплисома будет раскручивать родительскую дуплексную ДНК в двухцепочечную матричную репликационную вилку ДНК в направлении 5 'на 3'. Ведущая нить - это нить-матрица, которая реплицируется в том же направлении, что и движение репликационной вилки. Это позволяет синтезировать вновь синтезированную цепь, комплементарную исходной цепи, от 5 'до 3' в том же направлении, что и движение репликационной вилки.[82]
После того, как праймер РНК был добавлен примазой к 3'-концу ведущей цепи, синтез ДНК будет продолжаться в направлении от 3 'до 5' по отношению к ведущей цепи без прерывания. ДНК-полимераза ε будет непрерывно добавлять нуклеотиды к матричной цепи, поэтому для синтеза ведущей цепи требуется только один праймер и она имеет непрерывную активность ДНК-полимеразы.[83]
Отстающая прядь
Репликация ДНК на отстающая нить прерывистый. При синтезе отстающих цепей движение ДНК-полимераза в направлении, противоположном репликационной вилке, требует использования нескольких Праймеры РНК. ДНК-полимераза будет синтезировать короткие фрагменты ДНК, называемые Фрагменты Окадзаки которые добавляются к 3 'концу грунтовки. Эти фрагменты могут иметь длину от 100 до 400 нуклеотидов у эукариот.[84]
В конце синтеза фрагмента Окадзаки ДНК-полимераза δ проходит в предыдущий фрагмент Окадзаки и замещает его 5'-конец, содержащий праймер РНК и небольшой сегмент ДНК. В результате образуется однонитевой лоскут РНК-ДНК, который необходимо расщепить, а разрыв между двумя фрагментами Окадзаки должен быть запломбирован ДНК-лигазой I. Этот процесс известен как созревание фрагмента Окадзаки и может осуществляться двумя способами: процесс с одним механизмом короткие закрылки, а другой касается длинных закрылков.[85] ДНК-полимераза δ способна замещать до 2–3 нуклеотидов ДНК или РНК перед ее полимеризацией, образуя короткий субстрат «лоскута» для Fen1, который может удалять нуклеотиды из лоскута, по одному нуклеотиду за раз.
Повторяя циклы этого процесса, ДНК-полимераза δ и Fen1 могут координировать удаление праймеров РНК и оставлять разрыв ДНК на отстающей цепи.[86] Было высказано предположение, что этот итерационный процесс предпочтительнее, чем клеточный, потому что он жестко регулируется и не создает больших лоскутов, которые необходимо иссекать.[87] В случае нарушения регуляции активности Fen1 / ДНК-полимеразы δ клетка использует альтернативный механизм для создания и обработки длинных створок с помощью Dna2, которая обладает как геликазной, так и нуклеазной активностями.[88] Нуклеазная активность Dna2 необходима для удаления этих длинных лоскутов, оставляя более короткий лоскут для обработки Fen1. Электронная микроскопия исследования показывают, что нагрузка нуклеосом на отстающую цепь происходит очень близко к сайту синтеза.[89] Таким образом, созревание фрагмента Окадзаки представляет собой эффективный процесс, который происходит сразу после синтеза зарождающейся ДНК.
Репликативные ДНК-полимеразы
После того, как репликативная геликаза размотала дуплекс родительской ДНК, открыв две одноцепочечные ДНК-матрицы, необходимы репликативные полимеразы для генерации двух копий родительского генома. Функция ДНК-полимеразы является узкоспециализированной и выполняет репликацию на определенных шаблонах и в узких локализациях. На вилке репликации эукариот есть три различных репликативных полимеразных комплекса, которые способствуют репликации ДНК: полимераза α, полимераза δ и полимераза ε. Эти три полимеразы необходимы для жизнеспособности клетки.[90]
Поскольку ДНК-полимеразам требуется праймер для начала синтеза ДНК, полимераза α (Pol α) действует как репликативная примаза. Pol α связан с РНК-примазой, и этот комплекс выполняет задачу праймирования путем синтеза праймера, который содержит короткий 10-нуклеотидный участок РНК, за которым следуют от 10 до 20 оснований ДНК.[3] Важно, что это прайминговое действие происходит при инициации репликации в ориджинах, чтобы начать синтез ведущей цепи, а также на 5'-конце каждого фрагмента Окадзаки на отстающей цепи.
Однако Pol α не может продолжать репликацию ДНК и должен быть заменен другой полимеразой для продолжения синтеза ДНК.[91] Переключение полимеразы требует загрузчиков зажимов, и было доказано, что нормальная репликация ДНК требует скоординированных действий всех трех ДНК-полимераз: Pol α для праймингового синтеза, Pol ε для репликации ведущей цепи и Pol δ, который постоянно загружается, для генерации Фрагменты Окадзаки во время синтеза отстающей цепи.[92]
- Полимераза α (Pol α): Образует комплекс с малой каталитической субъединицей (PriS) и большой некаталитической (PriL) субъединицей.[93] Во-первых, синтез праймера РНК позволяет синтезировать ДНК ДНК-полимеразой альфа. Происходит один раз в начале на ведущей нити и в начале каждого фрагмента Окадзаки на отстающей нити. Субъединицы Pri действуют как примаза, синтезируя праймер РНК. ДНК Pol α удлиняет вновь образованный праймер с нуклеотидами ДНК. Примерно через 20 нуклеотидов удлинение передается Pol ε на ведущей цепи и Pol δ на отстающей цепи.[94]
- Полимераза δ (Pol δ): Высокопроизводительный и корректирующий, 3 '-> 5' экзонуклеазная активность. In vivo, это основная полимераза, участвующая в обеих отстающих цепях и синтез ведущей цепи.[95]
- Полимераза ε (Pol ε): Высокопроизводительный и корректирующий, 3 '-> 5' экзонуклеазная активность. Сильно связан с pol δ, in vivo он функционирует в основном при проверке ошибок pol δ.[95]
Cdc45 – Mcm – GINS геликазный комплекс
ДНК геликасы и полимеразы должны оставаться в тесном контакте на вилке репликации. Если раскручивание происходит слишком далеко до синтеза, обнажаются большие участки одноцепочечной ДНК. Это может активировать сигнализацию повреждения ДНК или вызвать процессы репарации ДНК. Чтобы предотвратить эти проблемы, реплисома эукариот содержит специализированные белки, которые предназначены для регулирования активности геликазы перед репликационной вилкой. Эти белки также обеспечивают стыковочные участки для физического взаимодействия между геликазами и полимеразами, тем самым гарантируя, что раскручивание дуплекса связано с синтезом ДНК.[96]
Для функционирования ДНК-полимераз необходимо размотать двухцепочечную спираль ДНК, чтобы открыть две матрицы одноцепочечной ДНК для репликации. ДНК-геликазы ответственны за раскручивание двухцепочечной ДНК во время репликации хромосомы. Геликазы в эукариотических клетках чрезвычайно сложны.[97] Каталитическое ядро геликазы состоит из шести белков поддержания минихромосом (Mcm2-7), образующих гексамерный звенеть. Вдали от ДНК белки Mcm2-7 образуют одиночный гетерогексамер и загружаются в неактивной форме в начале репликации ДНК в виде двойных гексамеров, расположенных «голова к голове» вокруг двухцепочечной ДНК.[97][98] Белки Mcm рекрутируются в источники репликации, а затем перераспределяются по геномной ДНК во время S-фазы, что указывает на их локализацию в репликационной вилке.[47]
Загрузка белков Mcm может происходить только во время G1 of the cell cycle, and the loaded complex is then activated during S phase by recruitment of the Cdc45 protein and the GINS complex to form the active Cdc45–Mcm–GINS (CMG) helicase at DNA replication forks.[99][100] Mcm activity is required throughout the S phase for DNA replication.[35][101] A variety of regulatory factors assemble around the CMG helicase to produce the ‘Replisome Progression Complex’ which associates with DNA polymerases to form the eukaryotic replisome, the structure of which is still quite poorly defined in comparison with its bacterial counterpart.[53][102]
The isolated CMG helicase and Replisome Progression Complex contain a single Mcm protein ring complex suggesting that the loaded double hexamer of the Mcm proteins at origins might be broken into two single hexameric rings as part of the initiation process, with each Mcm protein complex ring forming the core of a CMG helicase at the two replication forks established from each origin.[53][99] The full CMG complex is required for DNA unwinding, and the complex of CDC45-Mcm-GINS is the functional DNA helicase in eukaryotic cells.[103]
Ctf4 and And1 proteins
The CMG complex interacts with the replisome through the interaction with Ctf4 and And1 proteins. Ctf4/And1 proteins interact with both the CMG complex and DNA polymerase α.[104] Ctf4 is a polymerase α accessory factor, which is required for the recruitment of polymerase α to replication origins.[105]
Mrc1 and Claspin proteins
Mrc1/Claspin proteins couple leading-strand synthesis with the CMG complex helicase activity. Mrc1 interacts with polymerase ε as well as Mcm proteins.[106] The importance of this direct link between the helicase and the leading-strand polymerase is underscored by results in cultured human cells, where Mrc1/Claspin is required for efficient replication fork progression.[107] These results suggest that efficient DNA replication also requires the coupling of helicases and leading-strand synthesis...
Ядерный антиген пролиферирующих клеток
DNA polymerases require additional factors to support DNA replication. DNA polymerases have a semiclosed 'hand' structure, which allows the polymerase to load onto the DNA and begin translocating. This structure permits DNA polymerase to hold the single-stranded DNA template, incorporate dNTPs at the active site, and release the newly formed double-stranded DNA. However, the structure of DNA polymerases does not allow a continuous stable interaction with the template DNA.[1]
To strengthen the interaction between the polymerase and the template DNA, DNA sliding clamps associate with the polymerase to promote the процессивность of the replicative polymerase. In eukaryotes, the sliding clamp is a homotrimer ring structure known as the ядерный антиген пролиферирующих клеток (PCNA). The PCNA ring has polarity with surfaces that interact with DNA polymerases and tethers them securely to the DNA template. PCNA-dependent stabilization of DNA polymerases has a significant effect on DNA replication because PCNAs are able to enhance the polymerase processivity up to 1,000-fold.[108][109] PCNA is an essential cofactor and has the distinction of being one of the most common interaction platforms in the replisome to accommodate multiple processes at the replication fork, and so PCNA is also viewed as a regulatory cofactor for DNA polymerases.[110]
Фактор репликации C
PCNA fully encircles the DNA template strand and must be loaded onto DNA at the replication fork. At the leading strand, loading of the PCNA is an infrequent process, because DNA replication on the leading strand is continuous until replication is terminated. However, at the lagging strand, DNA polymerase δ needs to be continually loaded at the start of each Okazaki fragment. This constant initiation of Okazaki fragment synthesis requires repeated PCNA loading for efficient DNA replication.
PCNA loading is accomplished by the фактор репликации C (RFC) complex. The RFC complex is composed of five ATPases: Rfc1, Rfc2, Rfc3, Rfc4 and Rfc5.[111] RFC recognizes primer-template junctions and loads PCNA at these sites.[112][113] The PCNA homotrimer is opened by RFC by ATP hydrolysis and is then loaded onto DNA in the proper orientation to facilitate its association with the polymerase.[114][115] Clamp loaders can also unload PCNA from DNA; a mechanism needed when replication must be terminated.[115]
Stalled replication fork
Репликация ДНК at the replication fork can be halted by a shortage of deoxynucleotide triphosphates (dNTPs) or by DNA damage, resulting in репликационный стресс.[116] This halting of replication is described as a stalled replication fork. A fork protection complex of proteins stabilizes the replication fork until DNA damage or other replication problems can be fixed.[116] Prolonged replication fork stalling can lead to further DNA damage. Stalling signals are deactivated if the problems causing the replication fork are resolved.[116]
Прекращение

Termination of eukaryotic DNA replication requires different processes depending on whether the chromosomes are circular or linear. Unlike linear molecules, circular chromosomes are able to replicate the entire molecule. However, the two DNA molecules will remain linked together. This issue is handled by decatenation of the two DNA molecules by a type II topoisomerase. Type II topoisomerases are also used to separate linear strands as they are intricately folded into a nucleosome within the cell.
As previously mentioned, linear chromosomes face another issue that is not seen in circular DNA replication. Due to the fact that an RNA primer is required for initiation of DNA synthesis, the lagging strand is at a disadvantage in replicating the entire chromosome. While the leading strand can use a single RNA primer to extend the 5' terminus of the replicating DNA strand, multiple RNA primers are responsible for lagging strand synthesis, creating Okazaki fragments. This leads to an issue due to the fact that DNA polymerase is only able to add to the 3' end of the DNA strand. The 3'-5' action of DNA polymerase along the parent strand leaves a short single-stranded DNA (ssDNA) region at the 3' end of the parent strand when the Okazaki fragments have been repaired. Since replication occurs in opposite directions at opposite ends of parent chromosomes, each strand is a lagging strand at one end. Over time this would result in progressive shortening of both дочерние хромосомы. This is known as the end replication problem.[1]
The end replication problem is handled in eukaryotic cells by теломер регионы и теломераза. Telomeres extend the 3' end of the parental chromosome beyond the 5' end of the daughter strand. This single-stranded DNA structure can act as an origin of replication that recruits telomerase. Telomerase is a specialized DNA polymerase that consists of multiple protein subunits and an RNA component. The RNA component of telomerase anneals to the single stranded 3' end of the template DNA and contains 1.5 copies of the telomeric sequence.[84] Telomerase contains a protein subunit that is a обратная транскриптаза называется теломераза обратная транскриптаза or TERT. TERT synthesizes DNA until the end of the template telomerase RNA and then disengages.[84] This process can be repeated as many times as needed with the extension of the 3' end of the parental DNA molecule. This 3' addition provides a template for extension of the 5' end of the daughter strand by lagging strand DNA synthesis. Regulation of telomerase activity is handled by telomere-binding proteins.
Replication fork barriers
Prokaryotic DNA replication is bidirectional; within a replicative origin, replisome complexes are created at each end of the replication origin and replisomes move away from each other from the initial starting point. In prokaryotes, bidirectional replication initiates at one replicative origin on the circular chromosome and terminates at a site opposed from the initial start of the origin.[117] These termination regions have DNA sequences known as Тер места. Эти Тер sites are bound by the Tus protein. В Тер-Tus complex is able to stop helicase activity, terminating replication.[118]
In eukaryotic cells, termination of replication usually occurs through the collision of the two replicative forks between two active replication origins. The location of the collision varies on the timing of origin firing. In this way, if a replication fork becomes stalled or collapses at a certain site, replication of the site can be rescued when a replisome traveling in the opposite direction completes copying the region. There are programmed replication fork barriers (RFBs) bound by RFB proteins in various locations, throughout the genome, which are able to terminate or pause replication forks, stopping progression of the replisome.[117]
Replication factories
It has been found that replication happens in a localised way in the cell nucleus. Contrary to the traditional view of moving replication forks along stagnant DNA, a concept of replication factories emerged, which means replication forks are concentrated towards some immobilised 'factory' regions through which the template DNA strands pass like conveyor belts. [119]
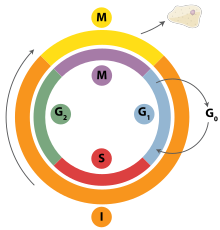
Cell cycle regulation
DNA replication is a tightly orchestrated process that is controlled within the context of the клеточный цикл. Progress through the cell cycle and in turn DNA replication is tightly regulated by the formation and activation of pre-replicative complexes (pre-RCs) which is achieved through the activation and inactivation of cyclin-dependent kinases (Cdks, CDKs). Specifically it is the interactions of циклины and cyclin dependent kinases that are responsible for the transition from G1 into S-phase.
During the G1 phase of the cell cycle there are low levels of CDK activity. This low level of CDK activity allows for the formation of new pre-RC complexes but is not sufficient for DNA replication to be initiated by the newly formed pre-RCs. During the remaining phases of the cell cycle there are elevated levels of CDK activity. This high level of CDK activity is responsible for initiating DNA replication as well as inhibiting new pre-RC complex formation.[2] Once DNA replication has been initiated the pre-RC complex is broken down. Due to the fact that CDK levels remain high during the S phase, G2, and M phases of the cell cycle no new pre-RC complexes can be formed. This all helps to ensure that no initiation can occur until the cell division is complete.
In addition to cyclin dependent kinases a new round of replication is thought to be prevented through the downregulation of Cdt1. This is achieved via degradation of Cdt1 as well as through the inhibitory actions of a protein known as близнецы. Geminin binds tightly to Cdt1 and is thought to be the major inhibitor of re-replication.[2] Geminin first appears in S-phase and is degraded at the metaphase-anaphase transition, possibly through ubiquination by комплекс, способствующий анафазе (БТР).[120]
Разные контрольные точки клеточного цикла are present throughout the course of the cell cycle that determine whether a cell will progress through division entirely. Importantly in replication the G1, or restriction, checkpoint makes the determination of whether or not initiation of replication will begin or whether the cell will be placed in a resting stage known as грамм0. Cells in the G0 stage of the cell cycle are prevented from initiating a round of replication because the minichromosome maintenance proteins are not expressed. Transition into the S-phase indicates replication has begun.
Replication checkpoint proteins
In order to preserve genetic information during cell division, DNA replication must be completed with high fidelity. In order to achieve this task, eukaryotic cells have proteins in place during certain points in the replication process that are able to detect any errors during DNA replication and are able to preserve genomic integrity. These checkpoint proteins are able to stop the cell cycle from entering mitosis in order to allow time for DNA repair. Checkpoint proteins are also involved in some DNA repair pathways, while they stabilize the structure of the replication fork to prevent further damage. These checkpoint proteins are essential to avoid passing down mutations or other chromosomal aberrations to offspring.
Eukaryotic checkpoint proteins are well conserved and involve two phosphatidylinositol 3-kinase-related kinases (PIKKs), ATR и Банкомат. Both ATR and ATM share a target phosphorylation sequence, the SQ/TQ motif, but their individual roles in cells differ.[121]
ATR is involved in arresting the cell cycle in response to DNA double-stranded breaks. ATR has an obligate checkpoint partner, ATR-interacting-protein (ATRIP), and together these two proteins are responsive to stretches of single-stranded DNA that are coated by replication protein A (RPA).[122] The formation of single-stranded DNA occurs frequently, more often during replication stress. ATR-ATRIP is able to arrest the cell cycle to preserve genome integrity. ATR is found on chromatin during S phase, similar to RPA and claspin.[123]
The generation of single-stranded DNA tracts is important in initiating the checkpoint pathways downstream of replication damage. Once single-stranded DNA becomes sufficiently long, single-stranded DNA coated with RPA are able to recruit ATR-ATRIP.[124] In order to become fully active, the ATR kinase rely on sensor proteins that sense whether the checkpoint proteins are localized to a valid site of DNA replication stress. В RAD9 -HUS1 -Rad1 (9-1-1) heterotrimeric clamp and its clamp loader RFCRad17 are able to recognize gapped or nicked DNA. The RFCRad17 clamp loader loads 9-1-1 onto the damaged DNA.[125] The presence of 9-1-1 on DNA is enough to facilitate the interaction between ATR-ATRIP and a group of proteins termed checkpoint mediators, such as TOPBP1 and Mrc1/claspin. TOPBP1 interacts with and recruits the phosphorylated Rad9 component of 9-1-1 and binds ATR-ATRIP, which phosphorylates Chk1.[126] Mrc1/Claspin is also required forthe complete activation of ATR-ATRIP that phosphorylates Chk1, the major downstream checkpoint effector kinase.[127] Claspin is a component of the replisome and contains a domain for docking with Chk1, revealing a specific function of Claspin during DNA replication: the promotion ofcheckpoint signaling at the replisome.[128]
Chk1 signaling is vital for arresting the cell cycle and preventing cells from entering mitosis with incomplete DNA replication or DNA damage. The Chk1-dependent Cdk inhibition is important for the function of the ATR-Chk1 checkpoint and to arrest the cell cycle and allow sufficient time for completion of DNA repair mechanisms, which in turn prevents the inheritance of damaged DNA. In addition, Chk1-dependent Cdk inhibition plays a critical role in inhibiting origin firing during S phase. This mechanism prevents continued DNA synthesis and is required for the protection of the genome in thepresence of replication stress and potential genotoxic conditions.[129] Thus, ATR-Chk1 activity further prevents potential replication problems at the level of single replication origins by inhibiting initiation of replication throughout the genome, until the signaling cascade maintaining cell-cycle arrest is turned off.
Replication through nucleosomes

Eukaryotic DNA must be tightly compacted in order to fit within the confined space of the nucleus. Chromosomes are packaged by wrapping 147 nucleotides around an octamer of гистон proteins, forming a нуклеосома. The nucleosome octamer includes two copies of each histone H2A, H2B, H3, и H4. Due to the tight association of histone proteins to DNA, eukaryotic cells have proteins that are designed to remodel histones ahead of the replication fork, in order to allow smooth progression of the replisome.[130] There are also proteins involved in reassembling histones behind the replication fork to reestablish the nucleosome conformation.[131]
There are several histone chaperones that are known to be involved in nucleosome assembly after replication. В ФАКТ complex has been found to interact with DNA polymerase α-primase complex, and the subunits of the FACT complex interacted genetically with replication factors.[132][133] The FACT complex is a heterodimer that does not hydrolyze ATP, but is able to facilitate "loosening" of histones in nucleosomes, but how the FACT complex is able to relieve the tight association of histones for DNA removal remains unanswered.[134]
Another histone chaperone that associates with the replisome is Asf1, which interacts with the Mcm complex dependent on histone dimers H3-H4.[135] Asf1 is able to pass newly synthesized H3-H4 dimer to deposition factors behind the replication fork and this activity makes the H3-H4 histone dimers available at the site of histone deposition just after replication.[136] Asf1 (and its partner Rtt109) has also been implicated in inhibiting gene expression from replicated genes during S-phase.[137]
The heterotrimeric chaperone chromatin assembly factor 1 (CAF-1 ) is a chromatin formation protein that is involved in depositing histones onto both newly replicated DNA strands to form chromatin.[138] CAF-1 contains a PCNA-binding motif, called a PIP-box, that allows CAF-1 to associate with the replisome through PCNA and is able to deposit histone H3-H4 dimers onto newly synthesized DNA.[139][140] The Rtt106 chaperone is also involved in this process, and associated with CAF-1 and H3-H4 dimers during chromatin formation.[141] These processes load newly synthesized histones onto DNA.
After the deposition of histones H3-H4, nucleosomes form by the association of histone H2A-H2B. This process is thought to occur through the FACT complex, since it already associated with the replisome and is able to bind free H2A-H2B, or there is the possibility of another H2A-H2B chaperone, Nap1.[142] Electron microscopy studies show that this occurs very quickly, as nucleosomes can be observed forming just a few hundred base pairs after the replication fork.[143] Therefore, the entire process of forming newnucleosomes takes place just after replication due to the coupling of histone chaperones to the replisome.
Comparisons between prokaryotic and eukaryotic DNA replication
По сравнению с prokaryotic DNA replication, the completion of eukaryotic DNA replication is more complex and involves multiple истоки репликации and replicative proteins to accomplish. Prokaryotic DNA is arranged in a circular shape, and has only one replication origin when replication starts. By contrast, eukaryotic DNA is linear. When replicated, there are as many as one thousand origins of replication.[144]
Eukaryotic DNA is bidirectional. Here the meaning of the word bidirectional is different. Eukaryotic linear DNA has many origins (called O) and termini (called T). "T" is present to the right of "O". One "O" and one "T" together form one replicon. After the formation of pre-initiation complex, when one replicon starts elongation, initiation starts in second replicon. Now, if the first replicon moves in clockwise direction, the second replicon moves in anticlockwise direction, until "T" of first replicon is reached. At "T", both the replicons merge to complete the process of replication. Meanwhile, the second replicon is moving in forward direction also, to meet with the third replicon. This clockwise and counter-clockwise movement of two replicons is termed as bidirectional replication.
Eukaryotic DNA replication requires precise coordination of all DNA polymerases and associated proteins to replicate the entire genome each time a cell divides. This process is achieved through a series of steps of protein assemblies at origins of replication, mainly focusing the regulation of DNA replication on the association of the MCM helicase with the DNA. These origins of replication direct the number of protein complexes that will form to initiate replication. In prokaryotic DNA replication regulation focuses on the binding of the DnaA initiator protein to the DNA, with initiation of replication occurring multiple times during one cell cycle.[84] Both prokaryotic and eukaryotic DNA use ATP binding and hydrolysis to direct helicase loading and in both cases the helicase is loaded in the inactive form. However, eukaryotic helicases are double hexamers that are loaded onto double stranded DNA whereas prokaryotic helicases are single hexamers loaded onto single stranded DNA.[145]
Segregation of chromosomes is another difference between prokaryotic and eukaryotic cells. Rapidly dividing cells, such as bacteria, will often begin to segregate chromosomes that are still in the process of replication. In eukaryotic cells chromosome segregation into the daughter cells is not initiated until replication is complete in all chromosomes.[84] Despite these differences, however, the underlying process of replication is similar for both prokaryotic and eukaryotic DNA.
| Prokaryotic DNA Replication | Репликация эукариотической ДНК |
|---|---|
| Occurs inside the cytoplasm | Occurs inside the nucleus |
| Only one origin of replication per molecule of DNA | Have many origins of replication in each chromosome |
| Origin of replication is about 100-200 or more nucleotides in length | Each origin of replication is formed of about 150 nucleotides |
| Replication occurs at one point in each chromosome | Replication occurs at several points simultaneously in each chromosome |
| Only have one origin of replication | Has multiple origins of replication |
| Initiation is carried out by protein DnaA and DnaB | Initiation is carried out by the Origin Recognition Complex |
| Topoisomerase is needed | Topoisomerase is needed |
| Replication is very rapid | Replication is very slow |
Eukaryotic DNA replication protein list
List of major proteins involved in eukaryotic DNA replication:
| Протеин | Function in Eukaryotic DNA replication |
|---|---|
| AND1 | Loads DNA polymerase α onto chromatin together with CMG complex on the lagging strand. Also known as Ctf4 in budding yeast. |
| Cdc45 | Required for initiation and elongation steps of DNA replication. A part of the Mcm2-7 helicase complex. Required after pre-RC step for loading of various proteins for initiation and elongation. |
| Cdc45-Mcm-GINS (CMG) complex | Functional DNA helicase in eukaryotic cells |
| Cdc6 | Required for assembly of Mcm2-7 complex at ORC, in conjunction with Cdt1 . |
| Cdc7-Dbf4 kinase or Dbf4-dependent kinase (DDK) | Protein kinase required for initiation of DNA replication, probably through phosphorylation of the minichromosome maintenance proteins. |
| Cdt1 | Loads Mcm2-7 complex on DNA at ORC in pre-RC/licensing step. Inhibited in metazoans by geminin. |
| Claspin | Couple leading-strand synthesis with the CMG complex helicase activity. Works with Mrc1 |
| Ctf4 | Loads DNA polymerase α onto chromatin together with CMG complex on the lagging strand. Homolog in metazoans is known as AND-1. |
| Циклинзависимая киназа (CDK) | Cyclin-dependent protein kinase required for initiation of replication and for other subsequent steps. |
| Dna2 | 5' flap endonuclease and helicase involved in processing Okazaki fragments. |
| ДНК-лигаза I | Joins Okazaki fragments during DNA replication. Ligase activity also needed for DNA repair and recombination. |
| DNA polymerase α (Pol α) | Contains primase activity that is necessary to initiate DNA synthesis on both leading and lagging strands. |
| ДНК-полимераза δ (Pol δ) | Required to complete synthesis of Okazaki fragments on the lagging strand that have been started by DNA polymerase α. |
| DNA polymerase ε (Pol ε) | The leading strand polymerase. Synthesizes DNA at the replication fork. Binds early at origins via Dbp11 and needed to load DNA polymerase α. |
| Dpb11 | DNA replication initiation protein. Loads DNA polymerase ε onto pre-replication complexes at origins. |
| Fen1 | 5' flap endonuclease involved in processing Okazaki fragments. |
| Близнецы | Protein found in metazoans and absent from yeasts. Binds to and inactivates Cdt1, thereby regulating pre-replicative/initiation complex formation. Also suggested to promote pre-RC formation by binding and thus preventing Cdt1 degradation |
| GINS | Tetrameric complex composed of Sld5, Psf1, Psf2, Psf3. Associates with pre-replicative complex around the time of initiation and moves with replication forks during elongation step. Required for elongation stage of DNA replication and maybe part of the Mcm helicase complex. |
| Minichromosome maintenance proteins (Mcm) | Six different proteins of the AAA+ ATPase family that form a hexamer in solution. This hexamer is recruited and loaded by ORC, Cdc6 and Cdt1 and forms a double hexamer that is topologically linked around DNA to form a salt-resistant pre-replicative complex. On replication initiation, Mcm2-7 moves away from ORC with replication fork. |
| Mcm10 | Required for initiation and elongation stages of DNA replication. Implicated in chromatin binding of Cdc45 and DNA polymerase α. Also required for stability of DNA polymerase α catalytic subunit in the budding yeast С. cerevisiae. |
| Mrc1 | Couple leading-strand synthesis with the CMG complex helicase activity. Metazoan homolog is known as Claspin. |
| Комплекс распознавания происхождения (ORC) | Heterohexameric complex composed of Orc1–Orc6 proteins. Binds to DNA and assembles Mcm2-7 complex onto chromatin together with Cdc6 and Cdt1. |
| Ядерный антиген пролиферирующих клеток (PCNA) | Trimeric protein with ring shaped structure, encloses DNA preventing dissociation of DNA polymerase. Acts as a sliding clamp for polymerases δ and ε, thereby improving processivity of replicative polymerases. |
| Фактор репликации C (RFC) | Loads PCNA on primed templates and is involved in the switch between DNA polymerase a and the replicative polymerases δ and ε. |
| Replication fork barriers (RFBs) | Bound by RFB proteins in various locations throughout the genome. Are able to terminate or pause replication forks, stopping progression of the replisome. |
| Репликационный белок А (RPA) | Heterotrimeric single-stranded binding protein. Stabilizes single-stranded DNA at replication fork. |
| РНКаза H | Ribonuclease which digests RNA hybridized to DNA. Involved in Okazaki fragment processing. |
| Sld2 | Functions in initiation of replication. Key substrate of CDK, phosphorylation promotes interaction with Dpb11. Required for initiation of replication. |
| Sld3 | Functions in initiation of replication. Key substrate of CDK, phosphorylation promotes interaction with Dpb11. Required for initiation of replication. |
| Теломераза | A ribonucleoprotein that adds DNA sequence "TTAGGG" repeats to the 3' end of DNA strands in telomeres. |
| Топоизомеразы | Regulate the overwinding or underwinding of DNA |
Смотрите также
Рекомендации
- ^ а б c d Leman AR, Noguchi E (March 2013). "The replication fork: understanding the eukaryotic replication machinery and the challenges to genome duplication". Гены. 4 (1): 1–32. Дои:10.3390/genes4010001. ЧВК 3627427. PMID 23599899.
- ^ а б c d Blow JJ, Dutta A (June 2005). "Preventing re-replication of chromosomal DNA". Обзоры природы Молекулярная клеточная биология. 6 (6): 476–86. Дои:10.1038/nrm1663. ЧВК 2688777. PMID 15928711.
- ^ а б Fisher PA, Wang TS, Korn D (July 1979). "Enzymological characterization of DNA polymerase alpha. Basic catalytic properties processivity, and gap utilization of the homogeneous enzyme from human KB cells". Журнал биологической химии. 254 (13): 6128–37. PMID 447699.
- ^ Araki H (2011). "Initiation of chromosomal DNA replication in eukaryotic cells; contribution of yeast genetics to the elucidation". Genes & Genetic Systems. 86 (3): 141–9. Дои:10.1266/ggs.86.141. PMID 21952204.
- ^ Майорано Д., Моро Дж., Мешали М. (апрель 2000 г.). «XCDT1 необходим для сборки пререпликативных комплексов у Xenopus laevis». Природа. 404 (6778): 622–5. Bibcode:2000Натура.404..622М. Дои:10.1038/35007104. PMID 10766247. S2CID 4416138.
- ^ а б c Bell SP, Dutta A (2002). "DNA replication in eukaryotic cells". Ежегодный обзор биохимии. 71: 333–74. Дои:10.1146/annurev.biochem.71.110601.135425. PMID 12045100.
- ^ Tye BK (1999). "MCM proteins in DNA replication". Ежегодный обзор биохимии. 68 (1): 649–86. Дои:10.1146/annurev.biochem.68.1.649. PMID 10872463.
- ^ Ticau S, Friedman LJ, Ivica NA, Gelles J, Bell SP (April 2015). "Single-molecule studies of origin licensing reveal mechanisms ensuring bidirectional helicase loading". Клетка. 161 (3): 513–525. Дои:10.1016/j.cell.2015.03.012. ЧВК 4445235. PMID 25892223.
- ^ а б Zhai Y, Li N, Jiang H, Huang X, Gao N, Tye BK (July 2017). "Unique Roles of the Non-identical MCM Subunits in DNA Replication Licensing". Молекулярная клетка. 67 (2): 168–179. Дои:10.1016/j.molcel.2017.06.016. PMID 28732205.
- ^ Coster G, Diffley JF (July 2017). "Bidirectional eukaryotic DNA replication is established by quasi-symmetrical helicase loading". Science. 357 (6348): 314–318. Bibcode:2017Sci...357..314C. Дои:10.1126/science.aan0063. ЧВК 5608077. PMID 28729513.
- ^ а б c d е Zhai Y, Cheng E, Wu H, Li N, Yung PY, Gao N, Tye BK (March 2017). "Open-ringed structure of the Cdt1-Mcm2-7 complex as a precursor of the MCM double hexamer". Структурная и молекулярная биология природы. 24 (3): 300–308. Дои:10.1038/nsmb.3374. PMID 28191894. S2CID 3929807.
- ^ Bell SP (March 2002). "The origin recognition complex: from simple origins to complex functions". Гены и развитие. 16 (6): 659–72. Дои:10.1101/gad.969602. PMID 11914271.
- ^ Bell, S.P .; Stillman, B. (1992-05-14). "ATP-dependent recognition of eukaryotic origins of DNA replication by a multiprotein complex". Природа. 357 (6374): 128–134. Bibcode:1992Natur.357..128B. Дои:10.1038/357128a0. ISSN 0028-0836. PMID 1579162. S2CID 4346767.
- ^ а б c d е ж Li N, Lam WH, Zhai Y, Cheng J, Cheng E, Zhao Y, Gao N, Tye BK (July 2018). "Structure of the origin recognition complex bound to DNA replication origin". Природа. 559 (7713): 217–222. Bibcode:2018Natur.559..217L. Дои:10.1038/s41586-018-0293-x. PMID 29973722. S2CID 49577101.
- ^ Miotto B, Ji Z, Struhl K (August 2016). "Selectivity of ORC binding sites and the relation to replication timing, fragile sites, and deletions in cancers". Труды Национальной академии наук Соединенных Штатов Америки. 113 (33): E4810-9. Дои:10.1073/pnas.1609060113. ЧВК 4995967. PMID 27436900.
- ^ Bleichert F, Leitner A, Aebersold R, Botchan MR, Berger JM (June 2018). "Conformational control and DNA-binding mechanism of the metazoan origin recognition complex". Труды Национальной академии наук Соединенных Штатов Америки. 115 (26): E5906–E5915. Дои:10.1073/pnas.1806315115. ЧВК 6042147. PMID 29899147.
- ^ Chesnokov IN (2007). "Multiple functions of the origin recognition complex". Международный обзор цитологии. 256: 69–109. Дои:10.1016/S0074-7696(07)56003-1. ISBN 9780123737007. PMID 17241905.
- ^ Matsuda K, Makise M, Sueyasu Y, Takehara M, Asano T, Mizushima T (December 2007). "Yeast two-hybrid analysis of the origin recognition complex of Saccharomyces cerevisiae: interaction between subunits and identification of binding proteins". FEMS дрожжевые исследования. 7 (8): 1263–9. Дои:10.1111/j.1567-1364.2007.00298.x. PMID 17825065.
- ^ Kreitz S, Ritzi M, Baack M, Knippers R (March 2001). "The human origin recognition complex protein 1 dissociates from chromatin during S phase in HeLa cells". Журнал биологической химии. 276 (9): 6337–42. Дои:10.1074/jbc.M009473200. PMID 11102449.
- ^ Speck C, Chen Z, Li H, Stillman B (November 2005). "ATPase-dependent cooperative binding of ORC and Cdc6 to origin DNA". Структурная и молекулярная биология природы. 12 (11): 965–71. Дои:10.1038/nsmb1002. ЧВК 2952294. PMID 16228006.
- ^ Coleman TR, Carpenter PB, Dunphy WG (October 1996). "The Xenopus Cdc6 protein is essential for the initiation of a single round of DNA replication in cell-free extracts". Клетка. 87 (1): 53–63. Дои:10.1016/S0092-8674(00)81322-7. PMID 8858148. S2CID 16897247.
- ^ Rialland M, Sola F, Santocanale C (April 2002). «Важная роль человеческого CDT1 в репликации ДНК и лицензировании хроматина». Journal of Cell Science. 115 (Pt 7): 1435–40. PMID 11896191.
- ^ Tanaka S, Diffley JF (March 2002). "Interdependent nuclear accumulation of budding yeast Cdt1 and Mcm2-7 during G1 phase". Природа клеточной биологии. 4 (3): 198–207. Дои:10.1038/ncb757. PMID 11836525. S2CID 45861829.
- ^ Frigola J, He J, Kinkelin K, Pye VE, Renault L, Douglas ME, Remus D, Cherepanov P, Costa A, Diffley JF (June 2017). "Cdt1 stabilizes an open MCM ring for helicase loading". Nature Communications. 8: 15720. Bibcode:2017NatCo...815720F. Дои:10.1038/ncomms15720. ЧВК 5490006. PMID 28643783.
- ^ Ticau S, Friedman LJ, Champasa K, Corrêa IR, Gelles J, Bell SP (March 2017). "Mechanism and timing of Mcm2-7 ring closure during DNA replication origin licensing". Структурная и молекулярная биология природы. 24 (3): 309–315. Дои:10.1038/nsmb.3375. ЧВК 5336523. PMID 28191892.
- ^ Nishitani H, Lygerou Z, Nishimoto T, Nurse P (April 2000). «Белок Cdt1 необходим для лицензирования ДНК для репликации в делящихся дрожжах». Природа. 404 (6778): 625–8. Bibcode:2000Натура.404..625Н. Дои:10.1038/35007110. PMID 10766248. S2CID 205005540.
- ^ Yuan Z, Riera A, Bai L, Sun J, Nandi S, Spanos C, Chen ZA, Barbon M, Rappsilber J, Stillman B, Speck C, Li H (March 2017). "Structural basis of Mcm2-7 replicative helicase loading by ORC-Cdc6 and Cdt1". Структурная и молекулярная биология природы. 24 (3): 316–324. Дои:10.1038/nsmb.3372. ЧВК 5503505. PMID 28191893.
- ^ Вольшлегель Дж. А., Дуайер Б. Т., Дхар С. К., Цветик С., Уолтер Дж. К., Датта А. (декабрь 2000 г.). «Ингибирование репликации эукариотической ДНК путем связывания геминина с Cdt1». Science. 290 (5500): 2309–12. Bibcode:2000Sci ... 290.2309W. Дои:10.1126 / science.290.5500.2309. PMID 11125146.
- ^ Maine GT, Sinha P, Tye BK (March 1984). "Mutants of S. cerevisiae defective in the maintenance of minichromosomes". Генетика. 106 (3): 365–85. ЧВК 1224244. PMID 6323245.
- ^ Hua XH, Newport J (January 1998). "Identification of a preinitiation step in DNA replication that is independent of origin recognition complex and cdc6, but dependent on cdk2". Журнал клеточной биологии. 140 (2): 271–81. Дои:10.1083/jcb.140.2.271. ЧВК 2132576. PMID 9442103.
- ^ Rowles A, Tada S, Blow JJ (June 1999). "Changes in association of the Xenopus origin recognition complex with chromatin on licensing of replication origins". Journal of Cell Science. 112 (12): 2011–8. ЧВК 3605702. PMID 10341218.
- ^ а б c Li N, Zhai Y, Zhang Y, Li W, Yang M, Lei J, Tye BK, Gao N (August 2015). "Structure of the eukaryotic MCM complex at 3.8 Å". Природа. 524 (7564): 186–91. Bibcode:2015Natur.524..186L. Дои:10.1038/nature14685. PMID 26222030. S2CID 4468690.
- ^ Noguchi Y, Yuan Z, Bai L, Schneider S, Zhao G, Stillman B, Speck C, Li H (November 2017). "Cryo-EM structure of Mcm2-7 double hexamer on DNA suggests a lagging-strand DNA extrusion model". Труды Национальной академии наук Соединенных Штатов Америки. 114 (45): E9529–E9538. Дои:10.1073/pnas.1712537114. ЧВК 5692578. PMID 29078375.
- ^ а б Dutta A, Bell SP (1997). "Initiation of DNA replication in eukaryotic cells". Ежегодный обзор клеточной биологии и биологии развития. 13: 293–332. Дои:10.1146/annurev.cellbio.13.1.293. PMID 9442876.
- ^ а б Labib K, Tercero JA, Diffley JF (June 2000). "Uninterrupted MCM2-7 function required for DNA replication fork progression". Science. 288 (5471): 1643–7. Bibcode:2000Sci...288.1643L. Дои:10.1126/science.288.5471.1643. PMID 10834843.
- ^ Schwacha A, Bell SP (November 2001). "Interactions between two catalytically distinct MCM subgroups are essential for coordinated ATP hydrolysis and DNA replication". Молекулярная клетка. 8 (5): 1093–104. Дои:10.1016/S1097-2765(01)00389-6. PMID 11741544.
- ^ Boyer PD (January 1993). "The binding change mechanism for ATP synthase--some probabilities and possibilities". Biochimica et Biophysica Acta (BBA) - Биоэнергетика. 1140 (3): 215–50. Дои:10.1016/0005-2728(93)90063-l. PMID 8417777.
- ^ Hingorani MM, Washington MT, Moore KC, Patel SS (May 1997). "The dTTPase mechanism of T7 DNA helicase resembles the binding change mechanism of the F1-ATPase". Труды Национальной академии наук Соединенных Штатов Америки. 94 (10): 5012–7. Bibcode:1997PNAS...94.5012H. Дои:10.1073/pnas.94.10.5012. ЧВК 24622. PMID 9144181.
- ^ Yan H, Merchant AM, Tye BK (November 1993). "Cell cycle-regulated nuclear localization of MCM2 and MCM3, which are required for the initiation of DNA synthesis at chromosomal replication origins in yeast". Гены и развитие. 7 (11): 2149–60. Дои:10.1101/gad.7.11.2149. PMID 8224843.
- ^ Young MR, Suzuki K, Yan H, Gibson S, Tye BK (October 1997). "Nuclear accumulation of Saccharomyces cerevisiae Mcm3 is dependent on its nuclear localization sequence". Гены в клетки. 2 (10): 631–43. Дои:10.1046/j.1365-2443.1997.1510349.x. PMID 9427284.
- ^ Labib K, Diffley JF, Kearsey SE (November 1999). "G1-phase and B-type cyclins exclude the DNA-replication factor Mcm4 from the nucleus". Природа клеточной биологии. 1 (7): 415–22. Дои:10.1038/15649. PMID 10559985. S2CID 23407351.
- ^ Lei M, Tye BK (April 2001). "Initiating DNA synthesis: from recruiting to activating the MCM complex". Journal of Cell Science. 114 (Pt 8): 1447–54. PMID 11282021.
- ^ Leatherwood J (December 1998). "Emerging mechanisms of eukaryotic DNA replication initiation". Текущее мнение в области клеточной биологии. 10 (6): 742–8. Дои:10.1016/S0955-0674(98)80117-8. PMID 9914182.
- ^ а б Mimura S, Takisawa H (October 1998). "Xenopus Cdc45-dependent loading of DNA polymerase alpha onto chromatin under the control of S-phase Cdk". Журнал EMBO. 17 (19): 5699–707. Дои:10.1093/emboj/17.19.5699. ЧВК 1170898. PMID 9755170.
- ^ а б Zou L, Stillman B (April 1998). "Formation of a preinitiation complex by S-phase cyclin CDK-dependent loading of Cdc45p onto chromatin". Science. 280 (5363): 593–6. Bibcode:1998Sci...280..593Z. Дои:10.1126/science.280.5363.593. PMID 9554851.
- ^ а б c Mimura S, Masuda T, Matsui T, Takisawa H (June 2000). "Central role for cdc45 in establishing an initiation complex of DNA replication in Xenopus egg extracts". Гены в клетки. 5 (6): 439–52. Дои:10.1046/j.1365-2443.2000.00340.x. PMID 10886370.
- ^ а б c Aparicio OM, Weinstein DM, Bell SP (October 1997). "Components and dynamics of DNA replication complexes in S. cerevisiae: redistribution of MCM proteins and Cdc45p during S phase". Клетка. 91 (1): 59–69. Дои:10.1016/S0092-8674(01)80009-X. PMID 9335335. S2CID 10353164.
- ^ Tercero JA, Labib K, Diffley JF (May 2000). "DNA synthesis at individual replication forks requires the essential initiation factor Cdc45p". Журнал EMBO. 19 (9): 2082–93. Дои:10.1093/emboj/19.9.2082. ЧВК 305696. PMID 10790374.
- ^ Hennessy KM, Lee A, Chen E, Botstein D (June 1991). "A group of interacting yeast DNA replication genes". Гены и развитие. 5 (6): 958–69. Дои:10.1101/gad.5.6.958. PMID 2044962.
- ^ Aparicio OM, Stout AM, Bell SP (August 1999). "Differential assembly of Cdc45p and DNA polymerases at early and late origins of DNA replication". Труды Национальной академии наук Соединенных Штатов Америки. 96 (16): 9130–5. Bibcode:1999PNAS...96.9130A. Дои:10.1073/pnas.96.16.9130. ЧВК 17744. PMID 10430907.
- ^ Zou L, Stillman B (May 2000). "Assembly of a complex containing Cdc45p, replication protein A, and Mcm2p at replication origins controlled by S-phase cyclin-dependent kinases and Cdc7p-Dbf4p kinase". Молекулярная и клеточная биология. 20 (9): 3086–96. Дои:10.1128/mcb.20.9.3086-3096.2000. ЧВК 85601. PMID 10757793.
- ^ а б Walter J, Newport J (April 2000). "Initiation of eukaryotic DNA replication: origin unwinding and sequential chromatin association of Cdc45, RPA, and DNA polymerase alpha". Молекулярная клетка. 5 (4): 617–27. Дои:10.1016/S1097-2765(00)80241-5. PMID 10882098.
- ^ а б c Gambus A, Jones RC, Sanchez-Diaz A, Kanemaki M, van Deursen F, Edmondson RD, Labib K (April 2006). "GINS maintains association of Cdc45 with MCM in replisome progression complexes at eukaryotic DNA replication forks". Природа клеточной биологии. 8 (4): 358–66. Дои:10.1038/ncb1382. PMID 16531994. S2CID 21543095.
- ^ а б Kanemaki M, Sanchez-Diaz A, Gambus A, Labib K (June 2003). "Functional proteomic identification of DNA replication proteins by induced proteolysis in vivo". Природа. 423 (6941): 720–4. Bibcode:2003Natur.423..720K. Дои:10.1038/nature01692. PMID 12768207. S2CID 4345091.
- ^ Makarova KS, Wolf YI, Mekhedov SL, Mirkin BG, Koonin EV (2005). "Ancestral paralogs and pseudoparalogs and their role in the emergence of the eukaryotic cell". Исследования нуклеиновых кислот. 33 (14): 4626–38. Дои:10.1093/nar/gki775. ЧВК 1187821. PMID 16106042.
- ^ Moyer SE, Lewis PW, Botchan MR (July 2006). "Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase". Труды Национальной академии наук Соединенных Штатов Америки. 103 (27): 10236–10241. Bibcode:2006PNAS..10310236M. Дои:10.1073/pnas.0602400103. ЧВК 1482467. PMID 16798881.
- ^ Bochman ML, Schwacha A (July 2008). "The Mcm2-7 complex has in vitro helicase activity". Молекулярная клетка. 31 (2): 287–93. Дои:10.1016/j.molcel.2008.05.020. PMID 18657510.
- ^ Costa A, Ilves I, Tamberg N, Petojevic T, Nogales E, Botchan MR, Berger JM (April 2011). "The structural basis for MCM2-7 helicase activation by GINS and Cdc45". Структурная и молекулярная биология природы. 18 (4): 471–7. Дои:10.1038/nsmb.2004. ЧВК 4184033. PMID 21378962.
- ^ Yuan Z, Bai L, Sun J, Georgescu R, Liu J, O'Donnell ME, Li H (March 2016). "Structure of the eukaryotic replicative CMG helicase suggests a pumpjack motion for translocation". Структурная и молекулярная биология природы. 23 (3): 217–24. Дои:10.1038/nsmb.3170. ЧВК 4812828. PMID 26854665.
- ^ Homesley L, Lei M, Kawasaki Y, Sawyer S, Christensen T, Tye BK (April 2000). "Mcm10 and the MCM2-7 complex interact to initiate DNA synthesis and to release replication factors from origins". Гены и развитие. 14 (8): 913–26. Дои:10.1101/gad.14.8.913 (неактивно 01.09.2020). ЧВК 316538. PMID 10783164.CS1 maint: DOI неактивен по состоянию на сентябрь 2020 г. (связь)
- ^ Кристенсен Т.В., Тай Б.К. (июнь 2003 г.). «Drosophila MCM10 взаимодействует с членами пререпликационного комплекса и необходим для правильной конденсации хромосом». Молекулярная биология клетки. 14 (6): 2206–15. Дои:10.1091 / mbc.e02-11-0706. ЧВК 194871. PMID 12808023.
- ^ Ли С., Лячко И., Бутен Р., Кельман З., Тай Б.К. (январь 2010 г.). «Альтернативные механизмы координации полимеразы альфа и геликазы MCM». Молекулярная и клеточная биология. 30 (2): 423–35. Дои:10.1128 / MCB.01240-09. ЧВК 2798462. PMID 19917723.
- ^ ван Дерсен Ф., Сенгупта С., Де Пикколи Дж., Санчес-Диас А., Лабиб К. (май 2012 г.). «Mcm10 связывается с загруженной ДНК-геликазой в источниках репликации и определяет новый этап ее активации». Журнал EMBO. 31 (9): 2195–206. Дои:10.1038 / emboj.2012.69. ЧВК 3343467. PMID 22433841.
- ^ Масаи Х, Сато Н., Такеда Т., Араи К. (декабрь 1999 г.). «Киназный комплекс CDC7 как молекулярный переключатель репликации ДНК». Границы биологических наук. 4 (1–3): D834-40. Дои:10.2741 / masai. PMID 10577390.
- ^ Цзян В., Уэллс, штат Нью-Джерси, Хантер Т. (май 1999 г.). «Многоступенчатая регуляция репликации ДНК посредством Cdk фосфорилирования HsCdc6». Труды Национальной академии наук Соединенных Штатов Америки. 96 (11): 6193–8. Bibcode:1999PNAS ... 96.6193J. Дои:10.1073 / пнас.96.11.6193. ЧВК 26858. PMID 10339564.
- ^ а б Цзян В., Макдональд Д., Хоуп Т.Дж., Хантер Т. (октябрь 1999 г.). «Комплекс протеинкиназы Cdc7-Dbf4 млекопитающих необходим для инициации репликации ДНК». Журнал EMBO. 18 (20): 5703–13. Дои:10.1093 / emboj / 18.20.5703. ЧВК 1171637. PMID 10523313.
- ^ Кумагаи Х., Сато Н., Ямада М., Махони Д., Сегеззи В., Лис Е., Араи К., Масаи Х. (июль 1999 г.). «Новый белок, регулируемый ростом и клеточным циклом, ASK, активирует человеческую Cdc7-родственную киназу и необходим для перехода G1 / S в клетках млекопитающих». Молекулярная и клеточная биология. 19 (7): 5083–95. Дои:10.1128 / MCB.19.7.5083. ЧВК 84351. PMID 10373557.
- ^ Лей М., Кавасаки Ю., Янг М.Р., Кихара М., Сугино А., Тай Б.К. (декабрь 1997 г.). «Mcm2 является мишенью регуляции Cdc7-Dbf4 во время инициации синтеза ДНК». Гены и развитие. 11 (24): 3365–74. Дои:10.1101 / gad.11.24.3365. ЧВК 316824. PMID 9407029.
- ^ Харди С.Ф., Дрига О., Сеематтер С., Пал П.М., Склафани Р.А. (апрель 1997 г.). "mcm5 / cdc46-bob1 обходит требования для активатора S фазы Cdc7p". Труды Национальной академии наук Соединенных Штатов Америки. 94 (7): 3151–5. Bibcode:1997PNAS ... 94.3151H. Дои:10.1073 / пнас.94.7.3151. ЧВК 20337. PMID 9096361.
- ^ Камимура Ю., Так Ю.С., Сугино А., Араки Х. (апрель 2001 г.). «Sld3, который взаимодействует с Cdc45 (Sld4), функционирует для репликации хромосомной ДНК в Saccharomyces cerevisiae». Журнал EMBO. 20 (8): 2097–107. Дои:10.1093 / emboj / 20.8.2097. ЧВК 125422. PMID 11296242.
- ^ Канемаки М., Лабиб К. (апрель 2006 г.). «Различная роль Sld3 и GINS во время создания и развития вилок репликации ДНК эукариот». Журнал EMBO. 25 (8): 1753–63. Дои:10.1038 / sj.emboj.7601063. ЧВК 1440835. PMID 16601689.
- ^ Масумото Х., Сугино А., Араки Х. (апрель 2000 г.). «Dpb11 контролирует ассоциацию между ДНК-полимеразами альфа и эпсилон и автономно реплицирующейся областью последовательности почкующихся дрожжей». Молекулярная и клеточная биология. 20 (8): 2809–17. Дои:10.1128 / mcb.20.8.2809-2817.2000. ЧВК 85497. PMID 10733584.
- ^ Араки Х., Лим С.Х., Фонгдара А., Сугино А. (декабрь 1995 г.). «Dpb11, который взаимодействует с ДНК-полимеразой II (эпсилон) в Saccharomyces cerevisiae, играет двойную роль в прогрессии S-фазы и в контрольной точке клеточного цикла». Труды Национальной академии наук Соединенных Штатов Америки. 92 (25): 11791–5. Дои:10.1073 / пнас.92.25.11791. ЧВК 40488. PMID 8524850.
- ^ Гловер Дж. Н., Уильямс Р. С., Ли М. С. (ноябрь 2004 г.). «Взаимодействие между повторами BRCT и фосфопротеинами: запутано надвое». Тенденции в биохимических науках. 29 (11): 579–85. Дои:10.1016 / j.tibs.2004.09.010. PMID 15501676.
- ^ Масумото Х., Мурамацу С., Камимура Й., Араки Х. (февраль 2002 г.). «S-Cdk-зависимое фосфорилирование Sld2, необходимое для репликации хромосомной ДНК у почкующихся дрожжей». Природа. 415 (6872): 651–5. Bibcode:2002Натура.415..651М. Дои:10.1038 / природа713. PMID 11807498. S2CID 4300863.
- ^ Такаяма Ю., Камимура Ю., Окава М., Мурамацу С., Сугино А., Араки Н. (май 2003 г.). «GINS, новый мультибелковый комплекс, необходимый для репликации хромосомной ДНК у почкующихся дрожжей». Гены и развитие. 17 (9): 1153–65. Дои:10.1101 / gad.1065903. ЧВК 196052. PMID 12730134.
- ^ Лабиб К., Гамбус А. (июнь 2007 г.). «Ключевая роль комплекса GINS в вилках репликации ДНК». Тенденции в клеточной биологии. 17 (6): 271–8. Дои:10.1016 / j.tcb.2007.04.002. PMID 17467990.
- ^ Мурамацу С., Хираи К., Так Ю.С., Камимура Ю., Араки Х. (март 2010 г.). «CDK-зависимое образование комплекса между репликационными белками Dpb11, Sld2, Pol (epsilon} и GINS у почкующихся дрожжей». Гены и развитие. 24 (6): 602–12. Дои:10.1101 / gad.1883410. ЧВК 2841337. PMID 20231317.
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (июль 1958 г.). «Ферментативный синтез дезоксирибонуклеиновой кислоты. I. Приготовление субстратов и частичная очистка фермента от Escherichia coli». Журнал биологической химии. 233 (1): 163–70. PMID 13563462.
- ^ Алани Э., Трешер Р., Гриффит Дж. Д., Колоднер Р. Д. (сентябрь 1992 г.). «Характеристика свойств ДНК-связывания и стимуляции обмена цепей y-RPA, дрожжевого однонитевого ДНК-связывающего белка». Журнал молекулярной биологии. 227 (1): 54–71. Дои:10.1016/0022-2836(92)90681-9. PMID 1522601.
- ^ Гулиан М., Ричардс С.Х., Херд С.Дж., Бигсби Б.М. (октябрь 1990 г.). «Прерывистый синтез ДНК очищенными белками млекопитающих». Журнал биологической химии. 265 (30): 18461–71. PMID 2170412.
- ^ Маккаллох С.Д., Кункель Т.А. (январь 2008 г.). «Верность синтеза ДНК эукариотическими полимеразами репликативного и транслезионного синтеза». Клеточные исследования. 18 (1): 148–61. Дои:10.1038 / кр.2008.4. ЧВК 3639319. PMID 18166979.
- ^ Pursell ZF, Isoz I, Lundström EB, Johansson E, Kunkel TA (июль 2007 г.). «Дрожжевая ДНК-полимераза эпсилон участвует в репликации ведущей цепи ДНК». Наука. 317 (5834): 127–30. Bibcode:2007Научный ... 317..127П. Дои:10.1126 / science.1144067. ЧВК 2233713. PMID 17615360.
- ^ а б c d е Уотсон Дж., Бейкер Т., Белл С., Ганн А., Левин М., Лосик Р., Молекулярная биология гена 6-е издание, Pearson Education Inc., 2008 г. ISBN 080539592X
- ^ Waga S, Bauer G, Stillman B (апрель 1994 г.). «Восстановление полной репликации ДНК SV40 с очищенными факторами репликации». Журнал биологической химии. 269 (14): 10923–34. PMID 8144677.
- ^ Гарг П., Стит К.М., Сабури Н., Йоханссон Э., Бургерс П.М. (ноябрь 2004 г.). «Холостой ход из-за дельта-ДНК-полимеразы сохраняет лигируемый разрыв во время репликации ДНК отстающей цепи». Гены и развитие. 18 (22): 2764–73. Дои:10.1101 / гад.1252304. ЧВК 528896. PMID 15520275.
- ^ Burgers PM (февраль 2009 г.). «Динамика полимеразы на вилке репликации эукариотической ДНК». Журнал биологической химии. 284 (7): 4041–5. Дои:10.1074 / jbc.R800062200. ЧВК 2640984. PMID 18835809.
- ^ Джин Ю.Х., Айягари Р., Резник М.А., Горденин Д.А., Бургерс П.М. (январь 2003 г.). «Созревание фрагмента Окадзаки в дрожжах. II. Взаимодействие между полимеразой и 3'-5'-экзонуклеазной активностью Pol delta в создании лигируемого разрыва». Журнал биологической химии. 278 (3): 1626–33. Дои:10.1074 / jbc.M209803200. PMID 12424237.
- ^ Сого Дж. М., Лопес М., Фойани М. (июль 2002 г.). «Реверс вилки и накопление оцДНК на остановившихся вилках репликации из-за дефектов контрольной точки». Наука. 297 (5581): 599–602. Bibcode:2002Sci ... 297..599S. Дои:10.1126 / science.1074023. PMID 12142537. S2CID 33502697.
- ^ Моррисон А., Араки Х., Кларк А.Б., Хаматаке Р.К., Сугино А. (сентябрь 1990 г.). «Третья важная ДНК-полимераза в S. cerevisiae». Клетка. 62 (6): 1143–51. Дои:10.1016 / 0092-8674 (90) 90391-Q. PMID 2169349. S2CID 29672985.
- ^ Waga S, Стиллман B (май 1994 г.). «Анатомия вилки репликации ДНК, выявленная при восстановлении репликации ДНК SV40 in vitro». Природа. 369 (6477): 207–12. Bibcode:1994Натура.369..207Вт. Дои:10.1038 / 369207a0. PMID 7910375. S2CID 4351628.
- ^ Цуримото Т., Стиллман Б. (январь 1991 г.). «Факторы репликации, необходимые для репликации ДНК SV40 in vitro. II. Переключение ДНК-полимеразы альфа и дельта во время инициации синтеза ведущей и отстающей цепи». Журнал биологической химии. 266 (3): 1961–8. PMID 1671046.
- ^ Барри ER, Bell SD (декабрь 2006 г.). «Репликация ДНК в архее». Обзоры микробиологии и молекулярной биологии. 70 (4): 876–87. Дои:10.1128 / MMBR.00029-06. ЧВК 1698513. PMID 17158702.
- ^ Берг Дж. М., Тимочко Дж. Л., Страйер Л. (2003). Биохимия. Гейдельберг / Берлин: Springer.
- ^ а б Джонсон Р. Э., Классен Р., Пракаш Л., Пракаш С. (июль 2015 г.). «Основная роль ДНК-полимеразы δ в репликации как ведущих, так и отстающих нитей ДНК». Молекулярная клетка. 59 (2): 163–175. Дои:10.1016 / j.molcel.2015.05.038. ЧВК 4517859. PMID 26145172.
- ^ Бьюн Т.С., Пацек М., Йи М.К., Уолтер Дж.С., Симприч К.А. (май 2005 г.). «Функциональное разделение активности геликазы MCM и ДНК-полимеразы активирует ATR-зависимую контрольную точку». Гены и развитие. 19 (9): 1040–52. Дои:10.1101 / gad.1301205. ЧВК 1091739. PMID 15833913.
- ^ а б Ремус Д., Беурон Ф., Толун Дж., Гриффит Дж. Д., Моррис ЕР, Диффли Дж. Ф. (ноябрь 2009 г.). «Согласованная загрузка двойных гексамеров Mcm2-7 вокруг ДНК во время лицензирования репликации ДНК». Клетка. 139 (4): 719–30. Дои:10.1016 / j.cell.2009.10.015. ЧВК 2804858. PMID 19896182.
- ^ Эврин С., Кларк П., Зеч Дж., Лурц Р., Сан Дж., Уле С., Ли Х., Стиллман Б., Спек С. (декабрь 2009 г.). «Двойной гексамерный комплекс MCM2-7 загружается в исходную ДНК во время лицензирования репликации эукариотической ДНК». Труды Национальной академии наук Соединенных Штатов Америки. 106 (48): 20240–5. Bibcode:2009PNAS..10620240E. Дои:10.1073 / pnas.0911500106. ЧВК 2787165. PMID 19910535.
- ^ а б Мойер С.Е., Льюис П.В., Ботчан М.Р. (июль 2006 г.). «Выделение комплекса Cdc45 / Mcm2-7 / GINS (CMG), кандидата на геликазу вилки репликации эукариотической ДНК». Труды Национальной академии наук Соединенных Штатов Америки. 103 (27): 10236–10241. Bibcode:2006ПНАС..10310236М. Дои:10.1073 / pnas.0602400103. ЧВК 1482467. PMID 16798881.
- ^ Лабиб К. (июнь 2010 г.). «Как Cdc7 и циклин-зависимые киназы запускают начало репликации хромосом в эукариотических клетках?». Гены и развитие. 24 (12): 1208–19. Дои:10.1101 / гад.1933010. ЧВК 2885657. PMID 20551170.
- ^ Пацек М., Уолтер Дж. К. (сентябрь 2004 г.). «Необходимость MCM7 и Cdc45 в раскручивании хромосом во время репликации эукариотической ДНК». Журнал EMBO. 23 (18): 3667–76. Дои:10.1038 / sj.emboj.7600369. ЧВК 517609. PMID 15329670.
- ^ Яо Нью-Йорк, О'Доннелл М. (июнь 2010 г.). "SnapShot: Реплисом". Клетка. 141 (6): 1088–1088.e1. Дои:10.1016 / j.cell.2010.05.042. ЧВК 4007198. PMID 20550941.
- ^ Коста А., Ильвес И., Тамберг Н., Петоевич Т., Ногалес Е., Ботчан М. Р., Бергер Дж. М. (апрель 2011 г.). "Структурная основа активации геликазы MCM2-7 GINS и Cdc45". Структурная и молекулярная биология природы. 18 (4): 471–7. Дои:10.1038 / nsmb.2004. ЧВК 4184033. PMID 21378962.
- ^ Бермудес В.П., Фарина А., Таппин И., Гурвиц Дж. (Март 2010 г.). «Влияние фактора установления сплоченности человека Ctf4 / AND-1 на репликацию ДНК». Журнал биологической химии. 285 (13): 9493–505. Дои:10.1074 / jbc.M109.093609. ЧВК 2843200. PMID 20089864.
- ^ Чжу В., Укомаду С., Джа С., Сенга Т., Дхар С.К., Вольшлегель Дж.А., Натт Л.К., Корнблут С., Датта А. (сентябрь 2007 г.). «Mcm10 и And-1 / CTF4 рекрутируют ДНК-полимеразу альфа на хроматин для инициации репликации ДНК». Гены и развитие. 21 (18): 2288–99. Дои:10.1101 / gad.1585607. ЧВК 1973143. PMID 17761813.
- ^ Лу Х, Комата М., Катоу Й, Гуань З., Рейс К.С., Бадд М., Ширахиге К., Кэмпбелл Д.Л. (октябрь 2008 г.). «Mrc1 и ДНК-полимераза-эпсилон действуют вместе, связывая репликацию ДНК и контрольную точку S-фазы». Молекулярная клетка. 32 (1): 106–17. Дои:10.1016 / j.molcel.2008.08.020. ЧВК 2699584. PMID 18851837.
- ^ Петерманн Э., Хелледей Т., Калдекотт К.В. (июнь 2008 г.). «Claspin способствует нормальной скорости репликационной вилки в клетках человека». Молекулярная биология клетки. 19 (6): 2373–8. Дои:10.1091 / mbc.E07-10-1035. ЧВК 2397295. PMID 18353973.
- ^ Браво Р., Франк Р., Бланделл ПА, Макдональд-Браво Х (1987). «Циклин / PCNA - вспомогательный белок ДНК-полимеразы-дельта». Природа. 326 (6112): 515–7. Bibcode:1987Натура.326..515Б. Дои:10.1038 / 326515a0. PMID 2882423. S2CID 4344147.
- ^ Прелич Г., Тан С.К., Костура М., Мэтьюз МБ, Со А.Г., Дауни К.М., Стиллман Б. (1987). «Функциональная идентичность ядерного антигена пролиферирующих клеток и вспомогательного белка ДНК-полимеразы-дельта». Природа. 326 (6112): 517–20. Bibcode:1987Натура.326..517P. Дои:10.1038 / 326517a0. PMID 2882424. S2CID 4345171.
- ^ Молдавский Г.Л., Пфандер Б., Йенч С. (май 2007 г.). «PCNA, маэстро репликационной вилки». Клетка. 129 (4): 665–79. Дои:10.1016 / j.cell.2007.05.003. PMID 17512402. S2CID 3547069.
- ^ Цай Дж, Яо Н., Гиббс Э., Финкельштейн Дж., Филлипс Б., О'Доннелл М., Гурвиц Дж. (Сентябрь 1998 г.). «Гидролиз АТФ, катализируемый фактором репликации С человека, требует участия нескольких субъединиц». Труды Национальной академии наук Соединенных Штатов Америки. 95 (20): 11607–12. Bibcode:1998PNAS ... 9511607C. Дои:10.1073 / pnas.95.20.11607. ЧВК 21688. PMID 9751713.
- ^ Цуримото Т., Стиллман Б. (январь 1991 г.). «Факторы репликации, необходимые для репликации ДНК SV40 in vitro. I. Специфическое для структуры ДНК распознавание соединения праймер-матрица эукариотическими ДНК-полимеразами и их вспомогательными белками». Журнал биологической химии. 266 (3): 1950–60. PMID 1671045.
- ^ Podust LM, Podust VN, Sogo JM, Hübscher U (июнь 1995 г.). «Вспомогательные белки ДНК-полимеразы млекопитающих: анализ нагрузки ядерного антигена пролиферирующих клеток, катализируемой фактором репликации C, на кольцевую двухцепочечную ДНК». Молекулярная и клеточная биология. 15 (6): 3072–81. Дои:10.1128 / MCB.15.6.3072. ЧВК 230538. PMID 7760803.
- ^ Чжан Г., Гиббс Э., Кельман З., О'Доннелл М., Гурвиц Дж. (Март 1999 г.). «Исследования взаимодействий между фактором репликации С человека и ядерным антигеном пролиферирующих клеток человека». Труды Национальной академии наук Соединенных Штатов Америки. 96 (5): 1869–74. Bibcode:1999PNAS ... 96.1869Z. Дои:10.1073 / pnas.96.5.1869. ЧВК 26703. PMID 10051561.
- ^ а б Яо Нью-Йорк, Джонсон А., Боумен Г. Д., Куриян Дж., О'Доннелл М. (июнь 2006 г.). «Механизм раскрытия зажима ядерного антигена пролиферирующих клеток фактором репликации C». Журнал биологической химии. 281 (25): 17528–39. Дои:10.1074 / jbc.M601273200. PMID 16608854.
- ^ а б c Сабатинос, С.А. (2010). «Восстановление остановившейся вилки репликации». Scitable Nature Education. 3 (9): 31.
- ^ а б Ротштейн Р., Мишель Б., Ганглофф С. (январь 2000 г.). «Пауза и рекомбинация репликационной вилки» или «Сделайте паузу»"". Гены и развитие. 14 (1): 1–10. PMID 10640269.
- ^ Бастия Д., Цзаман С., Крингс Г., Саксена М., Пэн Х, Гринберг М.М. (сентябрь 2008 г.). «Механизм прекращения репликации, выявленный посредством Tus-опосредованной полярной остановки скользящей геликазы». Труды Национальной академии наук Соединенных Штатов Америки. 105 (35): 12831–6. Bibcode:2008ПНАС..10512831Б. Дои:10.1073 / pnas.0805898105. ЧВК 2529109. PMID 18708526.
- ^ Фабрики репликации, Павел Хозак, Питер Кук, Тенденции в клеточной биологии, том 4, выпуск 2; Научное направление, DOI
- ^ МакГарри Т.Дж., Киршнер М.В. (июнь 1998 г.). «Геминин, ингибитор репликации ДНК, разрушается во время митоза». Клетка. 93 (6): 1043–53. Дои:10.1016 / S0092-8674 (00) 81209-X. PMID 9635433. S2CID 235485.
- ^ de Klein A, Muijtjens M, van Os R, Verhoeven Y, Smit B, Carr AM, Lehmann AR, Hoeijmakers JH (апрель 2000 г.). «Целенаправленное нарушение ATR гена контрольной точки клеточного цикла приводит к ранней эмбриональной летальности у мышей». Текущая биология. 10 (8): 479–82. Дои:10.1016 / S0960-9822 (00) 00447-4. PMID 10801416. S2CID 15401622.
- ^ Кортез Д., Гунтуку С., Цинь Дж., Элледж С.Дж. (ноябрь 2001 г.). «ATR и ATRIP: партнеры в сигнализации контрольных точек». Наука. 294 (5547): 1713–6. Bibcode:2001Sci ... 294.1713C. Дои:10.1126 / science.1065521. PMID 11721054. S2CID 32810119.
- ^ Дарт Д.А., Адамс К.Э., Акерман И., Лакин Н.Д. (апрель 2004 г.). «Рекрутирование ATR киназы контрольной точки клеточного цикла на хроматин во время S-фазы». Журнал биологической химии. 279 (16): 16433–40. Дои:10.1074 / jbc.M314212200. PMID 14871897.
- ^ Болл Х.Л., Майерс Дж. С., Кортез Д. (май 2005 г.). «Связывание ATRIP с репликационным белком A-одноцепочечной ДНК способствует локализации ATR-ATRIP, но не требуется для фосфорилирования Chk1». Молекулярная биология клетки. 16 (5): 2372–81. Дои:10.1091 / mbc.E04-11-1006. ЧВК 1087242. PMID 15743907.
- ^ Бермудес В.П., Линдси-Больц Л.А., Чезаре А.Дж., Манива Ю., Гриффит Дж. Д., Гурвиц Дж., Санкар А. (февраль 2003 г.). «Загрузка комплекса контрольных точек человека 9-1-1 на ДНК с помощью загрузчика контрольных точек hRad17-фактор репликации C in vitro». Труды Национальной академии наук Соединенных Штатов Америки. 100 (4): 1633–8. Bibcode:2003ПНАС..100.1633Б. Дои:10.1073 / pnas.0437927100. ЧВК 149884. PMID 12578958.
- ^ Zou L, Cortez D, Elledge SJ (январь 2002 г.). «Регулирование выбора субстрата ATR с помощью Rad17-зависимой загрузки комплексов Rad9 на хроматин». Гены и развитие. 16 (2): 198–208. Дои:10.1101 / gad.950302. ЧВК 155323. PMID 11799063.
- ^ Кумагай А., Данфи РГ (октябрь 2000 г.). «Claspin, новый белок, необходимый для активации Chk1 во время ответа контрольной точки репликации ДНК в экстрактах яиц Xenopus». Молекулярная клетка. 6 (4): 839–49. Дои:10.1016 / S1097-2765 (05) 00092-4. PMID 11090622.
- ^ Кумагай А., Данфи РГ (февраль 2003 г.). «Повторяющиеся фосфопептидные мотивы в Claspin опосредуют регулируемое связывание Chk1» (PDF). Природа клеточной биологии. 5 (2): 161–5. Дои:10.1038 / ncb921. PMID 12545175. S2CID 29331203.
- ^ Мяо Х., Сейлер Дж. А., Бурханс В. К. (февраль 2003 г.). «Регулирование происхождения репликации клеток и вирусов SV40 с помощью Chk1-зависимых внутренних и индуцированных УФ-излучением контрольных точек». Журнал биологической химии. 278 (6): 4295–304. Дои:10.1074 / jbc.M204264200. PMID 12424256.
- ^ Бар-Зив Р., Войчек Ю., Баркай Н. (сентябрь 2016 г.). «Динамика хроматина при репликации ДНК». Геномные исследования. 26 (9): 1245–56. Дои:10.1101 / гр.201244.115. ЧВК 5052047. PMID 27225843.
- ^ Люгер К., Мэдер А.В., Ричмонд Р.К., Сарджент Д.Ф., Ричмонд Т.Дж. (сентябрь 1997 г.). «Кристаллическая структура ядерной частицы нуклеосомы при разрешении 2,8 A». Природа. 389 (6648): 251–60. Bibcode:1997Натура.389..251Л. Дои:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Виттмейер Дж., Джосс Л., Формоза Т. (июль 1999 г.). «Spt16 и Pob3 из Saccharomyces cerevisiae образуют существенный, обильный гетеродимер, который является ядерным, ассоциированным с хроматином и соочищается с ДНК-полимеразой альфа». Биохимия. 38 (28): 8961–71. Дои:10.1021 / bi982851d. PMID 10413469.
- ^ Виттмейер Дж., Формоза Т. (июль 1997 г.). «Альфа-каталитическая субъединица ДНК-полимеразы Saccharomyces cerevisiae взаимодействует с Cdc68 / Spt16 и с Pob3, белком, подобным HMG1-подобному белку». Молекулярная и клеточная биология. 17 (7): 4178–90. Дои:10.1128 / MCB.17.7.4178. ЧВК 232271. PMID 9199353.
- ^ Xin H, Takahata S, Blanksma M, McCullough L, Stillman DJ, Formosa T (август 2009 г.). «yFACT обеспечивает глобальную доступность нуклеосомной ДНК без замещения H2A-H2B». Молекулярная клетка. 35 (3): 365–76. Дои:10.1016 / j.molcel.2009.06.024. ЧВК 2748400. PMID 19683499.
- ^ Groth A, Corpet A, Cook AJ, Roche D, Bartek J, Lukas J, Almouzni G (декабрь 2007 г.). «Регулирование прогрессии репликационной вилки через спрос и предложение на гистоны». Наука. 318 (5858): 1928–31. Bibcode:2007Научный ... 318.1928G. Дои:10.1126 / science.1148992. PMID 18096807. S2CID 22350778.
- ^ Даганзо С.М., Эрцбергер Дж. П., Лам В. М., Скордалакес Э., Чжан Р., Франко А. А., Брилл С. Дж., Адамс П. Д., Бергер Дж. М., Кауфман П. Д. (декабрь 2003 г.). «Структура и функция консервативного ядра белка отложения гистонов Asf1». Текущая биология. 13 (24): 2148–58. Дои:10.1016 / j.cub.2003.11.027. PMID 14680630. S2CID 15164132.
- ^ Войчек Ю., Бар-Зив Р., Баркай Н. (март 2016 г.). «Гомеостаз экспрессии при репликации ДНК». Наука. 351 (6277): 1087–90. Bibcode:2016Научный ... 351.1087V. Дои:10.1126 / science.aad1162. PMID 26941319. S2CID 32751800.
- ^ Стиллман Б. (май 1986 г.). «Сборка хроматина во время репликации ДНК SV40 in vitro». Клетка. 45 (4): 555–65. Дои:10.1016/0092-8674(86)90287-4. PMID 3011272. S2CID 21212160.
- ^ Шибахара К., Стиллман Б. (февраль 1999 г.). «Репликационно-зависимая маркировка ДНК с помощью PCNA способствует наследованию хроматина, связанному с CAF-1». Клетка. 96 (4): 575–85. Дои:10.1016 / S0092-8674 (00) 80661-3. PMID 10052459. S2CID 13912324.
- ^ Ролеф Бен-Шахар Т., Кастильо А.Г., Осборн М.Дж., Борден К.Л., Корнблатт Дж., Верро А. (декабрь 2009 г.). «Два принципиально различных пептида взаимодействия PCNA вносят вклад в функцию фактора сборки 1 хроматина». Молекулярная и клеточная биология. 29 (24): 6353–65. Дои:10.1128 / MCB.01051-09. ЧВК 2786881. PMID 19822659.
- ^ Хуанг С., Чжоу Х., Кацманн Д., Хохштрассер М., Атанасова Е., Чжан З. (сентябрь 2005 г.). «Rtt106p представляет собой гистоновый шаперон, участвующий в опосредованном гетерохроматином замалчивании». Труды Национальной академии наук Соединенных Штатов Америки. 102 (38): 13410–5. Bibcode:2005PNAS..10213410H. Дои:10.1073 / pnas.0506176102. ЧВК 1224646. PMID 16157874.
- ^ Ито Т., Тайлер Дж., Балджер М., Кобаяши Р., Кадонага Дж. Т. (октябрь 1996 г.). «АТФ-облегченная сборка хроматина с нуклеоплазмин-подобным белком из Drosophila melanogaster». Журнал биологической химии. 271 (40): 25041–8. Дои:10.1074 / jbc.271.40.25041. PMID 8798787.
- ^ Гассер Р., Коллер Т., Сого Дж. М. (май 1996 г.). «Стабильность нуклеосом на вилке репликации». Журнал молекулярной биологии. 258 (2): 224–39. Дои:10.1006 / jmbi.1996.0245. PMID 8627621.
- ^ Мезельсон М., Шталь Ф.В. (июль 1958 г.). «Репликация ДНК в Escherichia coli». Труды Национальной академии наук Соединенных Штатов Америки. 44 (7): 671–82. Bibcode:1958ПНАС ... 44..671М. Дои:10.1073 / pnas.44.7.671. ЧВК 528642. PMID 16590258.
- ^ Белл С.П., Кагуни Дж.М. (июнь 2013 г.). «Загрузка геликазы в хромосомных источниках репликации». Перспективы Колд-Спринг-Харбор в биологии. 5 (6): a010124. Дои:10.1101 / cshperspect.a010124. ЧВК 3660832. PMID 23613349.