Молекулярные модели ДНК - Molecular models of DNA


Молекулярные модели структур ДНК представляют собой молекулярная геометрия и топология дезоксирибонуклеиновой кислоты (ДНК ) молекул с использованием одного из нескольких способов с целью упрощения и представления основных, физических и химических свойств молекулярных структур ДНК, либо in vivo или же in vitro. Эти представления включают плотно упакованные сферы (CPK модели) из пластика, металлические провода для скелетные модели, графические вычисления и анимация на компьютере, художественный рендеринг. Компьютерные молекулярные модели также позволяют создавать анимацию и моделирование молекулярной динамики, что очень важно для понимания того, как функционирует ДНК. in vivo.
Более продвинутый компьютерный молекулярные модели ДНК вовлекают молекулярная динамика моделирование и квантовая механика расчеты вибровращений, делокализованные молекулярные орбитали (МО), электрические дипольные моменты, водородная связь, и так далее. Моделирование молекулярной динамики ДНК включает моделирование дезоксирибонуклеиновой кислоты (ДНК ) молекулярная геометрия и топология изменяется со временем в результате как внутри-, так и межмолекулярных взаимодействий ДНК. В то время как молекулярные модели молекул ДНК, такие как плотно упакованные сферы (модели CPK), сделанные из пластиковой или металлической проволоки для скелетные модели являются полезными представлениями статических структур ДНК, их полезность очень ограничена для представления сложной динамики ДНК. Компьютерное молекулярное моделирование позволяет создавать как анимацию, так и моделирование молекулярной динамики, что очень важно для понимания того, как функционирует ДНК. in vivo.
История

С самых ранних этапов структурных исследований ДНК дифракция рентгеновских лучей и биохимические методы, молекулярные модели, такие как Уотсон-Крика двойная спираль нуклеиновой кислоты модели были успешно использованы для решения «загадки» структуры ДНК, а также для выяснения того, как последняя связана с ее ключевыми функциями в живых клетках. О первых высококачественных рентгенограммах A-ДНК сообщил Розалинд Франклин и Раймонд Гослинг в 1953 г.[1] Розалинда Франклин сделала критическое наблюдение, что ДНК существует в двух различных формах, A и B, и дала наиболее четкие изображения обеих с помощью техники дифракции рентгеновских лучей.[2] Первые расчеты преобразования Фурье атомной спирали были опубликованы годом ранее Кокраном, Криком и Вандом:[3] а в 1953 г. последовало вычисление Криком преобразования Фурье спиральной катушки.[4]
Структурная информация генерируется из рентгеноструктурных исследований ориентированных волокон ДНК с помощью молекулярных моделей ДНК, которые сочетаются с кристаллографическим и математическим анализом рентгенограмм.
Первые сообщения о двойной спиральной молекулярной модели структуры B-ДНК были сделаны Джеймсом Уотсоном и Фрэнсисом Криком в 1953 году.[5][6] В том же году Морис Ф. Уилкинс, А. Стокс и Х.Р. Уилсон сообщили о первых рентгеновских снимках in vivo B-ДНК в частично ориентированных головках сперматозоидов лосося.[7]
Разработка первой правильной двойной спиральной молекулярной модели ДНК Криком и Ватсоном, возможно, была бы невозможна без биохимических доказательств спаривания нуклеотидных оснований ([A --- T]; [C --- G]) или Правила Чаргаффа.[8][9][10][11][12][13] Хотя такие первоначальные исследования структур ДНК с помощью молекулярных моделей были по существу статичными, их последствия для объяснения in vivo Функции ДНК были важны в областях биосинтеза белка и квазиуниверсальности генетического кода. Исследования эпигенетической трансформации ДНК in vivo однако развивались гораздо медленнее, несмотря на их важность для эмбриологии, морфогенеза и исследований рака. Такая химическая динамика и биохимические реакции ДНК намного сложнее, чем молекулярная динамика физических взаимодействий ДНК с водой, ионами и белками / ферментами в живых клетках.
Важность
Давняя постоянная динамическая проблема заключается в том, как «самовоспроизведение» ДНК происходит в живых клетках, что должно включать временное разворачивание сверхспиральных волокон ДНК. Хотя ДНК состоит из относительно жестких, очень больших удлиненных молекул биополимера, называемых волокна или цепочки (состоящие из повторяющихся нуклеотид единицы четырех основных типов, присоединенные к дезоксирибозной и фосфатной группам), его молекулярная структура in vivo претерпевает динамические изменения конфигурации, в которых участвуют динамически присоединенные молекулы и ионы воды. Суперспирализация, упаковка гистонами в хромосомных структурах и другие подобные супрамолекулярные аспекты также включают in vivo Топология ДНК который является даже более сложным, чем молекулярная геометрия ДНК, что делает молекулярное моделирование ДНК особенно сложной проблемой как для молекулярных биологов, так и для биотехнологов. Подобно другим большим молекулам и биополимерам, ДНК часто существует в нескольких стабильных геометриях (т. Е. Проявляет конформационная изомерия ) и конфигурационные, квантовые состояния, близкие друг к другу по энергии на поверхности потенциальной энергии молекулы ДНК.
Такую изменяющуюся молекулярную геометрию также можно вычислить, по крайней мере в принципе, с помощью ab initio квантовая химия методы, которые могут обеспечить высокую точность для малых молекул, хотя заявления о том, что приемлемая точность может быть также достигнута для полинуклеотидов и конформаций ДНК, были недавно сделаны на основе колебательный круговой дихроизм (VCD) спектральные данные. Такие квантовые геометрии определяют важный класс ab initio молекулярные модели ДНК, исследования которых только начинаются, особенно связанные с результатами, полученными с помощью VCD в растворах. Более подробные сравнения с такими ab initio квантовые вычисления, в принципе, можно получить с помощью спектроскопии 2D-FT ЯМР и релаксационных исследований растворов полинуклеотидов или специально меченой ДНК, например, дейтериевыми метками.
В интересном повороте ролей было предложено использовать молекулу ДНК для квантовые вычисления через ДНК. И наноструктуры ДНК, и ДНК-вычисления биочипы были построены.
Основные концепции
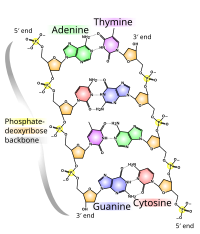
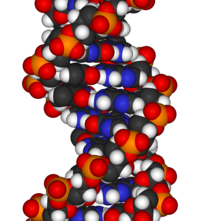
Химическая структура ДНК недостаточна для понимания сложности трехмерных структур ДНК. Напротив, анимированные молекулярные модели позволяют визуально исследовать трехмерную (3D) структуру ДНК. Показанная модель ДНК (крайняя справа) заполняет пространство, или CPK, модель двойной спирали ДНК. Анимированные молекулярные модели, такие как проволочный или скелетный тип, показанные в верхней части этой статьи, позволяют визуально исследовать трехмерную (3D) структуру ДНК. Другой тип модели ДНК - это модель заполнения пространства, или CPK.
Динамика водородных связей и протонный обмен сильно различаются на много порядков между двумя системами полностью гидратированной ДНК и молекул воды во льду. Таким образом, динамика ДНК сложна, включая наносекундные и несколько десятков пикосекундных временных масштабов, тогда как динамика жидкого льда находится в пикосекундной временной шкале, а динамика протонного обмена во льду - в миллисекундной временной шкале. Скорости обмена протонов в ДНК и прикрепленных белках могут варьироваться от пикосекунды до наносекунды, минут или лет, в зависимости от точного местоположения протонов, обменявшихся на большие биополимеры.
Простой гармонический осциллятор «вибрация» - это только упрощенное динамическое представление продольных колебаний переплетенных спиралей ДНК, которые, как было установлено, ангармонический а не гармонической, как это часто предполагается в квантовой динамическое моделирование ДНК.
Структура ДНК
Структура ДНК показывает разнообразие форм, как двухцепочечных, так и одноцепочечных. Механические свойства ДНК, которые напрямую связаны с ее структурой, представляют серьезную проблему для клетки. Каждый процесс, который связывает или читает ДНК может использовать или изменять механические свойства ДНК для распознавания, упаковки и модификации. Крайняя длина (a хромосома может содержать цепь ДНК длиной 10 см), относительная жесткость и спиральная структура ДНК привели к эволюция из гистоны и из ферменты Такие как топоизомеразы и геликасы управлять ДНК клетки. Свойства ДНК тесно связаны с ее молекулярной структурой и последовательностью, особенно со слабостью водородных связей и электронных взаимодействий, которые удерживают цепи ДНК вместе, по сравнению с силой связей внутри каждой цепи.
Экспериментальные методы, позволяющие напрямую измерить механические свойства ДНК, относительно новы, и визуализация с высоким разрешением в растворе часто затруднена. Тем не менее, ученые обнаружили большое количество данных о механических свойствах этого полимера, и влияние механических свойств ДНК на клеточные процессы является предметом активных текущих исследований.
ДНК, обнаруженная во многих клетках, может иметь макроскопическую длину: несколько сантиметров для каждой хромосомы человека. Следовательно, клетки должны уплотняться или упаковка ДНК несет это в себе. В эукариоты это осуществляется катушкой белки названный гистоны, вокруг которого наматывается ДНК. Дальнейшее уплотнение этого ДНК-белкового комплекса дает хорошо известный митотический эукариотический хромосомы.
В конце 1970-х гг. неспиральные модели структуры ДНК были кратко рассмотрены как потенциальное решение проблем в Репликация ДНК в плазмиды и хроматин. Однако модели были отложены в пользу модели с двойной спиралью из-за последующих экспериментальных достижений, таких как Рентгеновская кристаллография дуплексов ДНК, а позже ядерная частица нуклеосомы, и открытие топоизомеразы. Такие модели, отличные от двойной спирали, в настоящее время не принимаются основным научным сообществом.[14][15]
Определение структуры ДНК с помощью молекулярного моделирования и рентгенограмм ДНК


После того, как ДНК была отделена и очищена стандартными биохимическими методами, образец находится в сосуде, как на рисунке в верхней части этой статьи. Ниже приведены основные этапы генерации структурной информации на основе рентгеноструктурных исследований ориентированных волокон ДНК, взятых из гидратированного образца ДНК с помощью молекулярных моделей ДНК, которые объединены с кристаллографическим и математическим анализом рентгенограмм. .
Паракристаллические решетчатые модели структур B-ДНК

А паракристаллический решетка, или паракристалл, представляет собой молекулярную или атомную решетку со значительными количествами (например, более чем несколько процентов) частичного разупорядочения расположения молекул. Предельными случаями модели паракристалла являются наноструктуры, Такие как очки, жидкости и т. д., которые могут иметь только локальный порядок, но не глобальный. На следующем рисунке показан простой пример паракристаллической решетки кварцевого стекла:
Жидкие кристаллы также имеют паракристаллический, а не кристаллический конструкции.
Высокогидратированная B-ДНК встречается в живых клетках в таком паракристаллическом состоянии, которое является динамическим, несмотря на относительно жесткую двойную спираль ДНК, стабилизированную параллельными водородными связями между парами нуклеотидных оснований в двух комплементарных спиральных цепях ДНК (см. Рисунки). ). Для простоты в большинстве молекулярных моделей ДНК не учитываются как вода, так и ионы, динамически связанные с B-ДНК, и поэтому они менее полезны для понимания динамического поведения B-ДНК. in vivo. Физико-математический анализ рентгеновских лучей.[16][17] и спектроскопические данные для паракристаллической B-ДНК, таким образом, намного сложнее, чем данные для дифрактограмм рентгеновских лучей кристаллической A-ДНК. Модель паракристалла также важна для технологических приложений ДНК, таких как ДНК-нанотехнологии. В настоящее время также разрабатываются новые методы, сочетающие дифракцию рентгеновских лучей ДНК с рентгеновской микроскопией гидратированных живых клеток.[18]
Геномные и биотехнологические приложения молекулярного моделирования ДНК

Существуют различные варианты использования молекулярного моделирования ДНК в исследованиях в области геномики и биотехнологии, от восстановления ДНК до ПЦР и Наноструктуры ДНК. Двумерные массивы соединений ДНК были визуализированы Атомно-силовая микроскопия.[19]
Молекулярное моделирование ДНК находит различное применение в геномика и биотехнология, с исследовательскими приложениями, от восстановления ДНК до ПЦР и наноструктур ДНК. К ним относятся компьютерные молекулярные модели таких разнообразных молекул, как РНК-полимераза, E. coli, матрица бактериальной ДНК-примазы, предполагающая очень сложную динамику на границах раздела между ферментами и матрицей ДНК, а также молекулярные модели мутагенного химического взаимодействия мощных молекул канцерогенов. с ДНК. Все они представлены в галерее ниже.
Технологическое применение включает биочип ДНК и наноструктуры ДНК, предназначенные для ДНК-вычисления и другие динамические приложения ДНК-нанотехнологии.[20][21][22][23][24][25]Изображение справа - это самособирающиеся наноструктуры ДНК. Структура «плитки» ДНК на этом изображении состоит из четырех разветвленных стыков, ориентированных под углом 90 °. Каждая плитка состоит из девяти олигонуклеотидов ДНК, как показано; такие плитки служат в качестве основного «строительного блока» для сборки наночастиц ДНК, показанных на микрофотографии АСМ.
Квадруплексная ДНК может быть вовлечен в некоторые виды рака.[26][27] Изображения квадруплексов ДНК находятся в галерее ниже.
Галерея моделей ДНК

Родовая модель вращающейся ДНК.

Упрощенный набросок двойной спиральной структуры А-ДНК.

Модель репликации ДНК, основанная на концепции двойной спирали.

Анимированная, заполняющая пространство молекулярная модель двойной спирали A-ДНК

Масштабная модель ДНК Крика-Уотсона, представленная в Музее Принсипи Фелипе.

Вид сбоку на молекулярные модели A-, B-, Z-ДНК.

Упрощенная модель двойной спирали А-ДНК.

Молекулярное моделирование РНК-полимеразы.

Молекулярное моделирование матрицы примазы бактериальной ДНК.

Молекулярное моделирование взаимодействия ДНК с молекулой канцерогена MGMT.
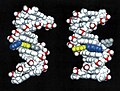
Трехмерная молекулярная модель ДНК, поврежденной канцерогенным 2-аминофлуореном (AF).
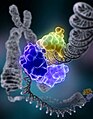
Рис.6. Молекулярное моделирование репарации ДНК

Анимированная скелетная модель А-ДНК.

Упрощенные модели хроматина.

Упрощенная модель структуры хромосом.

Гипотетический квадруплекс структур ДНК, богатых гуанином, которые могут быть вовлечены в рак.

Трехмерная молекулярная структура внутримолекулярного теломерного G-квадруплекса человека в растворе калия.

Молекулярная модель, заполняющая пространство ДНК

Модель сконструированного тетраэдра ДНК.

Модель ДНК длиной 15 м, Центр биоразнообразия Naturalis
Смотрите также
- G-квадруплекс
- Кристаллография
- Кристаллические решетки
- База данных свойств динуклеотидов (DiProDB ), предназначенный для сбора и анализа термодинамических, структурных и других динуклеотидных признаков.
- Рентгеновская микроскопия
- Рассеяние рентгеновских лучей
- Рассеяние нейтронов
- Последовательность нуклеиновой кислоты
- Вибрационный круговой дихроизм (VCD)
- Рамановская спектроскопия -микроскопия и когерентная антистоксова рамановская спектроскопия (ЛЕГКОВЫЕ АВТОМОБИЛИ)
- сэр Лоуренс Брэгг, ФРС
- Сравнение программного обеспечения для моделирования нуклеиновых кислот
- ЯНТАРЬ
- Очарование
- Abalone (молекулярная механика)
- Программное обеспечение для визуализации Sirius
- QMC @ Home
- ЯМР-спектроскопия (FT-ЯМР)
- ЯМР-визуализация микроскопия
- СВЧ спектроскопия
- FT-инфракрасный (ИК)
- FT-ближняя инфракрасная спектроскопия (NIR)
- Спектральная визуализация, гиперспектральное изображение, химическая визуализация
- Флуоресцентная корреляционная спектроскопия
- Флуоресцентная кросс-корреляционная спектроскопия и Фёрстеровский резонансный перенос энергии (FRET)
- Конфокальная микроскопия
Рекомендации
- ^ Франклин Р.Э., Гослинг Р.Г. (6 марта 1953 г.). «Структура волокон тимонуклеата натрия I. Влияние содержания воды». Acta Crystallogr. 6 (8): 673. Дои:10.1107 / S0365110X53001939.CS1 maint: несколько имен: список авторов (связь)
Франклин Р.Э., Гослинг Р.Г. (6 марта 1953 г.). "Структура тимонуклеатных волокон натрия II. Цилиндрически симметричная функция Паттерсона". Acta Crystallogr. 6 (8): 678. Дои:10.1107 / S0365110X53001940.CS1 maint: несколько имен: список авторов (связь) - ^ Хаммер, Дин Х .; Мэддокс, Бренда (2002). «Извилистая дорога к двойной спирали». Scientific American. 287 (6): 127–128. Bibcode:2002SciAm.287f.127H. Дои:10.1038 / scientificamerican1202-127. JSTOR 26060090.
- ^ Кокран, В., Крик, F.H.C., Ванд В. (1952). «Структура синтетических полипептидов. 1. Преобразование атомов на спирали». Acta Crystallogr. 5 (5): 581–6. Дои:10.1107 / S0365110X52001635.CS1 maint: несколько имен: список авторов (связь)
- ^ Крик, F.H.C. (1953а). "Преобразование Фурье спиральной катушки". Acta Crystallogr. 6 (8–9): 685–9. Дои:10.1107 / S0365110X53001952.
- ^ Уотсон, Джеймс Д., Крик, Фрэнсис Х.С. (25 апреля 1953 г.). «Структура нуклеиновой кислоты дезоксирибозы» (PDF). Природа. 171 (4356): 737–8. Bibcode:1953 г., природа. 171..737 Вт. Дои:10.1038 / 171737a0. PMID 13054692. S2CID 4253007.CS1 maint: несколько имен: список авторов (связь), .
- ^ Уотсон, Дж. Д.; Крик F.H.C. (1953b). «Структура ДНК». Симпозиумы Колд-Спринг-Харбор по количественной биологии. 18: 123–31. Дои:10.1101 / SQB.1953.018.01.020. PMID 13168976.
- ^ Уилкинс М.Х.Ф., А.Р. Стокс А. И Уилсон, Х.Р. (1953). «Молекулярная структура нуклеиновых кислот дезоксипентоза» (PDF). Природа. 171 (4356): 738–40. Bibcode:1953 г., природа. 171..738 Вт. Дои:10.1038 / 171738a0. PMID 13054693. S2CID 4280080.CS1 maint: несколько имен: список авторов (связь)
- ^ Элсон Д., Чаргафф Э (1952). «О содержании дезоксирибонуклеиновой кислоты в гаметах морского ежа». Experientia. 8 (4): 143–5. Дои:10.1007 / BF02170221. PMID 14945441. S2CID 36803326.
- ^ Чаргафф Э., Липшиц Р., Грин С. (1952). «Состав дезоксипентозных нуклеиновых кислот четырех родов морских ежей». J Biol Chem. 195 (1): 155–160. PMID 14938364.
- ^ Chargaff E, Lipshitz R, Green C, Hodes ME (1951). «Состав дезоксирибонуклеиновой кислоты спермы лосося». J Biol Chem. 192 (1): 223–230. PMID 14917668.
- ^ Чаргафф Э. (1951). «Некоторые недавние исследования состава и структуры нуклеиновых кислот». J Cell Physiol Suppl. 38 (Дополнение).
- ^ Magasanik B, Vischer E, Doniger R, Elson D, Chargaff E (1950). «Разделение и оценка рибонуклеотидов в малых количествах». J Biol Chem. 186 (1): 37–50. PMID 14778802.
- ^ Чаргафф Э. (1950). «Химическая специфичность нуклеиновых кислот и механизм их ферментативной деградации». Experientia. 6 (6): 201–9. Дои:10.1007 / BF02173653. PMID 15421335. S2CID 2522535.
- ^ Стокс, Т. Д. (1982). «Двойная спираль и покоробленная молния - образцовая сказка». Социальные исследования науки. 12 (2): 207–240. Дои:10.1177/030631282012002002. PMID 11620855. S2CID 29369576.
- ^ Гаутам, Н. (25 мая 2004 г.). «Ответ» на разнообразие вторичной структуры ДНК"" (PDF). Текущая наука. 86 (10): 1352–1353. Получено 25 мая 2012.
Однако открытие топоизомераз сняло «жало» в топологическом возражении против плектонемической двойной спирали. Более недавнее решение монокристаллической рентгеновской структуры ядерной частицы нуклеосомы показало почти 150 пар оснований ДНК (то есть около 15 полных витков) со структурой, которая во всех существенных отношениях аналогична модели Уотсона-Крика. . Это нанесло смертельный удар идее о том, что другие формы ДНК, особенно двойная спиральная ДНК, существуют как нечто иное, чем локальные или временные структуры.
[мертвая ссылка ] - ^ Хосеманн Р., Багчи Р.Н., Прямой анализ дифракции на веществе, North-Holland Publs., Амстердам - Нью-Йорк, 1962.
- ^ Баяну, И. (1978). «Рассеяние рентгеновских лучей частично неупорядоченными мембранными системами». Acta Crystallogr. А. 34 (5): 751–3. Bibcode:1978AcCrA..34..751B. Дои:10.1107 / S0567739478001540.
- ^ Ямамото Ю., Шинохара К. (октябрь 2002 г.). «Применение рентгеновской микроскопии в анализе живых гидратированных клеток». Анат. Rec. 269 (5): 217–23. Дои:10.1002 / ar.10166. PMID 12379938. S2CID 43009840.
- ^ Мао, Чэндэ; Солнце, Вэйцюн; Симан, Надриан К. (16 июня 1999 г.). "Разработанные двумерные массивы соединений Холлидея ДНК, визуализированные с помощью атомно-силовой микроскопии". Журнал Американского химического общества. 121 (23): 5437–43. Дои:10.1021 / ja9900398.
- ^ Robinson, Bruche H .; Симан, Надриан К. (август 1987 г.). «Дизайн биочипа: самоорганизующееся устройство памяти молекулярного масштаба». Белковая инженерия. 1 (4): 295–300. Дои:10.1093 / белок / 1.4.295. ISSN 0269-2139. PMID 3508280. Связь
- ^ Ротемунд, Пол В. К.; Экани-Нкодо, Аксель; Пападакис, Ник; Кумар, Ашиш; Файгенсон, Дебора Кучнир; Уинфри, Эрик (22 декабря 2004 г.). «Дизайн и характеристика программируемых ДНК-нанотрубок». Журнал Американского химического общества. 126 (50): 16344–52. Дои:10.1021 / ja044319l. PMID 15600335.
- ^ Керен, К .; Ротем С. Берман; Евгений Бухстаб; Ури Сиван; Эрез Браун (ноябрь 2003 г.). "Полевой транзистор на основе углеродных нанотрубок с ДНК-шаблоном". Наука. 302 (6549): 1380–2. Bibcode:2003Наука ... 302,1380K. CiteSeerX 10.1.1.454.2291. Дои:10.1126 / science.1091022. PMID 14631035. S2CID 29794525.
- ^ Чжэн, Цзивэнь; Константину, Памела Э .; Майкл, Кристина; Аливисатос, А. Пол; Kiehl, Richard A .; Симан Надриан К. (2006). «2D-массивы наночастиц демонстрируют организационную силу надежных мотивов ДНК». Нано буквы. 6 (7): 1502–4. Bibcode:2006NanoL ... 6.1502Z. Дои:10.1021 / nl060994c. ЧВК 3465979. PMID 16834438.
- ^ Коэн, Джастин Д .; Садовски, Джон П .; Дерван, Питер Б. (2007). «Обращение к одиночным молекулам на наноструктурах ДНК». Angewandte Chemie International Edition. 46 (42): 7956–9. Дои:10.1002 / anie.200702767. PMID 17763481.
- ^ Константину, Памела Э .; Ван, Тонг; Копач, Йенс; Израиль, Лиза Б .; Чжан, Сяопин; Дин, Баоцюань; Шерман, Уильям Б .; Ван, Син; Чжэн, Цзяньпин; Ша, Руоцзе; Симан, Надриан К. (2006). «Двойная когезия в структурной нанотехнологии ДНК». Органическая и биомолекулярная химия. 4 (18): 3414–9. Дои:10.1039 / b605212f. ЧВК 3491902. PMID 17036134.
- ^ «Архивная копия». Архивировано из оригинал 23 мая 2009 г.. Получено 17 мая 2009.CS1 maint: заархивированная копия как заголовок (связь)
- ^ "Kryptowährungen und Physik - Planetphysics". Архивировано из оригинал 31 марта 2009 г.. Получено 17 мая 2009.
дальнейшее чтение
- Применение новых методов к здоровому питанию, медицинской и сельскохозяйственной биотехнологии. (Июнь 2004 г.) И. К. Баяну, П. Р. Лозано, В. И. Присекару и Х. К. Лин., Q-bio / 0406047.
- Ф. Бессель, Untersuchung des Theils der planetarischen Störungen, Берлин Абхандлунген (1824 г.), статья 14.
- Сэр Лоуренс Брэгг, FRS. Кристаллическое состояние, общий обзор. Лондон: G. Bells and Sons, Ltd., тт. 1 и 2., 1966., 2024 с.
- Кантор, К.Р. и Шиммель, П.Р. Биофизическая химия, части I и II., Сан-Франциско: W.H. Фримен и Ко. 1980. 1800 страниц.
- Воет, Д. и Дж. Дж. Воет. Биохимия, 2-е изд., Нью-Йорк, Торонто, Сингапур: John Wiley & Sons, Inc., 1995, ISBN 0-471-58651-X., 1361 с.
- Уотсон, Г.Н. Трактат по теории функций Бесселя.., (1995) Издательство Кембриджского университета. ISBN 0-521-48391-3.
- Уотсон, Джеймс Д. Молекулярная биология гена. Нью-Йорк и Амстердам: W.A. Benjamin, Inc. 1965., 494 страницы.
- Вентворт, W.E. Физическая химия. Краткий курс., Молден (Массачусетс): Blackwell Science, Inc., 2000.
- Герберт Р. Уилсон, ФРС. Дифракция рентгеновских лучей на белках, нуклеиновых кислотах и вирусах., Лондон: Эдвард Арнольд (Паблишерс) Лтд., 1966.
- Курт Вутрих. ЯМР белков и нуклеиновых кислот., Нью-Йорк, Брисбен, Честер, Торонто, Сингапур: J. Wiley & Sons. 1986г., 292 с.
- Холлин П.Ф., Дэвид Уссери Д. (2004). «База данных CBS Genome Atlas: динамическое хранилище биоинформатических результатов и данных последовательностей ДНК». Биоинформатика. 20 (18): 3682–6. Дои:10.1093 / биоинформатика / bth423. PMID 15256401.
- Чжан СТ, Чжан Р., Оу ХЙ (2003). «База данных кривой Z: графическое представление последовательностей генома». Биоинформатика. 19 (5): 593–599. Дои:10.1093 / биоинформатика / btg041. PMID 12651717.
внешняя ссылка
- ДНК - игра с двойной спиралью С официального сайта Нобелевской премии
- MDDNA: структурная биоинформатика ДНК
- Двойная спираль 1953–2003 гг. Национальный центр биотехнологического образования
- DNAlive: веб-интерфейс для вычисления физических свойств ДНК. Также позволяет связать результаты с UCSC Браузер генома и динамика ДНК.
- Дополнительные сведения о математическом и молекулярном анализе структуры ДНК на основе рентгеновских данных
- Функции Бесселя, соответствующие преобразованиям Фурье атомных или молекулярных спиралей.[мертвая ссылка ]
- обзор принципов STM / AFM / SNOM с обучающими видео
Базы данных для молекулярных моделей и последовательностей ДНК
- дифракция рентгеновских лучей
- Идентификатор NDB: UD0017 База данных
- Рентгеновский Атлас - база данных
- Файлы PDB координат структур нуклеиновых кислот по данным дифракции рентгеновских лучей на кристаллах NA (включая ДНК)
- Структурные факторы загружаемых файлов в формате CIF
- Рассеяние нейтронов
- Источник нейтронов ISIS: Импульсный источник нейтронов ISIS: Всемирный центр науки о нейтронах и мюонах в Харвелле, недалеко от Оксфорда, Великобритания.
- Рентгеновская микроскопия
- Электронная микроскопия
- Базы данных ЯМР
- ЯМР Атлас - база данных
- mmcif загружаемые файлы координат нуклеиновых кислот в растворе из данных 2D-FT ЯМР
- Файлы ограничений ЯМР для НА в формате PDB
- Геномные и структурные базы данных
- База данных генома CBS - содержит примеры перекосов базы.
- База данных геномов по кривой Z - инструмент трехмерной визуализации и анализа геномов.
- Молекулярные модели ДНК и других нуклеиновых кислот: Координатные файлы моделей молекулярной структуры нуклеиновых кислот в форматах PDB и CIF
- Атомно-силовая микроскопия


















