Трансфер-мессенджер РНК - Transfer-messenger RNA
| Трансфер-мессенджер РНК | |
|---|---|
 | |
| Идентификаторы | |
| Символ | тмРНК |
| Рфам | RF00023 |
| Прочие данные | |
| РНК тип | ген |
| PDB структуры | PDBe |
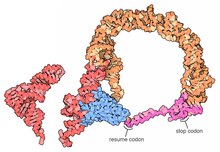
Трансфер-мессенджер РНК (сокращенно тмРНК, также известный как 10Sa РНК и по своему генетическому названию СсрА) является бактериальным РНК молекула с двойным тРНК -как и информационная РНК -подобные свойства. ТмРНК образует рибонуклеопротеин сложный (tmRNP) вместе с Small Protein B (SmpB ), Коэффициент удлинения Tu (EF-Tu ), и рибосомальный белок S1. В транс-трансляция, тмРНК и связанные с ней белки связываются с бактериальными рибосомы которые застопорились посреди биосинтез белка, например при достижении конца информационная РНК который потерял свой стоп-кодон. ТмРНК удивительно универсальна: она перерабатывает застопорившуюся рибосому, добавляет протеолиз -индукция тега к незаконченному полипептид, и способствует деградации аберрантного информационная РНК.[1] У большинства бактерий эти функции выполняются стандартным образом. цельные тмРНК. У других видов бактерий пермутированный ssrA ген производит двухкомпонентная тмРНК в котором две отдельные цепи РНК соединены спариванием оснований.
Открытие и ранняя работа
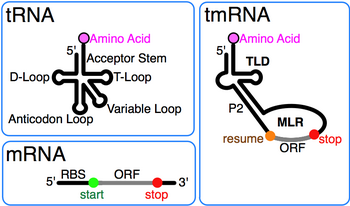
тмРНК сначала была обозначена как 10Sa РНК после смешанной электрофоретической фракции 10S кишечная палочка Далее РНК была разделена на тмРНК и аналогичный размер РНКаза P РНК (10Sb).[2] Наличие псевдоуридин в смешанной 10S РНК указывает на то, что тмРНК имеет модифицированные основания, обнаруженные также в тРНК. Сходство 3'-конца тмРНК с Т-стволовой петлей тРНК был впервые обнаружен при секвенировании ssrA из Микобактерии туберкулеза.[3] Последующее сравнение последовательностей выявило полный тРНК-подобный домен (TLD), образованный 5' и 3' концы тмРНК, включая акцепторный стержень с элементами, подобными элементам в тРНК аланина, которые способствуют ее аминоацилированию посредством аланин-тРНК лигаза.[4] Также были выявлены отличия от тРНК: the антикодон рука отсутствует в тмРНК, а Рука D область представляет собой петлю без пар оснований.
Структура
Вторичная структура стандартных цельных тмРНК

Полный Кишечная палочка тмРНК вторичная структура был разъяснен сравнительный анализ последовательности и структурное зондирование.[5][6] Уотсон-Крик и G-U пар оснований были идентифицированы путем сравнения бактериальных последовательностей тмРНК с использованием автоматизированных вычислительных методов в сочетании с ручным выравнивание процедуры.[7][8] На прилагаемом рисунке показан паттерн спаривания оснований этой прототипной тмРНК, которая организована в 12 филогенетически поддерживаемые спирали (также называемые парами от P1 до P12), некоторые из которых разделены на спиральные сегменты.
Отличительной чертой каждой тмРНК является консервированный тРНК-подобный домен (TLD), состоящий из спиралей 1, 12 и 2a (аналоги акцепторного стержня тРНК, Т-образного стержня и вариабельного стержня, соответственно) и содержащий 5 'монофосфатный и аланилируемый 3' концы CCA. МРНК-подобная область (MLR) в стандартной тмРНК представляет собой большую петлю, содержащую псевдоузлы и кодирующую последовательность (CDS) для метки. пептид, отмечен резюме кодон и стоп-кодон. Кодируемый пептид-метка (ANDENYALAA в Кишечная палочка) варьируется среди бактерий, возможно, в зависимости от набора доступных протеаз и адаптеров.[9]
тмРНК обычно содержат четыре псевдоузлы один (pk1) перед теговым пептидом CDS, а три других псевдоузла (pk2-pk4) ниже CDS. Области псевдоузла, хотя в целом законсервированы, эволюционно пластичны. Например, в (цельных) тмРНК цианобактерии, pk4 заменяется двумя тандемно расположенными меньшими псевдоузлами. Это говорит о том, что сворачивание тмРНК вне TLD может быть важным, однако в области псевдоузла отсутствуют консервативные остатки, а псевдоузлы являются одними из первых структур, которые теряются при ssrA последовательности расходятся в клонах пластид и эндосимбионтов. Спаривание оснований в области трех псевдоузлов Кишечная палочка тмРНК нарушается во время транс-перевод.[7][10]
Двухкомпонентные тмРНК
По кругу ssrA описан в трех основных линиях: i) все альфа-протеобактерии и примитивные митохондрии протистов якобид, ii) две непересекающиеся группы цианобактерии (Gloeobacter и кладу, содержащую Прохлорококк и много Синехококк), и iii) некоторые представители бета-протеобактерий (Cupriavidus и некоторые родоциклы).[11][12] Все они образуют одну и ту же общую форму из двух частей (акцептор и кодирующий элемент), эквивалентную стандартной форме с надрезом после рамки считывания. Ни один не сохраняет более двух псевдоузлы по сравнению с четырьмя (или более) стандартными тмРНК.
Alphaproteobacteria имеют две сигнатурные последовательности: замену типичной последовательности Т-петли TΨCRANY на GGCRGUA и последовательность AACAGAA в большой петле 3´-концевого псевдузла. В митохондриях MLR была потеряна, и произошла заметная повторная перестановка митохондрий. ssrA в результате получается небольшой цельный продукт в Якоба Либера.[13]
В цианобактерии представляют собой наиболее правдоподобный случай эволюции пермутированного гена из стандартного гена из-за значительного сходства последовательностей между двумя типами генов, поскольку они встречаются в разных Синехококк штаммы.
обработка тмРНК
Большинство тмРНК транскрибируются как более крупные предшественники, которые процессируются так же, как тРНК. Расщепление на конце 5´ происходит по рибонуклеаза P.[4] Множественные экзонуклеазы могут участвовать в процессинге 3´ конца тмРНК, хотя РНКаза Т и РНКаза PH наиболее эффективны.[14][15] В зависимости от вида бактерий 3'-CCA кодируется или добавляется тРНК нуклеотидилтрансфераза.
Подобный процессинг на внутренних сайтах пермутированной тмРНК-предшественницы объясняет ее физическое расщепление на две части. Двухкомпонентные тмРНК имеют два дополнительных конца, процессинг которых необходимо учитывать. Для альфа-протеобактерий один 5´ конец является необработанным стартовым сайтом транскрипции.[16] Дальний конец 3´ в некоторых случаях может быть результатом rho-независимого завершения.
Трехмерные конструкции
Структуры с высоким разрешением полных молекул тмРНК в настоящее время недоступны, и их может быть трудно получить из-за внутренней гибкости MLR. В 2007 г. кристаллическая структура Термус термофильный TLD привязан к SmpB белок получали с разрешением 3 Å. Эта структура показывает, что SmpB имитирует стебель D и антикодон канонической тРНК, тогда как спиральный участок 2a тмРНК соответствует вариабельному плечу тРНК.[18]А криоэлектронная микроскопия изучение тмРНК на ранней стадии транс-перевод показывает пространственное соотношение между рибосома и тмРНП (тмРНК, связанная с EF-Tu белок). TLD расположен рядом с центром, связанным с GTPase, в 50S субъединице рибосомы; спираль 5 и псевдоузлы pk2-pk4 образуют дугу вокруг клюва 30S субъединицы рибосомы.[19]
Транс-перевод

Кодирование тмРНК было обнаружено в 1995 году.[20] когда Симпсон с соавторами сверхэкспрессировали мышиный цитокин IL-6 в Кишечная палочка и обнаружил несколько усеченных цитокин -полученный пептиды каждый мечен на карбоксильных концах одним и тем же удлинением из 11 аминокислотных остатков (A) ANDENYALAA. За исключением N-терминала аланин, который происходит от 3'-конца самой тмРНК, эта последовательность тега была прослежена до короткой открытой рамки считывания в Кишечная палочка тмРНК. Признавая, что пептид-метка дает протеолиз, то транс-трансляционная модель действия тмРНК.[21]
Хотя подробности транс-трансляционный механизм изучается, по общему мнению, тмРНК сначала занимает пустой сайт А застопорившейся рибосома. Затем рибосома перемещается с 3 'конца усеченного информационная РНК на кодон возобновления MLR, за которым следует стадия предрасположенности к проскальзыванию, с которой трансляция продолжается нормально до тех пор, пока тмРНК в кадре стоп-кодон встречается. Транс-перевод является важным для некоторых видов бактерий, тогда как другим бактериям требуется тмРНК для выживания в стрессовых условиях роста.[22] В зависимости от организма меточный пептид может распознаваться множеством протеазы или адаптеры протеазы.[9]
Мобильные генетические элементы и ген тмРНК

ssrA является одновременно мишенью для одних мобильных ДНК и пассажирами для других. Было обнаружено, что она прервана тремя типами подвижных элементов. При использовании различных стратегий ни один из них не нарушает функцию генов: группа I интроны удаляются самосращиванием, риккетсиозные палиндромные элементы (RPE) вставьте на безобидные сайты и интегрируйте кодирование геномные острова разделить свою цель ssrA пока восстановлю отколовшуюся часть.[23][24][25][26]
Нехромосомный ssrA был впервые обнаружен при геномном исследовании микобактериофагов (у 10% фагов).[27] Другой мобильные элементы включая плазмиды и геномные острова, несущие ssrA. Один интересный случай Rhodobacter sphaeroides ATCC 17025, чей нативный ген тмРНК нарушен геномным островком; в отличие от всех остальных геномные острова в генах тмРНК (или тРНК) этот остров инактивировал нативный ген-мишень без восстановления, но компенсирует это, неся свой собственный ген тмРНК. Очень необычный родственник ssrA обнаружен в литическом микобактериофаге DS6A, который кодирует немногим больше, чем TLD.
Митохондриальные тмРНК (ssrA ген)
Кодируемая митохондриями структурно восстановленная форма тмРНК (мт-тмРНК) была впервые постулирована для Якобид жгутик Reclinomonas americana.[11] Впоследствии наличие митохондриального гена (ssrA) кодирования тмРНК, а также транскрипции и РНК сайты обработки были подтверждены для всех, кроме одного члена Якобиды.[28][13] Функциональное свидетельство, т. Е. Мт-тмРНК Аминоацилирование с аланин, доступен для Якоба Либера.[13] В последнее время, ssrA был также идентифицирован в митохондриальных геномах оомицеты.[29] Как и в α-Proteobacteria (предки митохондрии ), мт-тмРНК представляют собой двухкомпонентные молекулы РНК с круговой перестановкой, за исключением Якоба Либера где ген вернулся к кодированию конформации цельной тмРНК.[13]
Идентификация ssrA в митохондриальных геномах

Первоначально гены митохондриальной тмРНК считались короткими последовательностями, консервативными среди Якобиды и которые обладают потенциалом складываться в отдельную тРНК-подобную вторичную структуру. При наличии девяти комплектных Якобид мтДНК последовательности,[28] и значительно улучшенный инструмент поиска ковариации (Infernal;[30][31][32]) разработана ковариационная модель на основе Якобид митохондриальные тмРНК, которые идентифицировали митохондриальные ssrA гены также в оомицет. В настоящее время в общей сложности обнаружено 34 мт-тмРНК оомицетов шести родов: Альбуго, Bremia, Фитофтора, Псевдопероноспора, Пифий и Сапролегния. Модель ковариации, построенная с обоими Якобид и оомицет последовательности теперь доступны на Рфам под названием «мт-тмРНК».[29]
Структура мт-тмРНК
Стандартная бактериальная тмРНК состоит из тРНК (Ala) -подобного домена (позволяющего добавлять некодируемый аланин к мРНК, не имеющей стоп-кодирования) и мРНК-подобного домена, кодирующего белковый тег, который предназначен для полипептида. протеолиз. МРНК-подобный домен терялся в мт-тмРНК. Сравнительный анализ последовательностей указывает на особенности, типичные для мт-тмРНК.[29] Наиболее консервативной является первичная последовательность ствола акцептора аминоацила. Эта часть молекулы имеет неизменный остаток A в положении дискриминатора и пару G-U в положении 3 (за исключением Seculamonas ecuadoriensis, имеющий пару G-C); это положение является сайтом узнавания аланил-тРНК-синтазы. P2 представляет собой спираль переменной длины (от 3 до 10 пар оснований) и соответствует стеблю антикодона тРНК, но без петли антикодона (что не требуется для функции тмРНК). P2 стабилизирует тРНК-подобную структуру, но четыре нуклеотида, инвариантные для оомицетов и якобидов, предполагают дополнительную, в настоящее время неидентифицированную функцию. P3 имеет пять пар оснований и соответствует Т-плечу тРНК, но с разными консенсусными нуклеотидами как в парной области, так и в петле. Последовательность Т-петли сохраняется в оомицеты и Якобид, с небольшими отклонениями (например, Сапролегния Ferax). Наконец, вместо тРНК-подобного D-ствола с укороченной трехнуклеотидной D-петлей, характерной для бактериальных тмРНК, митохондриальные аналоги имеют очень вариабельную петлю длиной от 5 до 14 нуклеотидов. Промежуточная последовательность (Int.) Состоящей из двух частей mt-tmRNAs богата A + U и имеет неправильную длину (4-34 нуклеотида). ). Модели вторичной структуры одно- и двухкомпонентных мт-тмРНК см. На рисунке 1.
Обработка и экспрессия мт-тмРНК

Данные РНК-Seq Phytophthora sojae демонстрируют уровень экспрессии, аналогичный уровню экспрессии соседних митохондрий тРНК и четыре основных сайта процессинга подтверждают предсказанные концы зрелой мт-тмРНК.[29] Молекула-предшественник тмРНК, вероятно, процессируется РНКаза P и эндонуклеаза, обрабатывающая 3 ’тРНК (см. фиг. 2); Предполагается, что последнее действие приведет к удалению промежуточной последовательности. После добавления CCA к 3’-дискриминаторному нуклеотиду тмРНК может заряжаться аланил-тРНК синтетазой с аланином.
Смотрите также
Рекомендации
- ^ Кейлер К.С. (2008). «Биология трансляции». Ежегодный обзор микробиологии. 62: 133–51. Дои:10.1146 / annurev.micro.62.081307.162948. PMID 18557701.
- ^ Рэй Б.К., Апирион Д. (июль 1979 г.). «Характеристика 10S РНК: новая стабильная молекула РНК из Escherichia coli». Молекулярная и общая генетика. 174 (1): 25–32. Дои:10.1007 / BF00433301. PMID 384159.
- ^ Тяги Дж. С., Кингер АК (январь 1992 г.). «Идентификация структурного гена РНК 10Sa Mycobacterium tuberculosis». Исследования нуклеиновых кислот. 20 (1): 138. Дои:10.1093 / nar / 20.1.138. ЧВК 310338. PMID 1371186.
- ^ а б Комине Й, Китабатаке М., Йокогава Т., Нисикава К., Инокучи Х. (сентябрь 1994 г.). «Подобная тРНК структура присутствует в 10Sa РНК, небольшой стабильной РНК из Escherichia coli». Труды Национальной академии наук Соединенных Штатов Америки. 91 (20): 9223–7. Дои:10.1073 / пнас.91.20.9223. ЧВК 44784. PMID 7524073.
- ^ Уильямс КП, Бартель Д.П. (декабрь 1996 г.). «Филогенетический анализ вторичной структуры тмРНК». РНК. 2 (12): 1306–10. ЧВК 1369456. PMID 8972778.
- ^ Felden B, Himeno H, Muto A, McCutcheon JP, Atkins JF, Gesteland RF (январь 1997 г.). «Исследование структуры 10Sa РНК (тмРНК) Escherichia coli». РНК. 3 (1): 89–103. ЧВК 1369465. PMID 8990402.
- ^ а б Zwieb C, Wower I, Wower J (май 1999 г.). «Сравнительный анализ последовательности тмРНК». Исследования нуклеиновых кислот. 27 (10): 2063–71. Дои:10.1093 / nar / 27.10.2063. ЧВК 148424. PMID 10219077.
- ^ Андерсен Е.С., Линд-Томсен А., Кнудсен Б., Кристенсен С.Е., Хавгаард Дж. Х., Тораринссон Е., Ларсен Н., Цвиб К., Сестофт П., Кьемс Дж., Городкин Дж. (Ноябрь 2007 г.). «Полуавтоматическое улучшение выравнивания РНК». РНК. 13 (11): 1850–9. Дои:10.1261 / rna.215407. ЧВК 2040093. PMID 17804647.
- ^ а б Гур Э., Зауэр RT (октябрь 2008 г.). «Эволюция тега деградации ssrA в Mycoplasma: переключение специфичности на другую протеазу». Труды Национальной академии наук Соединенных Штатов Америки. 105 (42): 16113–8. Дои:10.1073 / pnas.0808802105. ЧВК 2570983. PMID 18852454.
- ^ Вовер И.К., Цвиб С., Вовер Дж. (Май 2005 г.). «Трансфер-мессенджер РНК разворачивается по мере прохождения рибосомы». РНК. 11 (5): 668–73. Дои:10.1261 / rna.7269305. ЧВК 1370753. PMID 15811920.
- ^ а б Кейлер К.С., Шапиро Л., Уильямс КП (июль 2000 г.). «тмРНК, которые кодируют индуцирующие протеолиз метки, обнаружены во всех известных бактериальных геномах: двухкомпонентная тмРНК функционирует в Caulobacter». Труды Национальной академии наук Соединенных Штатов Америки. 97 (14): 7778–83. Дои:10.1073 / pnas.97.14.7778. ЧВК 16621. PMID 10884408.
- ^ Шаркади С.М., Уильямс КП (2004). «Третья линия с двухкомпонентной тмРНК». Исследования нуклеиновых кислот. 32 (15): 4531–8. Дои:10.1093 / нар / гх795. ЧВК 516066. PMID 15326226.
- ^ а б c d Джейкоб Й., Сейф Э., Паке П.О., Ланг Б.Ф. (апрель 2004 г.). «Потеря мРНК-подобной области в митохондриальных тмРНК якобид». РНК. 10 (4): 605–14. Дои:10.1261 / rna.5227904. ЧВК 1370551. PMID 15037770.
- ^ Шривастава Р.А., Шривастава Н., Апирион Д. (май 1992 г.). «Характеристика фермента процессинга РНК РНКазы III из клеток дикого типа и сверхэкспрессирующих клеток Escherichia coli при переработке природных субстратов РНК». Международный журнал биохимии. 24 (5): 737–49. Дои:10.1016 / 0020-711X (92) 90007-N. PMID 1375563.
- ^ Ли З, Пандит С., Deutscher MP (март 1998 г.). «Экзорибонуклеолитическое обрезание 3 'является общей чертой созревания малых стабильных РНК в Escherichia coli». Труды Национальной академии наук Соединенных Штатов Америки. 95 (6): 2856–61. Дои:10.1073 / пнас.95.6.2856. ЧВК 19659. PMID 9501180.
- ^ Мао С., Бхардвадж К., Шаркади С.М., Фиш Р.И., Дрисколл Т., Вауэр Дж., Цвиб К., Собрал Б.В., Уильямс К.П. (2009). «Вариации гена тмРНК». РНК Биология. 6 (4): 355–61. Дои:10.4161 / rna.6.4.9172. PMID 19617710.
- ^ Someya T, Nameki N, Hosoi H, Suzuki S, Hatanaka H, Fujii M, Terada T, Shirouzu M, Inoue Y, Shibata T., Kuramitsu S, Yokoyama S, Kawai G (январь 2003 г.). «Структура раствора тмРНК-связывающего белка, SmpB, из Thermus thermophilus». Письма FEBS. 535 (1–3): 94–100. Дои:10.1016 / S0014-5793 (02) 03880-2. PMID 12560085.
- ^ а б Бесшо Ю., Сибата Р., Секин С., Мураяма К., Хигасидзима К., Хори-Такемото С., Широузу М., Курамицу С., Йокояма С. (май 2007 г.). «Структурная основа для функциональной мимикрии тРНК с длинным переменным плечом с помощью РНК-мессенджера». Труды Национальной академии наук Соединенных Штатов Америки. 104 (20): 8293–8. Дои:10.1073 / pnas.0700402104. ЧВК 1895943. PMID 17488812.
- ^ Валле М., Жилле Р., Каур С., Хенне А., Рамакришнан В., Франк Дж. (Апрель 2003 г.). «Визуализация входа тмРНК в остановившуюся рибосому». Наука. 300 (5616): 127–30. Дои:10.1126 / science.1081798. PMID 12677067.
- ^ Ту Г.Ф., Рид Г.Е., Чжан Дж. Г., Мориц Р.Л., Симпсон Р.Дж. (апрель 1995 г.). «С-концевое удлинение усеченных рекомбинантных белков в Escherichia coli с декапептидом 10Sa РНК». Журнал биологической химии. 270 (16): 9322–6. Дои:10.1074 / jbc.270.16.9322. PMID 7536743.
- ^ Кейлер К.С., Валлер ПР, Зауэр РТ (февраль 1996 г.). «Роль системы мечения пептидов в деградации белков, синтезированных из поврежденной информационной РНК». Наука. 271 (5251): 990–3. Дои:10.1126 / science.271.5251.990. PMID 8584937.
- ^ Тибонье М, Тиберж Дж. М., Де Реус Х (2008). Ахмед Н (ред.). «Транс-трансляция в Helicobacter pylori: необходимость спасения рибосом и необходимость маркировки белков для устойчивости к стрессу и компетентности». PLOS ONE. 3 (11): e3810. Дои:10.1371 / journal.pone.0003810. ЧВК 2584231. PMID 19043582.
- ^ Кирби Дж. Э., Тремпи Дж. Э., Готтесман С. (апрель 1994). «Иссечение P4-подобного криптического профага приводит к экспрессии протеазы Alp в Escherichia coli». Журнал бактериологии. 176 (7): 2068–81. Дои:10.1128 / jb.176.7.2068-2081.1994. ЧВК 205313. PMID 7511583.
- ^ Уильямс КП (январь 2002 г.). «Веб-сайт тмРНК: вторжение интроном». Исследования нуклеиновых кислот. 30 (1): 179–82. Дои:10.1093 / nar / 30.1.179. ЧВК 99078. PMID 11752287.
- ^ Двайер Д.С. (январь 2001 г.). «Эгоистичная ДНК и происхождение генов». Наука. 291 (5502): 252–3. Дои:10.1126 / science.291.5502.252. PMID 11253208.
- ^ Уильямс КП (февраль 2003 г.). «Трафик в гене тмРНК». Журнал бактериологии. 185 (3): 1059–70. Дои:10.1128 / JB.185.3.1059-1070.2003. ЧВК 142792. PMID 12533482.
- ^ Hatfull GF, Pedulla ML, Jacobs-Sera D, Cichon PM, Foley A, Ford ME, Gonda RM, Houtz JM, Hryckowian AJ, Kelchner VA, Namburi S, Pajcini KV, Popovich MG, Schleicher DT, Simanek BZ, Smith AL, Зданович Г.М., Кумар В., Пиблз К.Л., Джейкобс В.Р., Лоуренс Дж. Г., Хендрикс Р. В. (июнь 2006 г.). «Изучение метапротеома микобактериофага: геномика фага как образовательная платформа». PLoS Genetics. 2 (6): e92. Дои:10.1371 / journal.pgen.0020092. ЧВК 1475703. PMID 16789831.
- ^ а б Burger G, Gray MW, Забудьте L, Lang BF (2013). «Поразительно бактериоподобные и богатые генами митохондриальные геномы у протистов якобидов». Геномная биология и эволюция. 5 (2): 418–38. Дои:10.1093 / gbe / evt008. ЧВК 3590771. PMID 23335123.
- ^ а б c d Хафез М., Бургер Г., Стейнберг С.В., Ланг Б.Ф. (июль 2013 г.). «Вторая эукариотическая группа с тмРНК, кодируемой митохондриями: идентификация in silico и экспериментальное подтверждение». РНК Биология. 10 (7): 1117–24. Дои:10.4161 / rna.25376. ЧВК 3849159. PMID 23823571. Архивировано из оригинал на 2014-02-21. Получено 2014-02-13.
- ^ Эдди, С. «Адский сайт». Получено 14 августа 2016.
- ^ Эдди С.Р., Дурбин Р. (июнь 1994 г.). «Анализ последовательности РНК с использованием ковариационных моделей». Исследования нуклеиновых кислот. 22 (11): 2079–88. Дои:10.1093 / nar / 22.11.2079. ЧВК 308124. PMID 8029015.
- ^ Nawrocki EP, Kolbe DL, Eddy SR (май 2009 г.). «Infernal 1.0: вывод выравнивания РНК». Биоинформатика. 25 (10): 1335–7. Дои:10.1093 / биоинформатика / btp157. ЧВК 2732312. PMID 19307242.
дальнейшее чтение
- Хонг С.Дж., Тран QA, Кейлер К.С. (июль 2005 г.). «Регулируемая клеточным циклом деградация тмРНК контролируется РНКазой R и SmpB». Молекулярная микробиология. 57 (2): 565–75. Дои:10.1111 / j.1365-2958.2005.04709.x. ЧВК 3776457. PMID 15978085.


