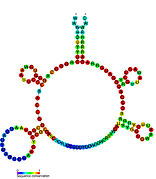
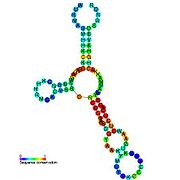
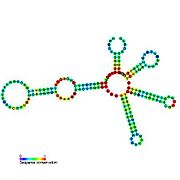
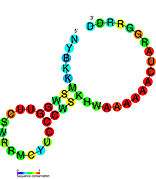
Riboswitch - Riboswitch

В молекулярная биология, а рибопереключатель является регуляторным сегментом информационная РНК молекула, которая связывает малая молекула, что приводит к изменению производство из белки кодируется мРНК.[1][2][3][4] Таким образом, мРНК, содержащая рибопереключатель, непосредственно участвует в регуляции своей собственной активности в ответ на концентрации ее эффектор молекула. Открытие того, что современные организмы используют РНК для связывания малых молекул и дискриминации близкородственных аналогов, расширило известные природные возможности РНК за пределы ее способности кодировать белки, катализировать реакции, или связать другую РНК или белок макромолекулы.
Первоначальное определение термина «рибопереключатель» указывало, что они непосредственно воспринимают малую молекулу. метаболит концентрации.[5] Хотя это определение остается широко используемым, некоторые биологи использовали более широкое определение, которое включает другие цис-регуляторные РНК. Однако в этой статье будут обсуждаться только рибопереключатели, связывающие метаболиты.
Наиболее известные рибопереключатели встречаются в бактерии, но функциональные рибопереключатели одного типа ( Рибровыключатель TPP ) были обнаружены в архее, растения и некоторые грибы. Рибопереключатели TPP также были предсказаны в археи,[6] но экспериментально не проверено.
История и открытия
До открытия рибопереключателей механизм, с помощью которого регулируются некоторые гены, участвующие во множестве метаболических путей, оставался загадкой. Накапливающиеся данные все чаще подтверждают беспрецедентную для того времени идею о том, что задействованные мРНК могут напрямую связывать метаболиты, влияя на их собственное регулирование. Эти данные включали консервативную РНК. второстепенные конструкции часто встречается в непереведенных регионах (UTR ) соответствующих генов и успех процедур по созданию искусственных малых молекул-связывающих РНК, называемых аптамеры.[7][8][9][10][11] В 2002 году были опубликованы первые исчерпывающие доказательства существования нескольких классов рибопереключателей, в том числе анализы связывания без белков, а рибопереключатели, связывающие метаболиты, были созданы как новый механизм регуляции генов.[5][12][13][14]
Многие из самых ранних обнаруженных рибопереключателей соответствовали «мотивам» (паттернам) консервативных последовательностей в 5 'UTR что, по-видимому, соответствует структурированной РНК. Например, сравнительный анализ вышележащих областей нескольких генов, которые, как ожидается, будут совместно регулироваться, привел к описанию S-бокса.[15] (теперь рибопереключатель SAM-I), THI-box[9] (область внутри рибопереключателя TPP), элемент RFN[8] (теперь рибопереключатель FMN) и B12-коробка[16] (часть рибопереключателя кобаламина), а в некоторых случаях экспериментальные демонстрации того, что они участвовали в регуляции генов посредством неизвестного механизма. Биоинформатика сыграл важную роль в более поздних открытиях, с увеличением автоматизации базовой стратегии сравнительной геномики. Баррик и другие. (2004)[17] используемый ВЗРЫВ найти UTR гомологичный ко всем UTR в Bacillus subtilis. Некоторые из этих гомологичных наборов были проверены на консервативную структуру, что дало 10 РНК-подобных мотивов. Три из них были позже экспериментально подтверждены как рибопереключатели glmS, глицина и PreQ1-I (см. Ниже). Последующие попытки сравнительной геномики с использованием дополнительных таксонов бактерий и улучшенных компьютерных алгоритмов определили дополнительные рибопереключатели, которые экспериментально подтверждены, а также консервативные структуры РНК, которые, как предполагается, функционируют как рибопереключатели.[18][19][20]
Механизмы
Рибопереключатели часто концептуально делятся на две части: аптамер и платформу самовыражения. Аптамер напрямую связывает небольшую молекулу, и платформа экспрессии претерпевает структурные изменения в ответ на изменения в аптамере. Платформа экспрессии - это то, что регулирует экспрессию генов.
Платформы экспрессии обычно отключают экспрессию генов в ответ на небольшую молекулу, но некоторые включают ее. Экспериментально продемонстрированы следующие механизмы рибопереключения.
- Рибервыключатель формирует rho-независимая терминация транскрипции шпильки приводят к преждевременному прекращению транскрипции.
- Складывание, опосредованное рибозвитком, секвестрирует сайт связывания рибосомы, тем самым подавляя перевод.
- Рибопереключатель - это рибозим который расщепляется в присутствии достаточных концентраций его метаболита.
- Альтернативные структуры рибопереключателя влияют на сращивание пре-мРНК.
- Рибопереключатель TPP в Neurospora crassa (грибок) контролирует альтернативное сращивание, чтобы условно произвести Открытая рамка чтения восходящего потока (uORF), тем самым влияя на экспрессию нижестоящих генов[21]
- Рибопереключатель TPP в установках модифицирует сращивание и альтернативную обработку 3'-концов[22][23]
- Рибопереключатель в Clostridium acetobutylicum регулирует соседний ген, который не является частью одного и того же транскрипта мРНК. В этой регуляции рибопереключатель вмешивается в транскрипцию гена. Механизм неясен, но может быть вызван конфликтами между двумя звеньями РНК-полимеразы, поскольку они одновременно транскрибируют одну и ту же ДНК.[24]
- Рибопереключатель в Listeria monocytogenes регулирует экспрессию своего нижнего гена. Однако транскрипты рибопереключателя впоследствии модулируют экспрессию гена, расположенного в другом месте генома.[25] Эта транс регуляция происходит посредством спаривания оснований с мРНК дистального гена. При повышении температуры бактерии рибопереключатель плавится, обеспечивая транскрипцию. Неопубликованные исследования бакалавров создали подобный рибопереключатель или «термодатчик» посредством случайного мутагенеза последовательности Listeria monocytogenes.[26]
Типы
Ниже приводится список экспериментально подтвержденных рибопереключателей, упорядоченных по лигандам.
- Кобаламин рибопереключатель (также B12-элемент), который связывает либо аденозилкобаламин (коферментная форма витамин B12 ) или аквокобаламин регулировать кобаламин биосинтез и транспорт кобаламина и подобных метаболитов, а также других генов.
- циклические рибопереключатели AMP-GMP связать сигнальную молекулу циклический AMP-GMP. Эти рибопереключатели структурно относятся к циклические рибопереключатели di-GMP-I (см. также «циклический ди-GMP» ниже).
- циклические ди-AMP рибопереключатели (также называется ydaO / yuaA) связывают сигнальную молекулу циклический ди-АМФ.
- циклические рибопереключатели di-GMP связать сигнальную молекулу циклический ди-GMP для того, чтобы регулировать множество генов, контролируемых этим вторым мессенджером. Известны два класса циклических рибопереключателей di-GMP: циклические рибопереключатели di-GMP-I и циклические рибопереключатели di-GMP-II. Эти классы не кажутся структурно связанными.
- фторидные рибопереключатели чувствуют ионы фтора и функционируют в выживании при высоких уровнях фторид.
- Рибопереключатель FMN (также RFN-элемент) связывает флавинмононуклеотид (FMN) для регулирования рибофлавин биосинтез и транспорт.
- Рибопереключатель glmS, который представляет собой рибозим, который расщепляется при достаточной концентрации глюкозамин-6-фосфат.
- Рибопереключатели глутамина связывать глутамин для регулирования генов, участвующих в глютамине и азот метаболизм, а также короткие пептиды неизвестной функции. Известны два класса глутаминовых рибопереключателей: glnA РНК-мотив и Нижестоящий пептидный мотив. Считается, что эти классы структурно связаны (см. Обсуждения в этих статьях).
- Глицин рибопереключатель связывает глицин, чтобы регулировать гены метаболизма глицина, включая использование глицина в качестве источника энергии. До 2012 года этот рибопереключатель считался единственным, что совместная привязка, так как он содержит смежные дуальные аптамеры. Хотя совместная работа более не доказана, причина двойных аптамеров все еще остается неоднозначной.[27]
- Рибопереключатель лизина (также L-коробка) связывает лизин регулировать биосинтез лизина, катаболизм и транспорт.
- марганцевые рибопереключатели связывать марганец ионы.
- Рибопереключатели NiCo связывают ионы металлов никель и кобальт.
- Рибопереключатели PreQ1 связывать пре-кевозин1, чтобы регулировать гены, участвующие в синтезе или транспорте этого предшественника в Queuosine. Известны три совершенно разных класса рибопереключателей PreQ1: Рибопереключатели PreQ1-I, Рибопереключатели PreQ1-II и Рибопереключатели PreQ1-III. Связывающий домен рибопереключателей PreQ1-I необычно мал среди встречающихся в природе рибопереключателей. Рибопереключатели PreQ1-II, которые встречаются только у определенных видов в родах Стрептококк и Лактококк, имеют совершенно другую структуру и больше по размеру, как и рибопереключатели PreQ1-III.
- Пуриновые рибопереключатели связывает пурины для регулирования метаболизма и транспорта пуринов. Различные формы связывания пуринового рибопереключателя гуанин (форма, первоначально известная как G-бокс) или аденин. Специфичность гуанина или аденина полностью зависит от взаимодействий Уотсона-Крика с одним пиримидин в рибопереключатель в положении Y74. В рибопереключателе гуанина этот остаток всегда цитозин (т.е. C74), в остатке аденина всегда присутствует урацил (например, U74). Гомологичные типы пуриновых рибопереключателей связываются дезоксигуанозин, но имеют более существенные различия, чем одиночная нуклеотидная мутация.
- Рибопереключатели SAH связывать S-аденозилгомоцистеин для регулирования генов, участвующих в переработке этого метаболита, который образуется при S-аденозилметионин используется в реакциях метилирования.
- Рибопереключатели SAM связывать S-аденозил метионин (SAM) для регулирования метионин биосинтез и транспорт SAM. Известны три различных рибопереключателя SAM: SAM-I (первоначально назывался S-коробка), ЗРК-II и SМК коробка рибопереключатель. SAM-I широко распространен у бактерий, но SAM-II обнаружен только в альфа-, бета- и некоторых гамма-протеобактерии. SМК коробочный рибопереключатель находится только в заказе Лактобациллы. Эти три разновидности рибопереключателя не имеют очевидного сходства с точки зрения последовательности или структуры. Четвертый сорт, Рибопереключатели SAM-IV, по-видимому, имеет ядро, сходное с лиганд-связывающим ядром, что и у рибосвитчей SAM-I, но в контексте отдельного каркаса.
- Рибопереключатели SAM-SAH связывают как SAM, так и SAH с аналогичным сродством. Поскольку они всегда могут регулировать гены, кодирующие метионин аденозилтрансфераза было высказано предположение, что физиологически значимым является только их связывание с SAM.
- Рибопереключатели с тетрагидрофолатом связывать тетрагидрофолат регулировать синтез и транспортные гены.
- Рибопереключатели ТПП (также THI-box) связывает тиаминпирофосфат (TPP) для регулирования тиамин биосинтез и транспорт, а также транспорт подобных метаболитов. Это единственный рибопереключатель, обнаруженный до сих пор у эукариот.[28]
- Рибопереключатели ZMP / ZTP смысл ZMP и ZTP, которые являются побочными продуктами метаболизма пуринов de novo, когда уровни 10-формилтетрагидрофолат низкие.
Предполагаемые рибопереключатели:
- Моко РНК-мотив предполагается связать кофактор молибдена, чтобы регулировать гены, участвующие в биосинтезе и транспорте этого кофермента, а также ферменты, которые используют его или его производные в качестве кофактора.
Рибопереключатели-кандидаты, связывающиеся с метаболитами, были идентифицированы с помощью биоинформатики и имеют умеренно сложный характер. второстепенные конструкции и несколько очень консервативных нуклеотид позиции, поскольку эти особенности типичны для рибопереключателей, которые должны специфически связывать небольшую молекулу. Кандидаты в рибопереключатели также постоянно расположены в 5'-UTR генов, кодирующих белок, и эти гены указывают на связывание метаболитов, поскольку они также являются особенностями большинства известных рибопереключателей. Предполагаемые кандидаты на рибопереключатель, хорошо согласующиеся с предыдущими критериями, следующие: crcB РНК-мотив, Мотив РНК manA, pfl РНК-мотив, ydaO / yuaA лидер, yjdF РНК-мотив, ykkC-yxkD лидер (и родственный мотив РНК ykkC-III) и yybP-ykoY лидер. Функции этих гипотетических рибопереключателей остаются неизвестными.
Вычислительные модели
Рибопереключатели также были исследованы с использованием подходов in-silico.[29][30][31] В частности, решения для предсказания рибопереключателей можно разделить на две большие категории:
- генные искатели рибопереключателей, то есть системы, направленные на обнаружение рибопереключателей с помощью геномных исследований, в основном основанные на механизмах поиска мотивов. Эта группа содержит Infernal, основополагающий компонент Рфам база данных,[32] и более специфические инструменты, такие как RibEx[33] или RiboSW.[34]
- предикторы конформационного переключения, т.е. методы, основанные на структурной классификации альтернативных структур, таких как paRNAss,[35] РНК-формы[36] и РНКбор.[37] Кроме того, были также предложены семейные подходы для предсказания структуры включения / выключения.[38]
Инструмент SwiSpot[39] каким-то образом охватывает обе группы, поскольку использует конформационные предсказания для оценки наличия рибопереключателей.
Гипотеза мира РНК
Рибопереключатели демонстрируют, что встречающиеся в природе РНК может специфически связывать небольшие молекулы, и многие ранее считали, что это свойство белки или искусственно сконструированные РНК, называемые аптамеры. Таким образом, существование рибопереключателей во всех сферах жизни добавляет некоторую поддержку Гипотеза мира РНК, который утверждает, что жизнь изначально существовала с использованием только РНК, а белки появились позже; эта гипотеза требует, чтобы все критические функции, выполняемые белками (включая связывание малых молекул), могли выполняться РНК. Было высказано предположение, что некоторые рибопереключатели могут представлять древние регуляторные системы или даже остатки РНК-мира. рибозимы чьи связывающие домены сохранены.[13][18][40]
В качестве мишеней антибиотиков
Рибопереключатели могут стать мишенью для романа антибиотики. Действительно, было показано, что некоторые антибиотики, механизм действия которых был неизвестен в течение десятилетий, воздействуют на рибопереключатели.[41] Например, когда антибиотик пиритиамин попадает в клетку, метаболизируется в пирофосфат пиритиамина. Было показано, что пирофосфат пиритиамина связывает и активирует рибопереключатель TPP, заставляя клетку прекращать синтез и импорт TPP. Поскольку пирофосфат пиритиамина не заменяет TPP в качестве кофермента, клетка умирает.
Инженерные рибопереключатели
Поскольку рибопереключатели являются эффективным методом контроля экспрессии генов в естественных организмах, возник интерес к разработке искусственных рибопереключателей.[42][43][44]для промышленного и медицинского применения, например, генная терапия.[45][46]
использованная литература
- ^ Нудлер Э., Миронов А.С. (январь 2004 г.). «Рибопереключатель контроля метаболизма бактерий». Тенденции в биохимических науках. 29 (1): 11–17. Дои:10.1016 / j.tibs.2003.11.004. PMID 14729327.
- ^ Такер Б.Дж., Брейкер Р.Р. (июнь 2005 г.). «Рибопереключатели как универсальные элементы контроля генов». Текущее мнение в структурной биологии. 15 (3): 342–348. Дои:10.1016 / j.sbi.2005.05.003. PMID 15919195.
- ^ Витрещак А.Г., Родионов Д.А., Миронов А.А., Гельфанд М.С. (январь 2004 г.). «Рибопереключатели: древнейший механизм регуляции экспрессии генов?». Тенденции в генетике. 20 (1): 44–50. CiteSeerX 10.1.1.312.9100. Дои:10.1016 / j.tig.2003.11.008. PMID 14698618.
- ^ Батей RT (июнь 2006 г.). «Структуры регуляторных элементов в мРНК». Текущее мнение в структурной биологии. 16 (3): 299–306. Дои:10.1016 / j.sbi.2006.05.001. PMID 16707260.
- ^ а б Нахви А., Сударсан Н., Эберт М.С., Зоу Х, Браун К.Л., Брейкер Р.Р. (сентябрь 2002 г.). «Генетический контроль с помощью мРНК, связывающей метаболит». Химия и биология. 9 (9): 1043–1049. Дои:10.1016 / S1074-5521 (02) 00224-7. PMID 12323379.
- ^ Сударсан Н., Баррик Дж. Э., Брейкер Р. Р. (июнь 2003 г.). «Метаболит-связывающие домены РНК присутствуют в генах эукариот». РНК. 9 (6): 644–647. Дои:10.1261 / rna.5090103. ЧВК 1370431. PMID 12756322.
- ^ Ноу X, Каднер Р.Дж. (июнь 2000 г.). «Аденозилкобаламин ингибирует связывание рибосом с btuB РНК». Труды Национальной академии наук Соединенных Штатов Америки. 97 (13): 7190–7195. Bibcode:2000PNAS ... 97.7190N. Дои:10.1073 / pnas.130013897. ЧВК 16521. PMID 10852957.
- ^ а б Гельфанд М.С., Миронов А.А., Жомантас Ю., Козлов Ю.И., Перумов Д.А. (ноябрь 1999 г.). «Консервативный элемент структуры РНК, участвующий в регуляции генов синтеза бактериального рибофлавина». Тенденции в генетике. 15 (11): 439–442. Дои:10.1016 / S0168-9525 (99) 01856-9. PMID 10529804.
- ^ а б Миранда-Риос Дж., Наварро М., Соберон М. (август 2001 г.). «Консервативная структура РНК (ти-бокс) участвует в регуляции экспрессии гена биосинтеза тиамина в бактериях». Труды Национальной академии наук Соединенных Штатов Америки. 98 (17): 9736–9741. Дои:10.1073 / pnas.161168098. ЧВК 55522. PMID 11470904.
- ^ Стормо Г.Д., Джи И (август 2001 г.). «Действуют ли мРНК как прямые сенсоры малых молекул для контроля их экспрессии?». Труды Национальной академии наук Соединенных Штатов Америки. 98 (17): 9465–9467. Bibcode:2001PNAS ... 98.9465S. Дои:10.1073 / pnas.181334498. ЧВК 55472. PMID 11504932.
- ^ Gold L, Brown D, He Y, Shtatland T, Singer BS, Wu Y (январь 1997 г.). «От форм олигонуклеотидов к геномному SELEX: новые биологические регуляторные петли». Труды Национальной академии наук Соединенных Штатов Америки. 94 (1): 59–64. Bibcode:1997PNAS ... 94 ... 59G. Дои:10.1073 / пнас.94.1.59. ЧВК 19236. PMID 8990161.
- ^ Миронов А.С., Гусаров И., Рафиков Р., Лопес Л.Е., Шаталин К., Кренева Р.А., Перумов Д.А., Нудлер Э. (ноябрь 2002 г.). «Чувство малых молекул с помощью возникающей РНК: механизм контроля транскрипции в бактериях». Ячейка. 111 (5): 747–756. Дои:10.1016 / S0092-8674 (02) 01134-0. PMID 12464185.
- ^ а б Винклер В., Нахви А., Брейкер Р. Р. (октябрь 2002 г.). «Производные тиамина напрямую связывают информационные РНК, регулируя экспрессию бактериальных генов». Природа. 419 (6910): 952–956. Bibcode:2002 Натур 419..952 Вт. Дои:10.1038 / природа01145. PMID 12410317.
- ^ Винклер В. К., Коэн-Чаламиш С., Брейкер Р. Р. (декабрь 2002 г.). «Структура мРНК, которая контролирует экспрессию гена путем связывания FMN». Труды Национальной академии наук Соединенных Штатов Америки. 99 (25): 15908–15913. Bibcode:2002PNAS ... 9915908W. Дои:10.1073 / pnas.212628899. ЧВК 138538. PMID 12456892.
- ^ Гранди Ф.Дж., Хенкин TM (ноябрь 1998 г.). «S-бокс регулон: новая глобальная система контроля терминации транскрипции для генов биосинтеза метионина и цистеина у грамположительных бактерий». Молекулярная микробиология. 30 (4): 737–749. Дои:10.1046 / j.1365-2958.1998.01105.x. PMID 10094622.
- ^ Франклунд CV, Каднер Р.Дж. (июнь 1997 г.). «Множественные транскрибируемые элементы контролируют экспрессию гена btuB Escherichia coli». Журнал бактериологии. 179 (12): 4039–4042. Дои:10.1128 / jb.179.12.4039-4042.1997. ЧВК 179215. PMID 9190822.
- ^ Баррик Дж. Э., Корбино К. А., Винклер В. К., Нави А., Мандал М., Коллинз Дж., Ли М., Рот А., Сударсан Н., Йона И., Викизер Дж. К., Брейкер Р. Р. (апрель 2004 г.). «Новые мотивы РНК предполагают расширение возможностей рибопереключателей в генетическом контроле бактерий». Труды Национальной академии наук Соединенных Штатов Америки. 101 (17): 6421–6426. Bibcode:2004ПНАС..101.6421Б. Дои:10.1073 / pnas.0308014101. ЧВК 404060. PMID 15096624.
- ^ а б Корбино К.А., Баррик Дж. Э., Лим Дж., Велц Р., Такер Б. Дж., Пускарц И., Мандал М., Рудник Н. Д., Брейкер Р. Р. (2005). «Доказательства второго класса рибопереключателей S-аденозилметионина и других регуляторных мотивов РНК у альфа-протеобактерий». Геномная биология. 6 (8): R70. Дои:10.1186 / gb-2005-6-8-r70. ЧВК 1273637. PMID 16086852.
- ^ Вайнберг З., Баррик Дж. Э., Яо З., Рот А., Ким Дж. Н., Гор Дж, Ван Дж. X, Ли Э. Р., Блок К. Ф., Сударсан Н., Неф С., Томпа М., Руццо В. Л., Брейкер Р. Р. (2007). «Идентификация 22 кандидатных структурированных РНК в бактериях с использованием системы сравнительной геномики CMfinder». Исследования нуклеиновых кислот. 35 (14): 4809–4819. Дои:10.1093 / нар / гкм487. ЧВК 1950547. PMID 17621584.
- ^ Weinberg Z, Wang JX, Bogue J, Yang J, Corbino K, Moy RH, Breaker RR (март 2010 г.). «Сравнительная геномика обнаруживает 104 кандидата структурированных РНК из бактерий, архей и их метагеномов». Геномная биология. 11 (3): R31. Дои:10.1186 / gb-2010-11-3-r31. ЧВК 2864571. PMID 20230605.
- ^ Cheah MT, Wachter A, Sudarsan N, Breaker RR (май 2007 г.). «Контроль альтернативного сплайсинга РНК и экспрессии генов эукариотическими рибопереключателями». Природа. 447 (7143): 497–500. Bibcode:2007Натура.447..497C. Дои:10.1038 / природа05769. PMID 17468745.
- ^ Wachter A, Tunc-Ozdemir M, Grove BC, Green PJ, Shintani DK, Breaker RR (ноябрь 2007 г.). «Рибопереключатель контроля экспрессии генов в растениях путем сплайсинга и альтернативного 3'-концевого процессинга мРНК». Растительная клетка. 19 (11): 3437–3450. Дои:10.1105 / tpc.107.053645. ЧВК 2174889. PMID 17993623.
- ^ Бокобза С., Адато А., Мандель Т., Шапира М., Нудлер Е., Ахарони А. (ноябрь 2007 г.). «Рибосвитч-зависимая регуляция генов и ее эволюция в царстве растений». Гены и развитие. 21 (22): 2874–2879. Дои:10.1101 / gad.443907. ЧВК 2049190. PMID 18006684.
- ^ Андре Дж., Эвен С., Путцер Х, Бургьер П., Кру С., Данчен А., Мартин-Верстрете I, Сутурина О. (октябрь 2008 г.). «Рибопереключатели S-box и T-box и антисмысловая РНК контролируют серный метаболический оперон Clostridium acetobutylicum». Исследования нуклеиновых кислот. 36 (18): 5955–5969. Дои:10.1093 / nar / gkn601. ЧВК 2566862. PMID 18812398.
- ^ Ло Э, Дюссургет О, Грипенланд Дж, Вайткявичюс К., Тиенсуу Т., Мандин П., Репоила Ф, Бухризер С., Коссарт П., Йоханссон Дж. (Ноябрь 2009 г.). «Транс-действующий рибопереключатель контролирует экспрессию регулятора вирулентности PrfA в Listeria monocytogenes». Ячейка. 139 (4): 770–779. Дои:10.1016 / j.cell.2009.08.046. PMID 19914169.
- ^ http://2011.igem.org/Team:BYU_Provo/Results
- ^ Шерман Э.М., Эскиаки Дж., Эльсайед Дж., Й. Д. (март 2012 г.). «Энергетически выгодное взаимодействие лидер-линкер устраняет кооперативность связывания лиганда в глициновых рибопереключателях». РНК. 18 (3): 496–507. Дои:10.1261 / rna.031286.111. ЧВК 3285937. PMID 22279151.
- ^ Bocobza SE, Aharoni A (октябрь 2008 г.). «Включение света на заводских рибовключателях». Тенденции Plant Sci. 13: 526–33. Дои:10.1016 / j.tplants.2008.07.004. PMID 18778966.
- ^ Баррик Дж. Э. (2009). «Прогнозирование регуляции рибопереключателя в геномном масштабе». Методы молекулярной биологии. 540: 1–13. Дои:10.1007/978-1-59745-558-9_1. ISBN 978-1-934115-88-6. PMID 19381548.
- ^ Бараш Д., Габданк I (январь 2010 г.). «Минимизация энергии применительно к рибопереключателям: перспективы и проблемы». РНК Биология. 7 (1): 90–97. Дои:10.4161 / rna.7.1.10657. PMID 20061789.
- ^ Чен, Ши-Цзе; Берк, Дональд Х; Адамиак, Р. В. (2015). Вычислительные методы для понимания рибопереключателей / Методы в энзимологии, том 553. Академическая пресса. ISBN 978-0-12-801618-3.
- ^ Nawrocki EP, Kolbe DL, Eddy SR (май 2009 г.). «Infernal 1.0: вывод выравнивания РНК». Биоинформатика. 25 (10): 1335–1337. Дои:10.1093 / биоинформатика / btp157. ЧВК 2732312. PMID 19307242.
- ^ Абреу-Гуджер С., Мерино Е. (июль 2005 г.). «RibEx: веб-сервер для поиска рибопереключателей и других консервативных бактериальных регуляторных элементов». Исследования нуклеиновых кислот. 33 (Проблема с веб-сервером): W690-2. Дои:10.1093 / нар / gki445. ЧВК 1160206. PMID 15980564.
- ^ Чанг TH, Хуанг HD, Wu LC, Yeh CT, Liu BJ, Horng JT (июль 2009 г.). «Вычислительная идентификация рибопереключателей на основе консервативных функциональных последовательностей и конформаций РНК». РНК. 15 (7): 1426–1430. Дои:10.1261 / rna.1623809. ЧВК 2704089. PMID 19460868.
- ^ Восс Б., Мейер С., Гигерих Р. (июль 2004 г.). «Оценка предсказуемости конформационного переключения в РНК». Биоинформатика. 20 (10): 1573–1582. Дои:10.1093 / биоинформатика / bth129. PMID 14962925.
- ^ Янссен С., Гигерих Р. (февраль 2015 г.). "Студия форм РНК". Биоинформатика. 31 (3): 423–425. Дои:10.1093 / биоинформатика / btu649. ЧВК 4308662. PMID 25273103.
- ^ Фрейхульт Э., Моултон В., Клот П. (август 2007 г.). «Больцмановская вероятность структурных соседей РНК и обнаружение рибосвитча». Биоинформатика. 23 (16): 2054–2062. Дои:10.1093 / биоинформатика / btm314. PMID 17573364.
- ^ Clote P, Lou F, Lorenz WA (апрель 2012 г.). «Максимально ожидаемая точность структурных соседей вторичной структуры РНК». BMC Bioinformatics. 13 Дополнение 5 (Дополнение 5): S6. Дои:10.1186 / 1471-2105-13-S5-S6. ЧВК 3358666. PMID 22537010.
- ^ Barsacchi M, Novoa EM, Kellis M, Bechini A (ноябрь 2016 г.). «SwiSpot: моделирование рибопереключателей путем определения последовательностей переключения». Биоинформатика. 32 (21): 3252–3259. Дои:10.1093 / биоинформатика / btw401. PMID 27378291.
- ^ Cochrane JC, Strobel SA (июнь 2008 г.). «Эффекторы рибосвитча как кофакторы белковых ферментов». РНК. 14 (6): 993–1002. Дои:10.1261 / rna.908408. ЧВК 2390802. PMID 18430893.
- ^ Блаунт К.Ф., Брейкер Р.Р. (декабрь 2006 г.). «Рибопереключатели как мишени для антибактериальных препаратов». Природа Биотехнологии. 24 (12): 1558–1564. Дои:10.1038 / nbt1268. PMID 17160062.
- ^ Бауэр Дж., Зюсс Б. (июнь 2006 г.). «Разработанные рибопереключатели как новые инструменты в молекулярной биологии». Журнал биотехнологии. 124 (1): 4–11. Дои:10.1016 / j.jbiotec.2005.12.006. PMID 16442180.
- ^ Диксон Н., Дункан Дж., Герлингс Т., Данстан М.С., Маккарти Дж. Э., Лейс Д., Миклфилд Дж. (Февраль 2010 г.). «Реинжиниринг ортогонально селективных рибопереключателей». Труды Национальной академии наук Соединенных Штатов Америки. 107 (7): 2830–2835. Bibcode:2010PNAS..107.2830D. Дои:10.1073 / pnas.0911209107. ЧВК 2840279. PMID 20133756.
- ^ Верхуниг А., Керхер Д., Бок Р. (апрель 2010 г.). «Индуцируемая экспрессия гена из пластидного генома с помощью синтетического рибопереключателя». Труды Национальной академии наук Соединенных Штатов Америки. 107 (14): 6204–6209. Bibcode:2010ПНАС..107.6204В. Дои:10.1073 / pnas.0914423107. ЧВК 2852001. PMID 20308585.
- ^ Кетцер П., Кауфманн Дж. К., Энгельхардт С., Боссов С., фон Калле С., Хартиг Дж. С., Унгерехтс Г., Неттельбек Д. М. (февраль 2014 г.). «Искусственные рибопереключатели для контроля экспрессии генов и репликации ДНК и РНК вирусов». Труды Национальной академии наук Соединенных Штатов Америки. 111 (5): E554–562. Bibcode:2014PNAS..111E.554K. Дои:10.1073 / pnas.1318563111. ЧВК 3918795. PMID 24449891.
- ^ Штробель Б., Клаузер Б., Хартиг Дж. С., Ламла Т., Гантнер Ф., Кройц С. (октябрь 2015 г.). «Опосредованное рибосвитчем ослабление цитотоксичности трансгена увеличивает выход аденоассоциированных вирусных векторов в клетках HEK-293». Молекулярная терапия. 23 (10): 1582–1591. Дои:10.1038 / мт.2015.123. ЧВК 4817922. PMID 26137851.
дальнейшее чтение
- Ферре-Д'Амаре, Адриан Р .; Винклер, Уэйд С. (2011). «Глава 5. Роль ионов металлов в регуляции с помощью рибопереключателей». В Астрид Сигель, Гельмут Сигель и Роланд К. О. Сигель (ред.). Структурная и каталитическая роль ионов металлов в РНК. Ионы металлов в науках о жизни. 9. Кембридж, Великобритания: RSC Publishing. С. 141–173. Дои:10.1039/9781849732512-00141. ISBN 978-1-84973-094-5. ЧВК 3454353. PMID 22010271.